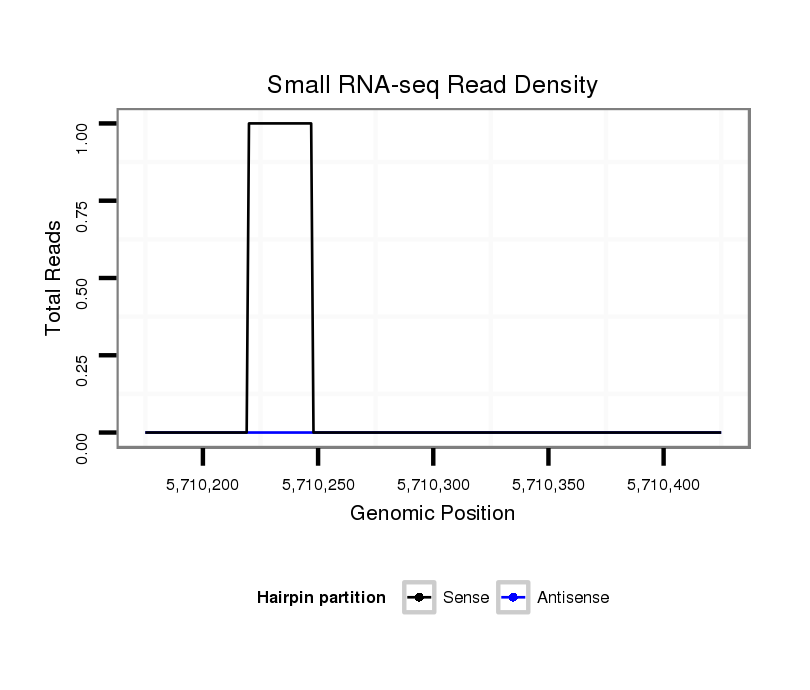
ID:dvi_5352 |
Coordinate:scaffold_12875:5710225-5710375 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_6847:1]; CDS [Dvir\GJ21493-cds]; intron [Dvir\GJ21493-in]
No Repeatable elements found
| -------------------------------###################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CCAACTGCGACTGCGGCGCTGGCAATTGAGGATGTGTCCAGACATTAGAGGTGAGTAGCGTCTAGCTTTTGGTTCTAAACTAAAAAATACCCTATACCAAATACCCTATAGTCTATATATATTTTGACAGCGTTTACTTTTAGAAAATTGACTTTTAAAATCTTTTGTCTTTATTGCCCAAATGTTATAAAACTTGAAACTTAAAGATAGCACTCAAATATGTGAATGCAACATAAGTACAGACTATACCT **************************************************..(((((((.....)))((((((......................)))))).........(((............))).(((.....((((((((.......)))))))).....))).(((((.((......)).)))))))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060679 140x9_testes_total |
SRR060681 Argx9_testes_total |
SRR060668 160x9_males_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|
| .............................................TAGAGGTGAGTAGCGTCTAGCTTTTGGT.................................................................................................................................................................................. | 28 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................................................................AGCGTTTACTTTGAAAAAATTA..................................................................................................... | 22 | 3 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 | 0 |
| ...............................................................................................................................AAGCGTTTACTTTGAAAAAATT...................................................................................................... | 22 | 3 | 8 | 0.13 | 1 | 0 | 0 | 1 | 0 | 0 |
| ......................................................................................................................................................................GTCTTGATTGCCGAAATCTT................................................................. | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 | 0 |
| ........................................................................................................................................CTTTTAGAACACCGACTTTT............................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
|
GGTTGACGCTGACGCCGCGACCGTTAACTCCTACACAGGTCTGTAATCTCCACTCATCGCAGATCGAAAACCAAGATTTGATTTTTTATGGGATATGGTTTATGGGATATCAGATATATATAAAACTGTCGCAAATGAAAATCTTTTAACTGAAAATTTTAGAAAACAGAAATAACGGGTTTACAATATTTTGAACTTTGAATTTCTATCGTGAGTTTATACACTTACGTTGTATTCATGTCTGATATGGA
**************************************************..(((((((.....)))((((((......................)))))).........(((............))).(((.....((((((((.......)))))))).....))).(((((.((......)).)))))))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060669 160x9_females_carcasses_total |
SRR060665 9_females_carcasses_total |
V116 male body |
SRR060666 160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|
| .....................................................TCGTCGCCGATCGAAAACCCA................................................................................................................................................................................. | 21 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 |
| .................................................CAACTCGTCGCCGATCGAAAA..................................................................................................................................................................................... | 21 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 |
| .........................................................................................................................................................................................ATATTTTGAGCTTAGAATTTG............................................. | 21 | 3 | 16 | 0.06 | 1 | 0 | 0 | 1 | 0 |
| ........................................................................................................................................................AAAATCTAAGAAAACAGAGAT.............................................................................. | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:5710175-5710425 + | dvi_5352 | CCAACTGCGACTGCGGCGCTGGCAATTGAGGATGTGTCCAGACATTAGAGGTGAGTAGCGTCTAGCTTTTGGTTCTAA---ACTAAAAAATACCCTATACCAAATACCCTATAGTCTATATATATTTTGACAGCGTTTACTTTTAGAAAATTGACTTTTAAAATCTTTTGT-------------------------------------------------CTTTATTGCCCAAATGTTAT--------AAAACTTGAAACTTAAAGATAGCACTCAA--ATATGTGAATGCAACATAAGTACAGACTATACCT |
| droMoj3 | scaffold_6496:24356654-24356814 - | GC---------------TCAGGCAATTGAGGTAATGGCCAGAAAGTGAGGTTAT----------------GGTGCCAATAAAT-----------GA--------------------------------------------TTTGAAAATACCCTG------------------TACTTA-----GTCAGAATTGATTGATAGGTTATTGAGTGA-------------------------CATAACATTGAAACATATCTTATACACAT-ACACATAAATATATATATATATAGTAT----------------- | |
| droGri2 | scaffold_14906:6034687-6034782 + | GG---------------TGC-------------------------------------------------------TAA---ATGAAAGAACCCCAAATACGAAATACCCTG--------------------------------------------------------------TAACAT-----ATTTGAATTAAATAGGTGGCCTTAAAAAGATTAT------------------------------------------------------------TATTTATTTGTTTTTTTT----------------- | |
| droWil2 | scf2_1100000004822:2693358-2693458 - | AA---------------ATTCCTAA--------------------------------------------------------------------------------------------------------------------------TAGTCAAAATAGTTAGTCATCTGCGGTATCATCTCTTATTTGAA------AACAGATATTTAAAAGATTTTATATATATTACATAACAG---------------------TTA-------------------------------TAAAA----------------- | |
| dp5 | 2:7585889-7585979 + | TA---------------ATATTTTT------------------------------------------------------------------------------------------------AAAATTTAAAAAAATTTAATT-------------------------------------------------------TATTAATTTTTAAAACTTTATTTTTTTATTTTTAAAATTTTTT--------CAGTTTTAAAAA-------------------------------TTAAA----------------- | |
| droPer2 | scaffold_4:3409499-3409556 - | CA---------------AATGTGAT--------------------------------------------------------------------------------------------------------------------------TAA-----------------------------------------------------------------------------------------------------ACTTAAAATTTAAATATGACAAATAAGCATAATTTAATGCATCAT----------------- | |
| droEle1 | scf7180000491199:1578804-1578853 + | TT---------------TAG------------------------------------------------------------------------------------------------------------------------TTTTAAAATCTTTTGTTCTAAAATCTTTCGT-------------------------------------------------TTTTAC-------------------------------TGA-------------------------------TCAAA----------------- | |
| droSim2 | x:8478723-8478759 - | TA---------------TTTGTGGA--------------------------------------------------------------------------------------------------------------------------TAATTTATTTTTACAATCTTTTGT--------------------------------------------------------------------------------------------------------------------------TTT----------------- | |
| droSec2 | scaffold_34:292942-292978 - | TA---------------TTTGTGGA--------------------------------------------------------------------------------------------------------------------------TAATTTATTTTTACAATCTTTTGT--------------------------------------------------------------------------------------------------------------------------TTT----------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
Generated: 05/16/2015 at 11:18 PM