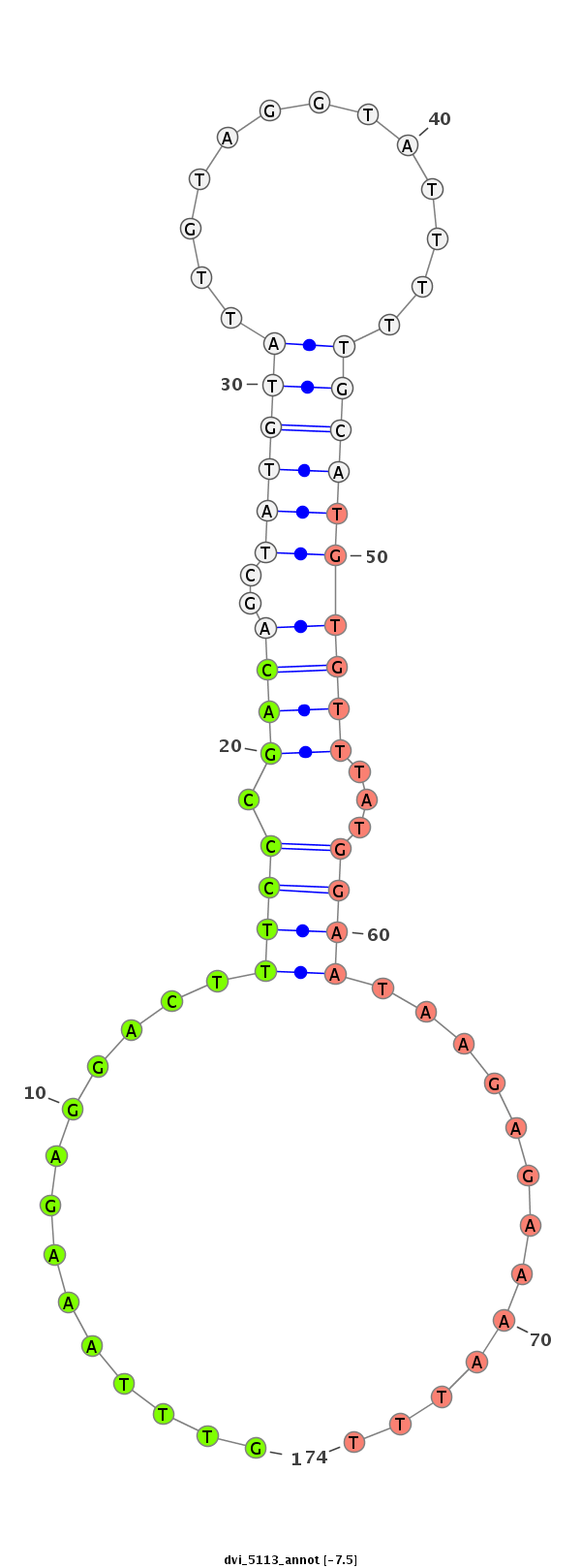
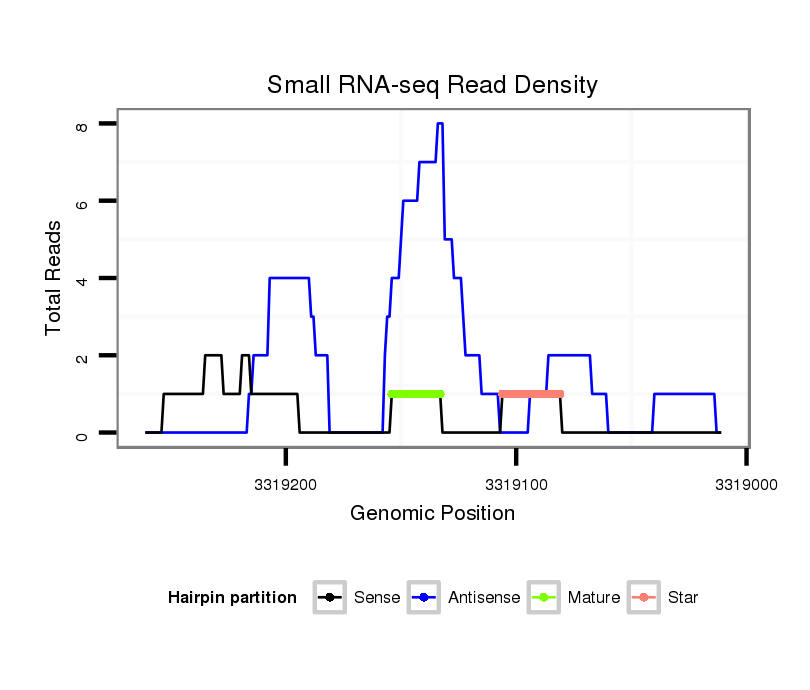
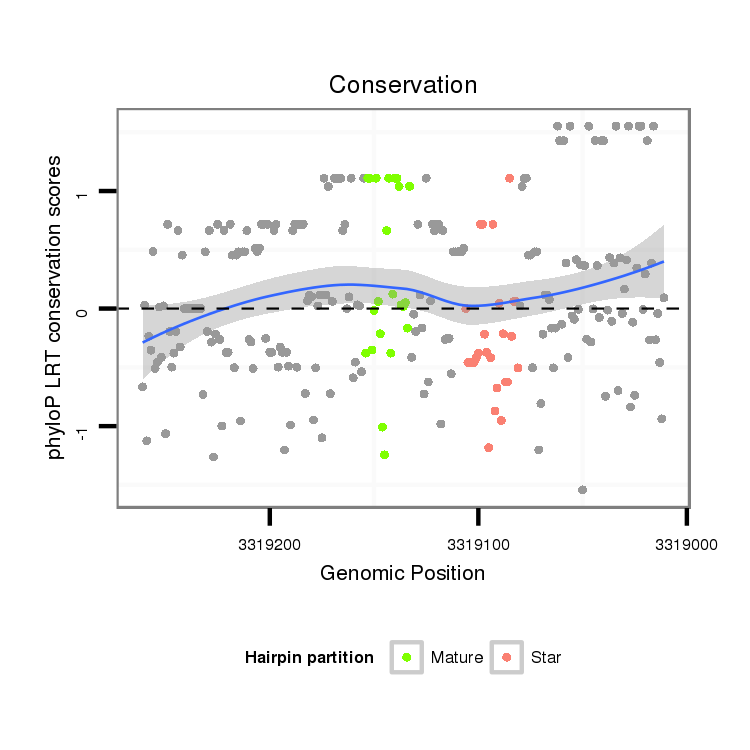
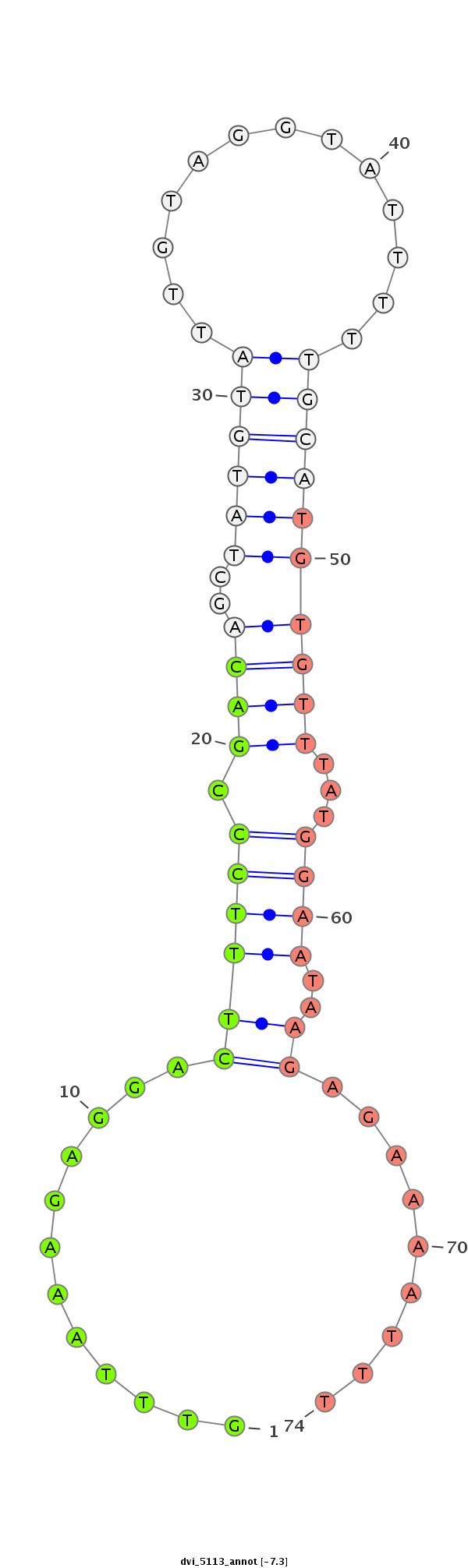
ID:dvi_5113 |
Coordinate:scaffold_12875:3319061-3319211 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -7.3 | -7.3 | -6.9 |
 |
 |
 |
CDS [Dvir\GJ20955-cds]; exon [dvir_GLEANR_6356:3]; intron [Dvir\GJ20955-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CCGTCCATGATGTCTAGGCGAACAGAATCATGGAGATTGGGCTCACAGATCTATAGTTATAGCGATGCCGAAGAATATTATGGTTTAATCAAAAATGTGGAGGAAAAGTTTAAAGAGGACTTTCCCGACAGCTATGTATTGTAGGTATTTTTGCATGTGTTTATGGAATAAGAGAAAATTTTCATCCTTTGGCAACTATAGAGAAAAATACGTTTATCGTGGTATGGTGACTATTGTAATACCCGACAACG ***********************************************************************************************************..............((((.((((..((((((.............))))))))))...)))).............********************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060682 9x140_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060679 140x9_testes_total |
SRR060689 160x9_testes_total |
SRR060687 9_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060681 Argx9_testes_total |
V116 male body |
M047 female body |
SRR060685 9xArg_0-2h_embryos_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060665 9_females_carcasses_total |
V053 head |
SRR060668 160x9_males_carcasses_total |
SRR060662 9x160_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................................................................................................................ATCCTTTGACCACTCTAGAGA............................................... | 21 | 3 | 1 | 6.00 | 6 | 4 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................CATCCTTTGACCACTCTAGAG................................................ | 21 | 3 | 3 | 2.33 | 7 | 2 | 1 | 0 | 1 | 1 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................ATCCTTTGACCACTCTAGAG................................................ | 20 | 3 | 5 | 2.20 | 11 | 3 | 3 | 0 | 2 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................ATCATGGAGATTGGGCTCAC............................................................................................................................................................................................................. | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........GATGTCTAGGCGAACAGAATCATGGA......................................................................................................................................................................................................................... | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................TCACAGATCTATAGTTATAGCGATG........................................................................................................................................................................................ | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................GTTTAAAGAGGACTTTCCCGAC.......................................................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................CATCCTTTGACCACTCTAGAGA............................................... | 22 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................TGTGTTTATGGAATAAGAGAAAATTT...................................................................... | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................ATCCTTTGACCACTCTAGA................................................. | 19 | 3 | 18 | 0.61 | 11 | 1 | 1 | 0 | 0 | 2 | 4 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................................................................................................................................CATCCTTTGACCACTCTAGA................................................. | 20 | 3 | 6 | 0.50 | 3 | 1 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................GCGGAAGAAAAGTTTAAAG........................................................................................................................................ | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...TCCACGATGTCCAGGCGAA..................................................................................................................................................................................................................................... | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................................CCTTTATGGTGGTATGGT....................... | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................................TAATCAAAGATGTGGACGA................................................................................................................................................... | 19 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................CATCCTTTGACCACTCTAG.................................................. | 19 | 3 | 13 | 0.15 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................................................................................TGGATTGTAGGTAATTTT................................................................................................... | 18 | 2 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .......................AGAATCATGGAGGTAGAGC................................................................................................................................................................................................................. | 19 | 3 | 19 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................CACCGTGGTATGGGGACT................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GGCAGGTACTACAGATCCGCTTGTCTTAGTACCTCTAACCCGAGTGTCTAGATATCAATATCGCTACGGCTTCTTATAATACCAAATTAGTTTTTACACCTCCTTTTCAAATTTCTCCTGAAAGGGCTGTCGATACATAACATCCATAAAAACGTACACAAATACCTTATTCTCTTTTAAAAGTAGGAAACCGTTGATATCTCTTTTTATGCAAATAGCACCATACCACTGATAACATTATGGGCTGTTGC
**********************************************************************..............((((.((((..((((((.............))))))))))...)))).............*********************************************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060677 Argx9_ovaries_total |
SRR060676 9xArg_ovaries_total |
SRR060655 9x160_testes_total |
SRR060674 9x140_ovaries_total |
SRR060673 9_ovaries_total |
SRR060675 140x9_ovaries_total |
SRR060678 9x140_testes_total |
SRR1106720 embryo_8-10h |
V047 embryo |
SRR060659 Argentina_testes_total |
SRR060667 160_females_carcasses_total |
M028 head |
M027 male body |
V053 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ........................................................................................................TTTCAAATTTCTCCTGAAAGGGCTGT......................................................................................................................... | 26 | 0 | 1 | 2.00 | 2 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................TCAATATCGCTACGGCTTCTTATAAT........................................................................................................................................................................... | 26 | 0 | 1 | 2.00 | 2 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................TGTCGATACATAACATCCATAAAAACG................................................................................................. | 27 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................CAAATTTCTCCTGAAAGGGCTGTCGAT..................................................................................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................GTCTAGATATCAATATCGCTACGGCTT................................................................................................................................................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................TATTCTCTTTTAAAAGTAGGAAACCGT......................................................... | 27 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................AGTTGTTATACCTCCTTTTCTA............................................................................................................................................. | 22 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................................CATACCACTGATAACATTATGGGCTGT... | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................GAAAGGGCTGTCGATACATAACATCCA......................................................................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................TTCTCCTGAAAGGGCTGTCGATACATA................................................................................................................ | 27 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................TTTCTCCTGAAAGGGCTGTCGATACAT................................................................................................................. | 27 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................TTTAAAAGTAGGAAACCGTTGATATC.................................................. | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................CTAGATATCAATATCGCTACGGCTTCT................................................................................................................................................................................. | 27 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TTCAAATTTCTCCTGAAAGGGCTGT......................................................................................................................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................AGTTTGTACACCTCATTTTCGA............................................................................................................................................. | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................CTCCTTTTCAAATTGATCC..................................................................................................................................... | 19 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................................CTGATAAGATTATAGGCT..... | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................AGTTTTGAGACCTCCTTTGCA.............................................................................................................................................. | 21 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................................................CCTAAAGGGGCTGTCGTTA.................................................................................................................... | 19 | 3 | 15 | 0.13 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 |
| ..........TCAGATCGGCTTGTCATAG.............................................................................................................................................................................................................................. | 19 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .GCACGTACTATAGATCAGC....................................................................................................................................................................................................................................... | 19 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:3319011-3319261 - | dvi_5113 | CCGTCCATGA-TGTCTAGGCGAACAGAATCATGGA--------GATTGGGCTCACAGATCTATAGTTAT-AGCGATGCCGAAGAATATTATGGTTTAATCAAAAATGTGGAGGAAAAGTTTAAAGAGGACTTTCCCGACAGCTATGTATTGTAGGTA--------------TTTTTGCATGTGTTTATGGAATAAGAGAAAATTTTC-----ATCCTTTGGCAACT----ATAGAGAAAAATACGTTTATCGTGGTATGGTGACTATTGTAATACCCGACAACG |
| droMoj3 | scaffold_6496:21823380-21823626 + | TCCTT---AT-TTTCGAGGAG---------CTGGG--------CATATGATTCGCACATCAG----TACGAATGAGTTTACTGACTATGTTGAATTGATCCGAAATGTCGAAGCAAAATTCAAGGCTGATTTTCCCGACTGCTATATCATGTAAGAGCTGTACTCTATATACATTT--------ATATAACTTTCTTGCCAACCATCGTCTAATCCCTTACTAATT----TTAGAGAGAGTTACTCGTATCGTGGCTACGTGGTTATCGTAATACCCGACAAAG | |
| droGri2 | scaffold_15112:2824676-2824916 + | CCAACCTCAACCATTTCAGC------------AGGGATAAAGGTTTTAGATT-------CTGAAGTTATGAATGATGTGACCGAATATGATGTACTCATCCAAAATGTGGAAAAGCAATTTCAAATCGATTTTCCAAGCAGTTACATCTTGTATGTA--------------T----GCA-ACACAGATGAGTTGAGCACCATTTACCATCTAAT-----CCCAACCCTTGTCAGAGAGAAGAACACGTCTCATGGCTTGGTCACCATAGCAATAGCCGACTTCG | |
| droBip1 | scf7180000396427:1589570-1589621 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCGAAAGGTTTACCTACCGCGGAATAATCATCTTTAGCATTCCAAAGGGGG | |
| droKik1 | scf7180000302228:69933-70080 - | GAGAT---AT-TT---------------------------------------------------------------------------TAAAATTAAAACAAAAAT--GAATTAATAGTTCAAAAA--ACCTTCACGGCTC--TCCTTT---------TGTAATTTAT-------------------------ATGTATTATTTTTC-----AT--------TTCC----ACAGTGAAAAGTTCTCCTTTCGAGGAATGATTACCTTTGTCATACCCAAGGAAA | |
| droBia1 | scf7180000302291:3160589-3160640 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCGCGAAGTACCCCTACCGAGGCATGATTACCTTCTGCATTCCGAACGGGA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
Generated: 05/16/2015 at 07:56 PM