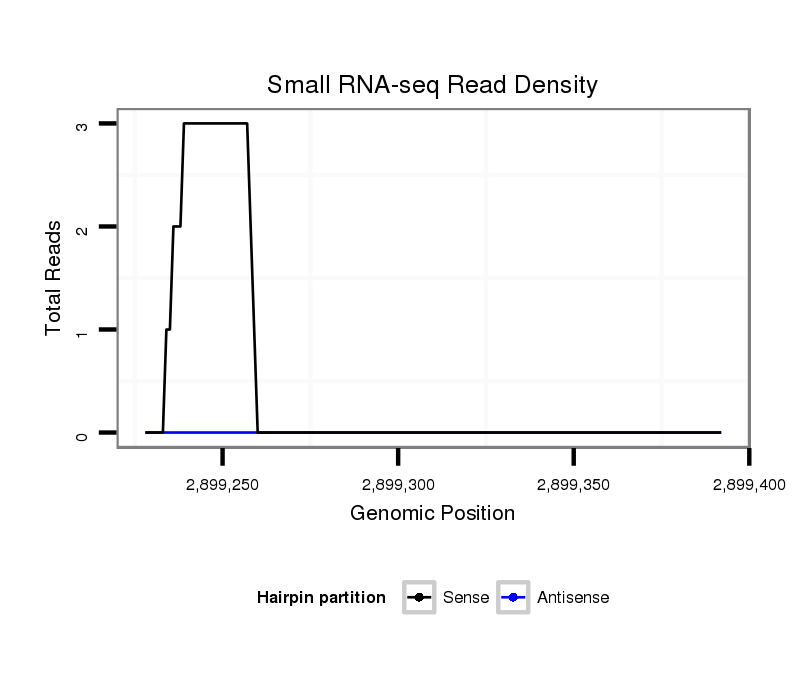
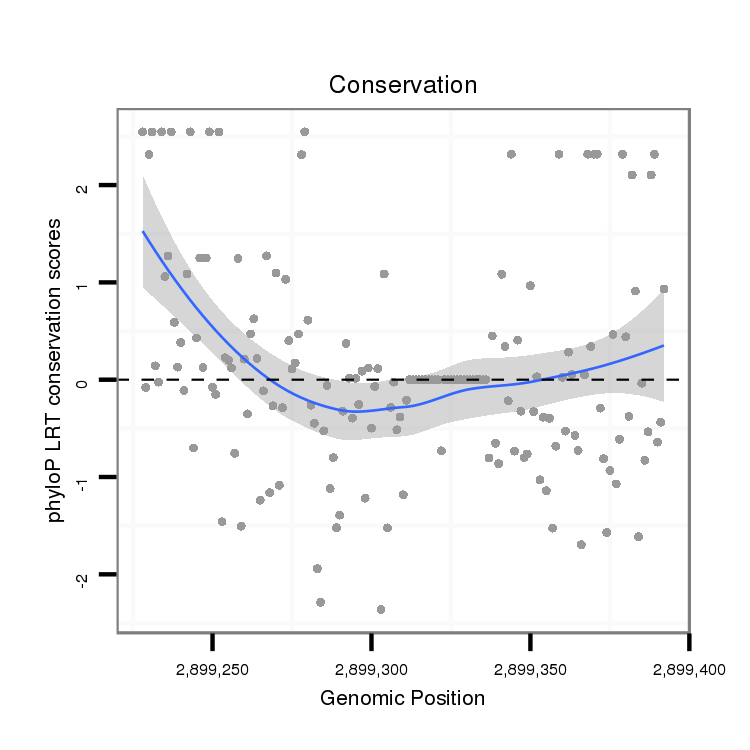
ID:dvi_5073 |
Coordinate:scaffold_12875:2899278-2899342 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ21363-cds]; CDS [Dvir\GJ21363-cds]; exon [dvir_GLEANR_6725:3]; exon [dvir_GLEANR_6725:2]; intron [Dvir\GJ21363-in]
No Repeatable elements found
| ##################################################-----------------------------------------------------------------################################################## AGGACAACATATTGGTGAAGAAACTTTTTTTGTATAATATATCACCAGGGGTAAGCGTTTGGCAACAATTTCATATTCCTTGAATATTTAAGATAAACGAATTTTCCTGCATTAGCTATCGGCCAAGCAATTGAGAGCCTATTTCAACACGTTCGGACGCGTCGA **************************************************.....((((((........((((.......)))).........))))))................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060656 9x160_ovaries_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060676 9xArg_ovaries_total |
SRR060679 140x9_testes_total |
SRR060682 9x140_0-2h_embryos_total |
GSM1528803 follicle cells |
SRR060659 Argentina_testes_total |
V116 male body |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ................................TATAATCTACCGCCAGGGGT................................................................................................................. | 20 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................GTCCAAGAAATTGAGAGCC.......................... | 19 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........ATATTGGTGAAGAAACTTTTTTT...................................................................................................................................... | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......ACATATTGGTGAAGAAACTTTTTTTG..................................................................................................................................... | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........TTGGTGAAGAAACTTTTTT....................................................................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................GTCCAAGAAATTGAGAGCCG......................... | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................................................................................................................................................AATACCTTCGGACGCGT... | 17 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........ATTCCTGAAGAAACTGTTTTT...................................................................................................................................... | 21 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...........TTGGTGAAGAAATTGTCTTT...................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TCCTGTTGTATAACCACTTCTTTGAAAAAAACATATTATATAGTGGTCCCCATTCGCAAACCGTTGTTAAAGTATAAGGAACTTATAAATTCTATTTGCTTAAAAGGACGTAATCGATAGCCGGTTCGTTAACTCTCGGATAAAGTTGTGCAAGCCTGCGCAGCT
**************************************************.....((((((........((((.......)))).........))))))................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M027 male body |
SRR060669 160x9_females_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060689 160x9_testes_total |
GSM1528803 follicle cells |
|---|---|---|---|---|---|---|---|---|---|---|---|
| .....TGGTCTAACCACTTGTTTGAA........................................................................................................................................... | 21 | 3 | 5 | 0.40 | 2 | 0 | 0 | 1 | 1 | 0 | 0 |
| ............................................................CCGTTGTCAAAGAAGAAGGAAC................................................................................... | 22 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................AACTCTCGGTGAAAGTTGCGC.............. | 21 | 3 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................CCGTTGTCAAAGAAGAAGGAA.................................................................................... | 21 | 3 | 12 | 0.17 | 2 | 2 | 0 | 0 | 0 | 0 | 0 |
| ..CTGTTGTATACCCGCTTTTTT.............................................................................................................................................. | 21 | 3 | 12 | 0.17 | 2 | 0 | 0 | 0 | 0 | 2 | 0 |
| ....................................................................................ACAAGTTCTCTTTGCTTAAAA............................................................ | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:2899228-2899392 + | dvi_5073 | AGGACAACATATTGGTGAAGAAACTTTTTTTGTATAATATATCACCAGGGGTAAGC-GTTTGGCA------------A----CAATTT--CAT------A-----TTCCT---------------------------------------------TGA-ATATTTAAGATAAACGAATTTTCCTGCATT---AGCTATCGGCCAAGCAATTGAGAGCCTATTTCAACACGTTCGGACGCGTCGA |
| droMoj3 | scaffold_6496:22309169-22309341 - | AGGATCACATTCTGGTGAAAAAGCTTTATATCATCAGTGTAGCTGCAGCAGTAAGT-AGTAAAA--ACAATTCT--------------TA---TTGTTATTATTGG-CCCATAT-----------------ATCAATGT----ATGG---TTTT-ATA-T----------C--------------AATGTTAAGATATCGTCCAAAAACTTGAGAGATTATTTCACCACTTTCGGAGCCGTGAA | |
| droGri2 | scaffold_15112:3260013-3260180 - | AGGACGACATATTGATCAAAAAACTATTTCTATTCAATGTGGCACCAGAGGTATGA-AAAAGCA--ACG--CGA--------------AC---TCATCGATTGTA--ATTTCAG-----------------CTGTGCAT----TTTA---TTCA-ATT-A--------------------------ATTCACAGCTGGTGCCAAAGCAGCTGGAAGCCTATTTCGTGAAATTTGGTTCTGTGAA | |
| droWil2 | scf2_1100000004954:3226370-3226423 + | AGGATGACATTCTAGTCCAGGAGTTATATCTGCACAACATACCAGCAGAGGTAA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | 3:19267932-19268099 + | AAGACCACATTCCAGTGCTGAAACTGTATTTATACAAGATCCCACCGGATGTAAGT-GTCCGCG--TCC--CCC--------------TT---TTGTCAAGCTTT--ACTCTGA-----------------AACAATAC----ACTG---TCCT-GTC-T--------------------------CATCGCAGCTAAATCAATCTACGCTGCATTCGCACTTCTCAAACTATGGACGCGTTTT | |
| droPer2 | scaffold_37:270858-271025 + | AAGACCACATTCCAGTGCTGAAACTGTATTTATACAAGATCCCACCGGATGTAAGT-GTCCGCG--TTC--CCC--------------TT---TTGTCAAGCTTT--ACTCTGA-----------------AACAATAC----ACTG---TCCT-GTC-T--------------------------CATCGCAGCTAAATCAATCTACGCTGCATTCGCACTTCTCAAACTATGGGCGCGTTTT | |
| droAna3 | scaffold_13266:10715815-10715970 + | AAGACCATATTCCCGTGAAAAAGATCTATCTGTACAAGGTGCCGCCGGAGGTGCCA--------------------------AGACCA--CTC------G-----CTCCCTAGA-----------------AATCATCA----ACTG---ATCACTCCTC-------------------------TTTTTGCAGTTGAATCAAAATGTCTTGGAGGAGCACTTTAATGTGTACGGTCGTGTGCT | |
| droBip1 | scf7180000396428:1144805-1144959 + | AAGACCACATTCCCGTCCAGAAGCTTTACCTGTATAAAGTGCCAGCGGAGGTGCCA--------------------------AGACCA--CTT------G-----TTTCCTAGA-----------------AATCATGA----ATTG---ATAATTCCTC--------------------------TCTTGCAGTTAAGTCAAAATGTCTTGGAGCTGCATTTCTGTGGGTACGGCCGAGTAGT | |
| droKik1 | scf7180000302682:1019336-1019496 + | AGGACCACATTCCAGTGCAGAAGATCCACATGTTCGGCTTGCCGCCGAAGGTAGTT-GCTCATCA------------GTTC------A--TCT------G-----CTGGTTCGG-----------------TTCTATA-ATCCAAT------CTTTCA-T--------------------------TCGTTTAGCTAAACAAGTTTGCTTTGGAGGTTCACTTTGGCATGTACGGTCATGTCCT | |
| droFic1 | scf7180000453858:524597-524769 - | AAGACCACATTAAAGTGCAGAAACTAAGTTGGTGCGATGCCCCAAGGGAGGTAGCCTTTTTA--A------------GATT------G--TCTGCAGT-G-----T------TATAAAATTCGTTTCCTTATCCA----CTCGTTAAATG---A-TTTTC--------------------------TGCAGAGTTTAAATAAGAGCTCGATACAAGCATACTTTGAAAGCTTTGGGCGCGT--- | |
| droEle1 | scf7180000491211:527152-527321 - | AGGACCACATTCCCGTGCAGAAGCTCTACTTCAGCAAGGTGCCTCCGGAGGTTGCGTCTTTC--T------------GGCCA-CGT--TGTTT------G-----T-----------CCTCTGTGTGCCTGGCTA----ACCCACTGTCG---A-GCC-C--------------------------TTTTGCAGCTGAACCAAAGCATTCTTGAGCATCACTTCCGGATCTATGTGCGCGTGCT | |
| droRho1 | scf7180000777716:13658-13816 + | AGGATCACATTCCCGTGCAGAAGGTCCACATTTACAATGTTTTGCCGAAGGTAGCGTTTTTC--T------------GG--------------------------G-----------CCTTTGGGTACCTCGTTA----ACCCATTGTTG---A-TTCTC--------------------------TCCAGAAGTTGAACAAAAAAATGCTTGAGCATTACTTTGGGATCTATGGACGCGTGCT | |
| droBia1 | scf7180000301506:2432892-2433056 + | AGGACCACATTCCCGTGCAGAAGCTCTACTTTTACAGGGTTCCTCCAGGGGTAGGT-CCTCC--T------------GCTC------C--TCA------G-----C-----------CCTTTACATACGCCACTA----AACCCCTGCTA---A-TCATC--------------------------TCCTGCAGCTCAGACAGGCCGATTTAAGGAAGCACTTCGGGATCTACGGGACTGTGCT | |
| droTak1 | scf7180000415346:220120-220286 - | AGGACCACATTCCCGTCCAGAAGCTCTACTTCTACAAGGTTCCGCCGGAGGTAGCGTGTTTC--T------------GGCCAACGCCT-----------------------------CTTTCGTTTACCCCACTA----ATCCCCTGCCG---A-TCCTC--------------------------TCCTGCAGCTCAGCCAGGCCATTTTGGAGCAGCACTTTGGGATCTACGGGCACGTGCT | |
| droEug1 | scf7180000409663:88485-88644 + | AAGACCACGTTCCCGTACAGAAGCTGTACTTCTACAAGGTTTCGCCGCAGGTACAT-GTACAATATAGCACCTTTGTGG-----------------------------T--------------------CCACTA----ATTTTCTGTTG---A-CCTTC--------------------------TCATGCATCTGAATCAGAGCATTCTGGAGCACCACTTTGAGGCCTATGGGCGCGTGCT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
Generated: 05/19/2015 at 01:15 PM