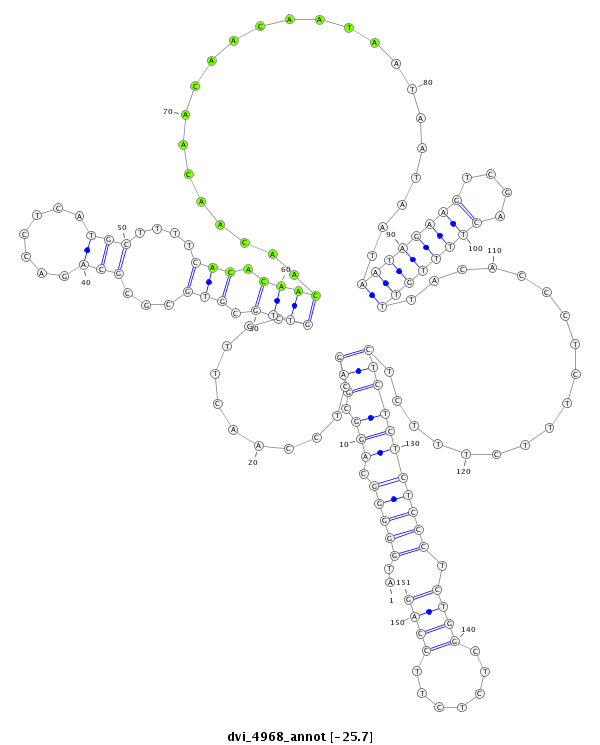
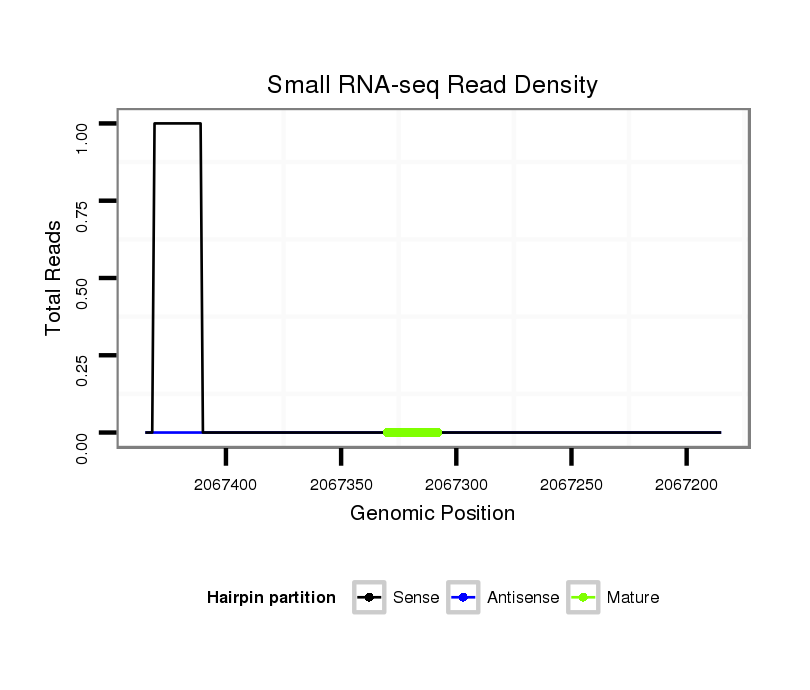
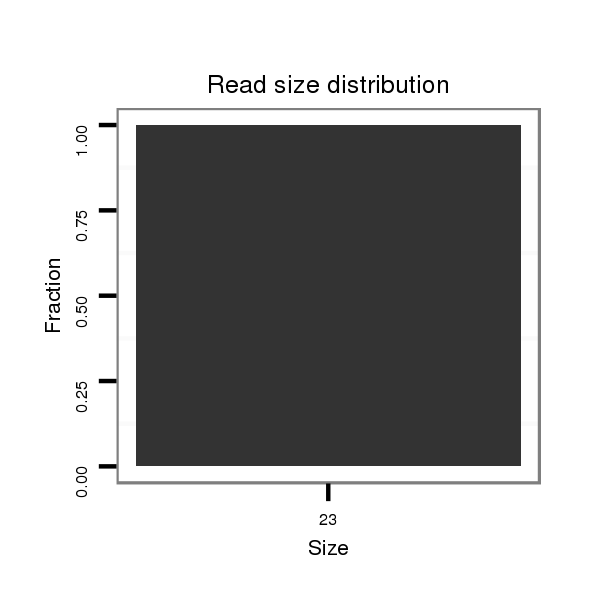
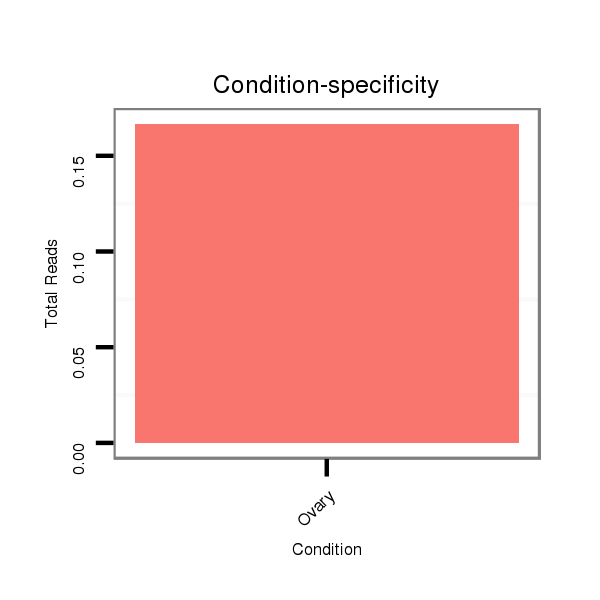
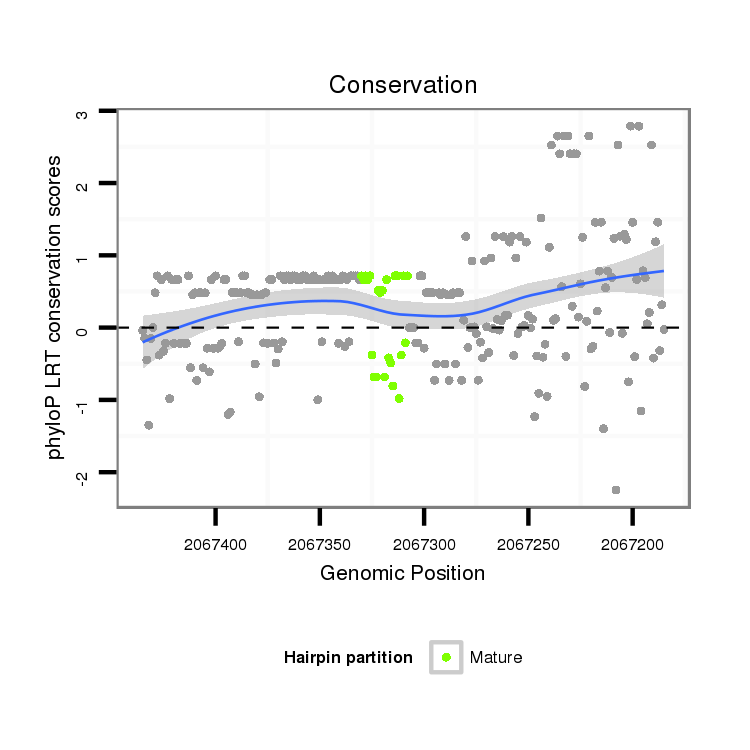
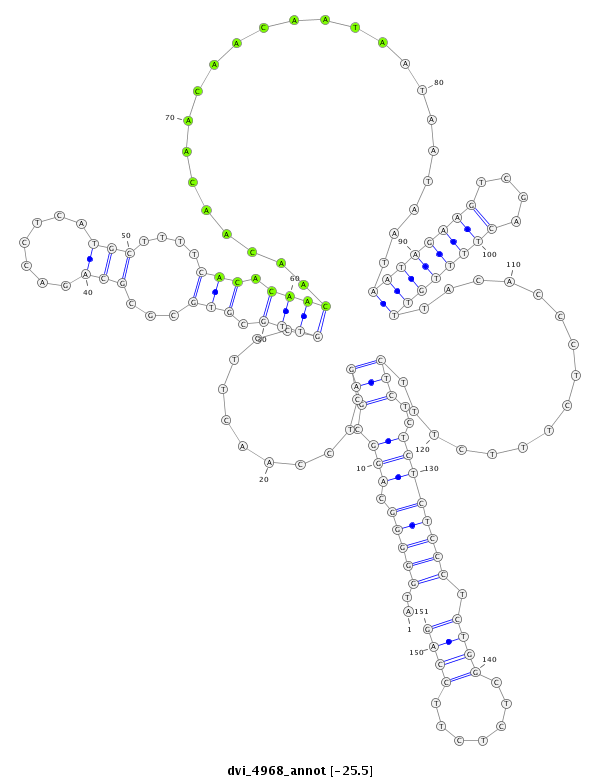
ID:dvi_4968 |
Coordinate:scaffold_12875:2067235-2067385 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -25.5 | -25.4 | -25.0 |
 |
 |
 |
exon [dvir_GLEANR_6429:2]; CDS [Dvir\GJ21035-cds]; intron [Dvir\GJ21035-in]
| Name | Class | Family | Strand |
| GA-rich | Low_complexity | Low_complexity | + |
| (TTG)n | Simple_repeat | Simple_repeat | + |
| mature | star |
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## AGGGTATTTGCGAGCAGGTAACATCTCATATAGGAAATGGGCAGGGGTTAATGGGGGCAGGCGAGCTCCAACTTCCGTTGCGTGCGCGCAGACCTCATGCTTTTCACACAACAACAACAACAACAATAATAATAATAATAGAAGTCGACTTTTGTTTACACCCTCTTTCTTTCTCTCTCTCTCCCTCTGGCTCTCTTCCAGCTCTGTGGCACGCTGCTGATTGTGGCAGGCTGTTATCTGTATACGGATAG **************************************************..(((((.(((.(((...........((((.(((...(((.......)))....))).))))........................((((((((....))))))))..................))))))))))).((((.......))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060676 9xArg_ovaries_total |
SRR060682 9x140_0-2h_embryos_total |
V116 male body |
M047 female body |
V047 embryo |
SRR060684 140x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................................................................................................................................................................GGCACGCTGCTGTTTGTGGA........................ | 20 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ....TATTTGCGAGCAGGTAACATC.................................................................................................................................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................ATAATAGAATTGGACGTTTG................................................................................................. | 20 | 3 | 16 | 0.19 | 3 | 0 | 0 | 2 | 0 | 0 | 1 |
| .........................................................................................................ACACAACAACAACAACAACAATA........................................................................................................................... | 23 | 0 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................TATTGGACATGGGAAGGGGT........................................................................................................................................................................................................... | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ............................TAGAGGAGATGGGCAGGG............................................................................................................................................................................................................. | 18 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ......................................................................................................................................ATAATAGAATACGACGTTTG................................................................................................. | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ............................................................................................................................................................................................GGTTCTCTTCCACCCCTGT............................................ | 19 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
|
TCCCATAAACGCTCGTCCATTGTAGAGTATATCCTTTACCCGTCCCCAATTACCCCCGTCCGCTCGAGGTTGAAGGCAACGCACGCGCGTCTGGAGTACGAAAAGTGTGTTGTTGTTGTTGTTGTTATTATTATTATTATCTTCAGCTGAAAACAAATGTGGGAGAAAGAAAGAGAGAGAGAGGGAGACCGAGAGAAGGTCGAGACACCGTGCGACGACTAACACCGTCCGACAATAGACATATGCCTATC
**************************************************..(((((.(((.(((...........((((.(((...(((.......)))....))).))))........................((((((((....))))))))..................))))))))))).((((.......))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
V116 male body |
SRR060658 140_ovaries_total |
M047 female body |
SRR060678 9x140_testes_total |
M028 head |
SRR060672 9x160_females_carcasses_total |
M027 male body |
SRR060666 160_males_carcasses_total |
SRR1106729 mixed whole adult body |
M061 embryo |
SRR060660 Argentina_ovaries_total |
SRR060662 9x160_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..............................................................................................................................................................................................................ACCGTGCGTCGACGAACAC.......................... | 19 | 2 | 7 | 1.29 | 9 | 9 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................CTGTCCGCACGAGGTTGA.................................................................................................................................................................................. | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................AAGGTCGGGACACAGGGCGA.................................... | 20 | 3 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................TTATTATTATTATTATCTGCA.......................................................................................................... | 21 | 1 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................AGAAAGAGAGAGAGAG.................................................................... | 16 | 0 | 20 | 0.15 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................AAATGTGGGAGATACAGAG.............................................................................. | 19 | 3 | 20 | 0.15 | 3 | 0 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............TCGTCGATTGTACAGTAT............................................................................................................................................................................................................................. | 18 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................AGTACGAATAGTGTCTTG........................................................................................................................................... | 18 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................AGGGATGCCGAGAGAAGTT................................................... | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................AGGAAAAATGTGTTGTTGTGG..................................................................................................................................... | 21 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................................CCGTCCGCCCGAGCTTG................................................................................................................................................................................... | 17 | 2 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................................................................................TGTTGTTGTTGTTGTTATTA......................................................................................................................... | 20 | 0 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................TTGTTGTTGTTGTTGTTATTAT........................................................................................................................ | 22 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................................................................GAGCTTGAAGGCACCGGA........................................................................................................................................................................ | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ........................................................................................................................................................................GAAAGAGAGAGAGAGGGAGACA............................................................. | 22 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................GGTTGAAGGCACCACACT...................................................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..........................................................................................................................................................AAATGTGGGAGATACAGAGA............................................................................. | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................TTGTTGTTGTTATTATTAT..................................................................................................................... | 19 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:2067185-2067435 - | dvi_4968 | AGGGTATTTGCGAGCAGGTAACATCTCATAT----AGGAAATGGGCAGGGGT--TAATGGGGGCAGGCGAGCTCCAACTTCCGTTGCGTG---CGCGCAGACCTCATGCTTTTCACACAACAACAACAACAACAATAATAATAATAATAGAAGTCGACT------TTTGTTTACACCCTCTTTCT----TTCTCTCT--------CTCTCCCTCTGGCTCTCTTCCAGCTCTGTGGCACGCTGCTGATTGTGGCAGGCTGTTATCTGTATACGGATAG |
| droMoj3 | scaffold_6496:23188441-23188687 + | AGGGTATTTGAAAACAGGTAATAACTAATTTGTGGGCGGAGAAGGTG---GTTGTAATGGAGCCA-ACGAGCTCCAACTTCCGTTGCGTG---AGCGCAGACCTCAAGCTTTTCACACAA------CTCGAATAATA---ATAACAATATGAGTTGCCTCCCTCTCTTTCTCACACACACTCTCT----CTCTCTCT--------CTCTCTCTCTC---TCCCTCTAGCTCTGTGGCACACTGCTGATTGTGGCTGGCTGTTATCTCTACACGGACAA | |
| droGri2 | scaffold_15112:4187262-4187416 - | AGGAAA-TGGCGATCTGGAGTCA--------GTGGGGGGAGTGGGGCGGGA---TA--------GGGCGGCATTCAACTTCCGTTGCGTGTTGTGTGCAGACCACATGTTTTTCACACACACACACCTCGAAACACA---GCAAC-------------------------------------------------------------------------------------------------ATCGAAAGTAAACGA---CTGTCTTCATACTCTGTT | |
| droWil2 | scf2_1100000004558:1798986-1799082 - | CTCTCT-------------------------------------------------------------------------------------------------------------------------------------------------------------------CT------------GT----TGCTCTCTCTCTATAAT--TCTCTTAATCTCTCTTCCAGCTATGTGGCATGGTTTTGATAGTTTCTGGCTGTTATCTCTATGCCGATAA | |
| dp5 | 3:10259532-10259587 + | T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTCAGCTTTGTGGTATTCTACTAATTTTATCCGGCTGCTACCTGTTTACCGACAA | |
| droPer2 | scaffold_4:5590982-5591047 + | TCTAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATCATTTTCAGCTTTGTGGTATTCTACTCATTTTATCCGGCTGCTACCTGTTTACCGACAA | |
| droAna3 | scaffold_13337:21518784-21518843 - | TT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTTTTAGATATGTGGCTTGCTGTTGATTGCATCGGGGTGCTACCTGTATTCCGACGG | |
| droBip1 | scf7180000396730:50182-50248 - | CTCGAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTCTCTTTCAGATATGTGGCTTGCTACTAATTGCTTCGGGCTGCTACCTGTATGCCGACGG | |
| droKik1 | scf7180000302808:147990-148050 - | CT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTGTTTCAGATATGTGGGTTACTGTTGGTCCTATCAGGTTGCTACCTCTACTCCGACAC | |
| droFic1 | scf7180000453784:146336-146395 + | TT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TATTTCAGATCTGCGGCTTGCTGCTGATCGTTTCTGGCTTGTACATATTTTCCGACCA | |
| droEle1 | scf7180000491255:2276730-2276785 + | T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TACAGATATGCGGTTTATTGATGATCGTATCCGGATTGTACCTGTTCTCTGACAA | |
| droRho1 | scf7180000780188:5514-5577 + | TGTTT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCTTTCCAGATATGTGGCTTATTGCTGATCGTATCAGGATTATATCTCTTTTCCGACAA | |
| droBia1 | scf7180000302428:6088516-6088622 - | TTCCTT-------------------------------------------------------------------------------------------------------------------------------------------------------------------CTCTCGAAGTCCTTCTCCTGTGATTCCT--------GTATGTCATTTTGTATCTTTCAGATATGTGGCTTGCTGCTGATTGCATCGGGCCTGTATATCTTTTCGGATAA | |
| droTak1 | scf7180000415825:108045-108104 - | TC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTTTTCAGATATGTGGCCTACTGCTGATTGTATCAGGCCTGTACATCCTTTCGGATAA | |
| droEug1 | scf7180000409456:13645-13697 + | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGCTTTGTGGCTTAATGCTGATCGTATCCGGCCTTTATATCTTTTCGGATAA | |
| dm3 | chr3L:13837107-13837164 + | TT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTCAGATATGTGGCTTGCTGCTGGTAGTATCAGGTCTATATATCTTCTCCGATAA | |
| droSim2 | 3l:13508745-13508802 + | TT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTCAGATATGTGGCTTGCTTCTGGTAGTATCAGGTCTATATATCTTCTCCGATAA | |
| droSec2 | scaffold_0:5980236-5980293 + | TT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTCAGATATGTGGCTTGCTGCTGGTAGTATCAGGTCTATATATCTTCTCCGATAA | |
| droYak3 | 3L:13919585-13919644 + | TT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTTTTAGATATGTGGCTTGCTGCTGGTGGTATCAGGTCTATACATCTTCTCCGATAA | |
| droEre2 | scaffold_4784:13827507-13827562 + | T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTAGATCTGCGGCTTGCTGCTGGTTGTATCAGGTCTATATATCTTCTCCGACAA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 04:41 AM