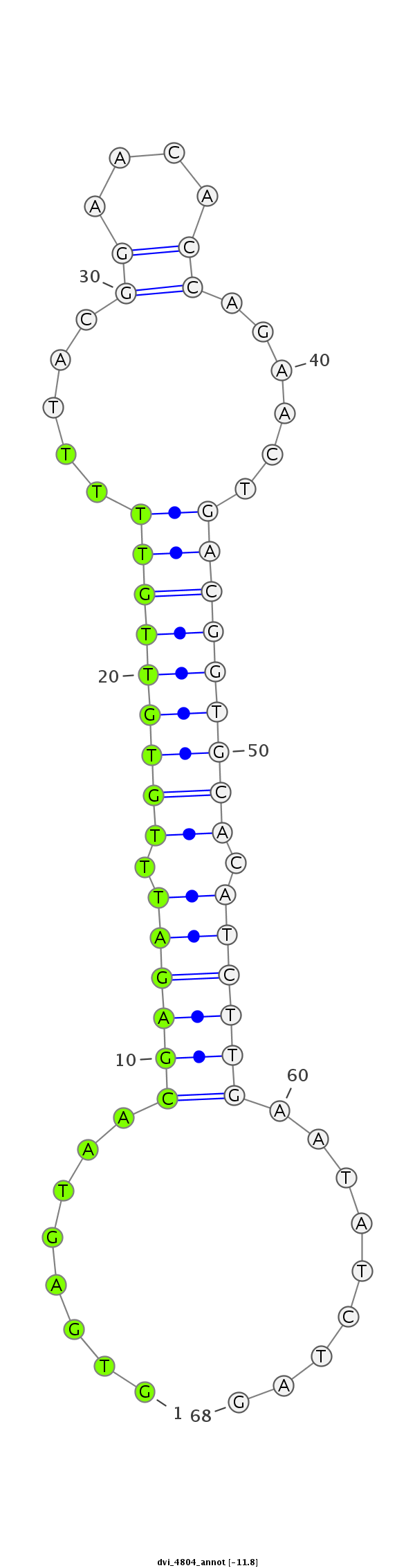


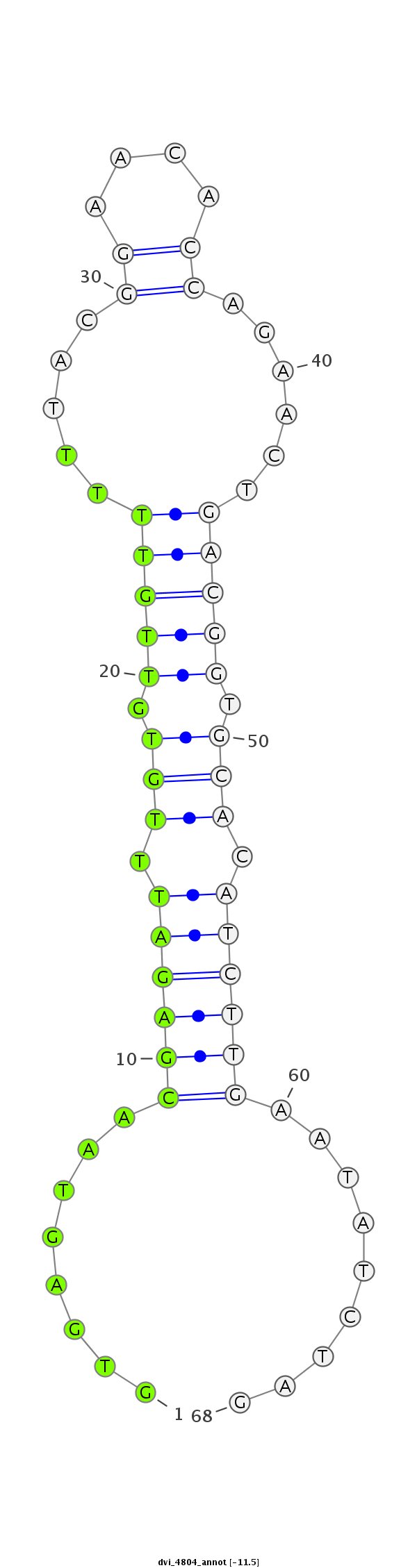
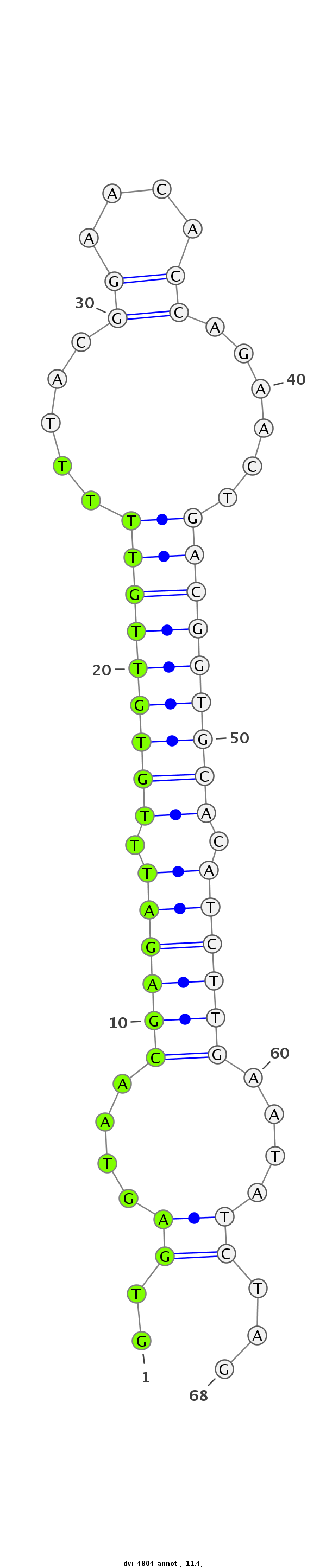
ID:dvi_4804 |
Coordinate:scaffold_12875:1028443-1028510 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -11.6 | -11.5 | -11.4 |
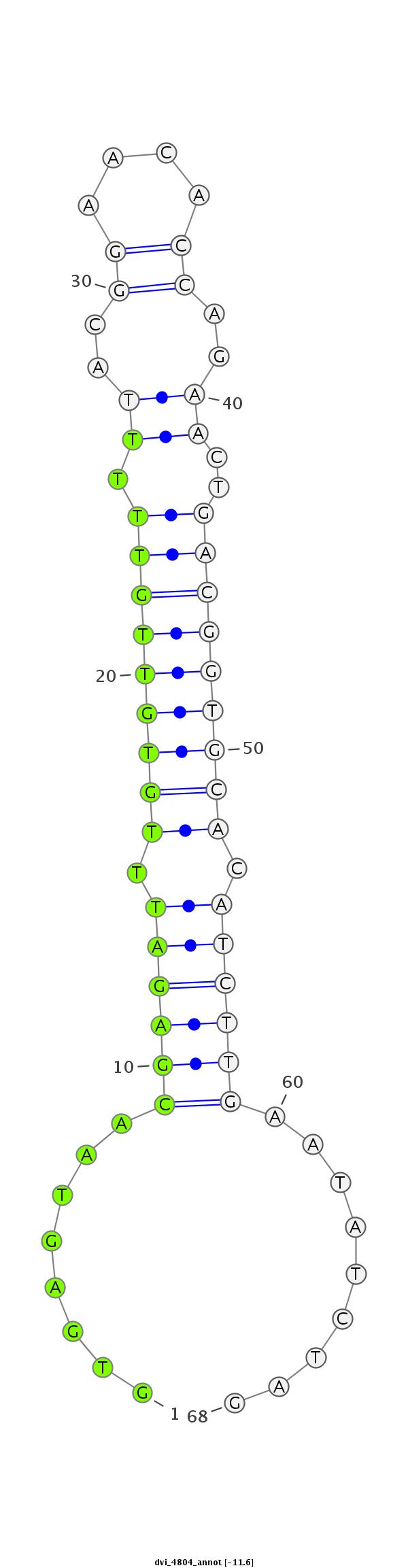 |
 |
 |
exon [dvir_GLEANR_6500:1]; CDS [Dvir\GJ21115-cds]; exon [dvir_GLEANR_6500:2]; CDS [Dvir\GJ21115-cds]; intron [Dvir\GJ21115-in]
No Repeatable elements found
| --------------------------------------############--------------------------------------------------------------------################################################## ATTTTTGGTTGAAATTCATAGAGCACAACAACAAAATAATGTTGAATCATGTGAGTAACGAGATTTGTGTTGTTTTTACGGAACACCAGAACTGACGGTGCACATCTTGAATATCTAGAAATTGCTGTGCTGCGCCTGCTTCCTGCTCCTGCTGGCCGCCATTGGCAG **************************************************........((((((.(((((((((.....((....))......))))))))).)))))).........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060678 9x140_testes_total |
SRR060655 9x160_testes_total |
SRR060679 140x9_testes_total |
SRR060660 Argentina_ovaries_total |
M027 male body |
SRR1106729 mixed whole adult body |
V116 male body |
SRR060666 160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................TTGAATCATGTGAGTAACGAG.......................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................CATAAATTGCTGTGCTGCGCCTG.............................. | 23 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................GTGAGTAACGAGATTTGTGTTGTTTT............................................................................................ | 26 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................GAATGTCTAGAAATTGCTGTG....................................... | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................................AACGAGATTTGTGTAGCT.............................................................................................. | 18 | 2 | 12 | 0.25 | 3 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................CTTGATTTCTGCTCCTGCTGG............. | 21 | 3 | 10 | 0.20 | 2 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 |
| ...........................................................................TTACGGCACGCCAGAAC............................................................................ | 17 | 2 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..........................................................................................................TTGAAGATCGAGAAATTGGT.......................................... | 20 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...............................................................................................................TAACTTGAAATTGCTGTCCT..................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TAAAAACCAACTTTAAGTATCTCGTGTTGTTGTTTTATTACAACTTAGTACACTCATTGCTCTAAACACAACAAAAATGCCTTGTGGTCTTGACTGCCACGTGTAGAACTTATAGATCTTTAACGACACGACGCGGACGAAGGACGAGGACGACCGGCGGTAACCGTC
**************************************************........((((((.(((((((((.....((....))......))))))))).)))))).........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
V047 embryo |
M027 male body |
|---|---|---|---|---|---|---|---|---|
| ............................GTTGTTTTATTTCAACTTGGT....................................................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 |
| ................................................................................................................................................CGAGGACCAACGGCGGTAA..... | 19 | 2 | 6 | 0.17 | 1 | 0 | 1 | 0 |
| .......CAACTTTAAGTAACAGGTGTT............................................................................................................................................ | 21 | 3 | 10 | 0.10 | 1 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12875:1028393-1028560 - | dvi_4804 | ATTTTTGGTTGAAATTCATAGA----GCA-CAACAA----C-AAAATAATGTTGAATCATGTGAGT--AACGAGATTTGTGTTGTTTT-TACGGAACACCAGAA---CTGACG-GTGCACATC-TTGAATATCTAGAAATTGCTGTGCTGC---GCCTGCTTCCTGC--------TCCT---GCTGGCCGCCATTGGCAG |
| droMoj3 | scaffold_6496:4086431-4086579 - | ATTC---GAGCAAAA---AAGA----AAA-C---AAATATCAAAAATCATGTTGAATCATGTGAGT--AACGAGAATTGATTT----GATATCGGTTAAGAGAA---CTGACT-TTGCA----------TATCTAGAAATTTCTCCT--------CCTGC---------TGGTGGTGCTCCTGTGCGCCAGCTTCAGCTT | |
| droGri2 | scaffold_15245:9942402-9942557 + | ATTT---GTTGAAATTCACAGAACTTGCA-CAA--------------CATGTTGAATCATGTGAGT--AACGAT---TGTTTTCTTTTGTATAGAATTAAGGAA---CTGATT-GTGCAAATCCTTGAATCCCTAGAAACTGCTCAACTGC---GCCTGCTTCC--TACT------------GTTCCTCGGTCTGGTCAG | |
| droEle1 | scf7180000491214:4424061-4424188 + | TGTTTTAGATC-------CAGTAGCTGCTCCAA--------------AATGCTGAAATACGTGAGTTTAATCGGATT----------TAAAGG-GTCACATTAG---TTGACCAATGTCTATC-------------------------TTCAGGCCCTTATTCTTGCCTT------------GCTGGTGGTTTTGGCCAA | |
| droYak3 | 2R:20517475-20517587 - | A-----------------------------------------------ATGCTGAAATATGTAAGTTCAATCGGATT----------TGCATCGGA-ATATTGGACAT-----------------TGACTAATCAGTCACCTCTCCACTGCAGACCCTCATCTTTGCCCT------------GTTCGTCGCTTTGGCCAG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/16/2015 at 10:38 PM