
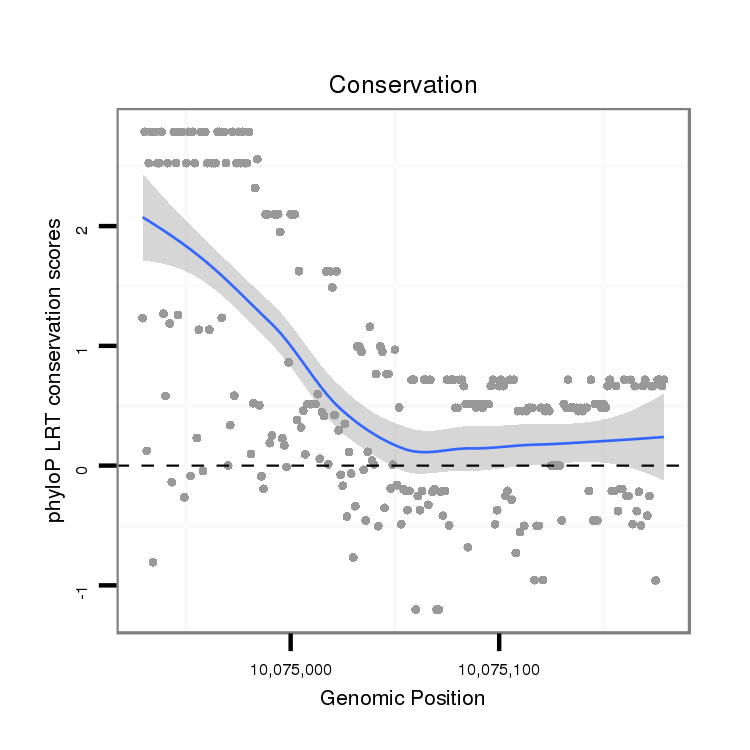
ID:dvi_4634 |
Coordinate:scaffold_12855:10074979-10075129 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_10994:2]; CDS [Dvir\GJ11115-cds]; intron [Dvir\GJ11115-in]
| Name | Class | Family | Strand |
| AT_rich | Low_complexity | Low_complexity | + |
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- TTGCTATGCACGGCGTCTATTCTTAGTTTATGTGCCATCAGTTTAGACAGGTTCGTTTTAAATAATTGTATAATATTTATTTATTTGATATCGTGAGTGAAAATAGAGTTACAGTCCGTCATCGCCACATTCTGTATGTCGCGAATAAATTATACTCATAGATGTATGTTCGAGTATATGCACCCTTTGGTAGGAGTATTTATGTTAATGACTAATATATGTGGAAACTCGTATACCTGACTTAGCTAACA **************************************************..............((((((...(((((((((((........))))))))..)))....))))))......((((.((((.....)))).)))).......((((((.....(((((((......)))))))((....))...))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060682 9x140_0-2h_embryos_total |
SRR060678 9x140_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
SRR060668 160x9_males_carcasses_total |
V053 head |
V116 male body |
SRR060669 160x9_females_carcasses_total |
SRR060664 9_males_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060665 9_females_carcasses_total |
SRR060666 160_males_carcasses_total |
SRR060659 Argentina_testes_total |
SRR060679 140x9_testes_total |
SRR060680 9xArg_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .............................................................................................................................................................................................................................TGGAAACTGGTAGACTTGA........... | 19 | 3 | 19 | 1.47 | 28 | 7 | 0 | 10 | 1 | 0 | 0 | 5 | 0 | 0 | 2 | 0 | 0 | 1 | 1 | 1 |
| ...............TCTATTCTTAGTTTATGTGCCATCA................................................................................................................................................................................................................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................ATACTAACAGATGTAGGTTCGA.............................................................................. | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................GACTAATATATGTGAAAA........................ | 18 | 1 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................GACTAATATATGTGAAAAT....................... | 19 | 2 | 10 | 0.40 | 4 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 |
| ..............................................................................................................................................................................TAGATGCACGATTTGGTAGGA........................................................ | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................AAGACTAATATATGTGAAA......................... | 19 | 2 | 13 | 0.23 | 3 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................AAGACTAATATATGTGAAAA........................ | 20 | 2 | 9 | 0.22 | 2 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................TCTCGTGGGTGAAATTAGA................................................................................................................................................ | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................TACAGGTTCGTGTTAAAAA........................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................TGGTATCGTGACTGAGAA.................................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................GACTAATATATGTGAAAT........................ | 18 | 2 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
AACGATACGTGCCGCAGATAAGAATCAAATACACGGTAGTCAAATCTGTCCAAGCAAAATTTATTAACATATTATAAATAAATAAACTATAGCACTCACTTTTATCTCAATGTCAGGCAGTAGCGGTGTAAGACATACAGCGCTTATTTAATATGAGTATCTACATACAAGCTCATATACGTGGGAAACCATCCTCATAAATACAATTACTGATTATATACACCTTTGAGCATATGGACTGAATCGATTGT
**************************************************..............((((((...(((((((((((........))))))))..)))....))))))......((((.((((.....)))).)))).......((((((.....(((((((......)))))))((....))...))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
SRR060678 9x140_testes_total |
V116 male body |
SRR060664 9_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|
| ...........................................................................................................................CGTTGTAAGAGATACAGGGC............................................................................................................ | 20 | 3 | 5 | 0.80 | 4 | 4 | 0 | 0 | 0 |
| ...............................................................................................................................................................................TATACGTGGACAACCATACTC....................................................... | 21 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 |
| ...........................................................................................................................CAGTGTAAGTCCTACAGCGC............................................................................................................ | 20 | 3 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 |
| ...........................................................................................................................CGTTGTAAGACCTACAGGGC............................................................................................................ | 20 | 3 | 7 | 0.14 | 1 | 1 | 0 | 0 | 0 |
| ...........................................................................................................................CGTTGTAAGAAATACAGGGC............................................................................................................ | 20 | 3 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 |
| ...................................................................................................................GGCAGCAGAGGTGTAAGTCA.................................................................................................................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 1 | 0 |
| ....................................................AGCACAGTTTATTAACATAG................................................................................................................................................................................... | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 |
| ...........................................................................................................CACGGTCAGGAAGTAGCGG............................................................................................................................. | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 1 | 0 |
| ...........................................................................................................................CGGTGTAAGAGCTACAGGGC............................................................................................................ | 20 | 3 | 10 | 0.10 | 1 | 1 | 0 | 0 | 0 |
| ............................................................................................................................GTTGTAAGAGATACAGGGC............................................................................................................ | 19 | 3 | 18 | 0.06 | 1 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12855:10074929-10075179 + | dvi_4634 | TTGCTATGCACGGCGTCTATTCTTAGTTTATGTGCCATCAGTTTAGACAGGT--TCGTT--TTAAATAATTGTATA---ATATTT-ATTTATTTGATATCGTGAGTG--AAAA--TA-GAGTTA---------------------------------------CAGTCCGTCATCGCCA---------------CATTCTGTATGTCGCGAATA---AATTATACTCATAGATGTATGTTCG--------AGTATATGCACCCT-----TTGGTAGGAGTATTTATGTTAATGACTAATATATGTGGAAACTCGTA-TACCTGACTTAGCTAACA |
| droMoj3 | scaffold_6540:4034596-4034805 + | TTGCTGTGCACGGCATCCATTCTAAGTTTGTGTGCCATAAGCTTGGACAGGT--GAGTA-ACGAAGCAATT--TTA---ATTTGTTG-----TAG---------------------------------------------------------------------------------CCAAGTCTCTGAAGAACATATTTCATATTTTGTAATTA---CATTATAC------------GTGTGCATGTATGAGCATGTTCTCTCTTGACTTTCACACGAG--------TTAATGACTAAT-------GAAACTCGTG-CACCTACCTAGGCAAACA | |
| droGri2 | scaffold_14830:1267687-1267952 + | TTGCTATGCACGGCATCCATTCTTAGTTTGTGTGCCATCAGCTTGGACAGGT--AAGTAA-TTAAAAAAT-AAAAAATAAT-----ATTTATACCGTTTCATATGTC--AAGGAGTACGAATTGCATACACATTCCCTTGACAGCGAATGCCTTCAATTCATA--TTC---CATTGGTG---------------TGTTGTACATGTCAGCTTCAACTAAT--TACTAATAGATGTATGTGTGTGTGTGTGAGTGTGT---------------------------GTGT---------GTGTGTGTGGAGGCGTATATTAGCGGACTAAGCCAACA | |
| droWil2 | scf2_1100000009706:989-1040 + | CTCCTTTGCACGGCCTCCATTCTGAGTTTATGTGCCATCAGCGTTGACAGGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | 2:25936758-25936813 - | CTGCTCTGCACGGCCTCCATTCTCAGTCTGTGCGCCATCAGCGTGGACAGGT--ACGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_6:1270326-1270381 - | CTGCTCTGCACGGCCTCCATTCTCAGTCTGTGCGCCATCAGCGTGGACAGGT--ACGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_13340:10835226-10835322 - | CTTCTCTGCACCGGCTCCATTCTTAGTCTCTGCGCCATCAGTCTGGACAGGT--GAGTTTGTGAACTAATTTCTTA---ATTTGTCG-----TTGATGTCATAGTTG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droBip1 | scf7180000396374:250731-250827 + | CTGCTCTGCACCGGATCCATCCTCAGCCTCTGCGCCATCAGTCTGGACAGGT--GAGTTTGTGAACTAATTTCTTA---ATTTGTCG-----TTGATGTCATAGTTG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droKik1 | scf7180000302247:464832-464923 - | CTGCTGTGCACCGGCTCCATCCTCAGCCTGTGCGCCATCAGTCTGGACAGGT--GAGTTGGTGAACTAATTTCTTA---ATTTGTCG-----TTGATGTCAT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000453912:1910229-1910285 - | CTCCTGTGCACCGGCTCCATCCTCAGTCTGTGCGCCATCAGTCTGGACAGGT--GAGTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droEle1 | scf7180000491080:2713569-2713625 - | CTGCTCTGCACCGGCTCCATCCTCAGCCTGTGCGCCATCAGTCTGGACAGGT--GAGTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droRho1 | scf7180000777337:36436-36492 - | CTGCTCTGCACCGGCTCCATCCTCAGCCTGTGCGCCATCAGTCTGGACAGGT--GAGTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droBia1 | scf7180000302113:3849111-3849221 - | CTGCTCTGCACCGGCTCCATCCTCAGCCTGTGCGCCATCAGCCTGGACAGGTAAGAGTTGGCCAACTAATTTCTTA---ATTTGCCG-----TTGATGTCATAGCTATGG---------T-CTG---------------------------------------T-------GGTC---------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000414009:417822-417897 - | CTGCTCTGCACCGGCTCCATTCTCAGCCTGTGCGCCATCAGTCTGGACAGGT--GAGTTGGCCAACTAATTTCTTA---AT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409562:89626-89744 + | CTGCTATGCACTGGGTCCATTCTCAGCCTGTGCGCCATCAGTCTGGACAGGT--GAGTTGGCCAACTAATTTCTTA---ATTTGTCG-----TTGATGTCATAGCAATGGCCA--TA-GA-CTG---------------------------------------TAGTCAGTC------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dm3 | chr3R:13566781-13566856 + | CTGCTCTGCAGCGGCTCCATCCTCAGCCTGTGCGCCATCAGTCTGGACAGGT--GAGTTGGCCAACTAATTTCGTA---AT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | 3r:7700853-7700928 - | CTGCTCTGCACCGGCTCCATTCTCAGCCTGTGCGCCATCAGTCTGGACAGGT--GAGTTGGCCAACTAATTTCGTA---AT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_12:594296-594371 - | CTGCTCTGCACCGGCTCCATCCTCAGCCTGTGCGCCATCAGTCTGGACAGGT--GAGTTGGCCAACTAATTTCGTA---AT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | 3R:2144743-2144818 + | CTGCTCTGCACCGGCTCCATCCTCAGCCTTTGCGCCATCAGCCTGGACAGGT--GAGTTGGCCAACTAATTTCGTA---AT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4770:8063770-8063845 - | CTGCTCTGCACCGGCTCCATCCTCAGCCTGTGCGCCATCAGTCTGGACAGGT--GAGTTGGGCAACTAATTTCGTA---AT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 08:08 PM