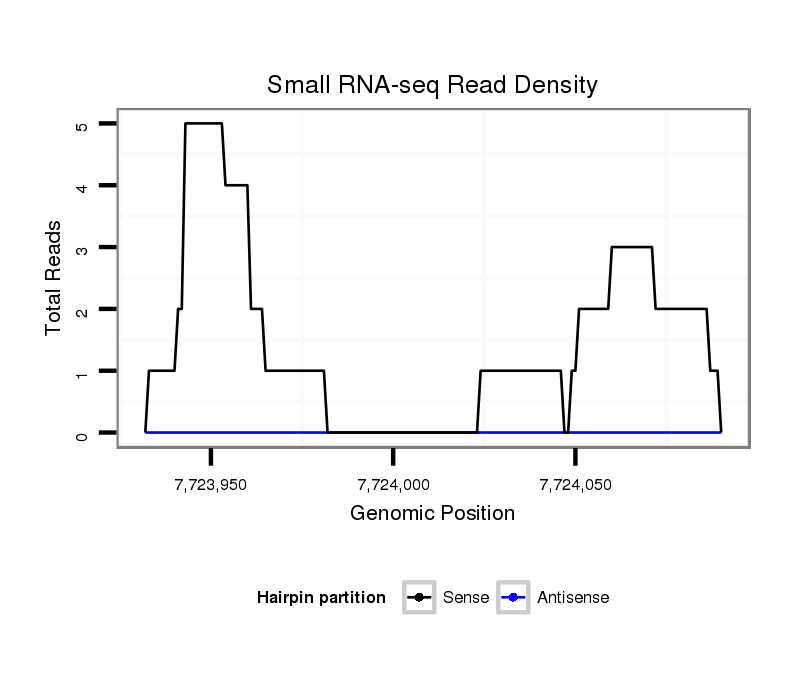
ID:dvi_4376 |
Coordinate:scaffold_12855:7723982-7724040 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_10895:2]; CDS [Dvir\GJ11010-cds]; exon [dvir_GLEANR_10895:1]; CDS [Dvir\GJ11010-cds]; intron [Dvir\GJ11010-in]
No Repeatable elements found
| ##################################################-----------------------------------------------------------################################################## ACCAATCGGCTATTCGGACGTTATGCTACAACTTGGCGGCCAGGAGGGGCGTAAGTGCAACGGCTTACTATATGTTTTAGTATTAATCATATTATTTTTTGCATTCTAGAACGCTCACCATATGCCGTTTACTTTGAACAGCCACGCGATGCGCTGCTG **************************************************.............((......((((...((((..........))))....))))...))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M028 head |
SRR060683 160_testes_total |
M047 female body |
SRR060657 140_testes_total |
SRR060666 160_males_carcasses_total |
SRR060670 9_testes_total |
SRR060678 9x140_testes_total |
SRR060679 140x9_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR1106727 larvae |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........ATTCGGACGTTATGCTAC.................................................................................................................................. | 18 | 0 | 1 | 2.00 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .CCAATCGGCTATTCGGACGTT......................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................CCATATGCCGTTTACTTTGAAC.................... | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................TATTTTTTGCATTCTAGAACGCT............................................ | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........ATTCGGACGTTATGCTACAACT.............................................................................................................................. | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........CTATTCGGACGTTATGCTACAACTTGGCGGCCAGGAGGGGC............................................................................................................. | 41 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...........................................................................................................................................AGCCACGCGATGCGCTGCT. | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................TGAACAGCCACGCGATGCGCTGCCG | 25 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................ATATGCCGTTTACTTTGAACA................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .............................................................................................................................................CCACGCGATGCGCTGCCG | 18 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................................................................TTACTTTGAACAGCCACGCGATGCGCT.... | 27 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................ACTTGGCGGCCAGGAGGGGCA............................................................................................................ | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
|
TGGTTAGCCGATAAGCCTGCAATACGATGTTGAACCGCCGGTCCTCCCCGCATTCACGTTGCCGAATGATATACAAAATCATAATTAGTATAATAAAAAACGTAAGATCTTGCGAGTGGTATACGGCAAATGAAACTTGTCGGTGCGCTACGCGACGAC
**************************************************.............((......((((...((((..........))))....))))...))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M027 male body |
V116 male body |
SRR060680 9xArg_testes_total |
SRR060659 Argentina_testes_total |
SRR060679 140x9_testes_total |
SRR060683 160_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060676 9xArg_ovaries_total |
M047 female body |
M061 embryo |
SRR060657 140_testes_total |
SRR060665 9_females_carcasses_total |
SRR060666 160_males_carcasses_total |
SRR060670 9_testes_total |
SRR060681 Argx9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..........................................................................................................ATGTTGGGAGTGGTATAC................................... | 18 | 2 | 3 | 3.00 | 9 | 2 | 2 | 1 | 1 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................ATGTTGGGAGTGGTATACA.................................. | 19 | 3 | 20 | 1.25 | 25 | 13 | 5 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 |
| .........................................................................................................................................TGTCGGGGCCCTACGCTAC... | 19 | 3 | 12 | 0.08 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................AACAAAATTAGTGTAATAAAAAA........................................................... | 23 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................GGAATACGAGGTTGAACGG.......................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................GGCGACTGCTATACGGCAA.............................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12855:7723932-7724090 + | dvi_4376 | ACCAATCGGCTATTCGGACGTTATGCTACAACTTGGCGGCCAGGAGGGGCGTAAGTGCAACGGCTTAC-TATATG-TTTT----------------AGTAT--------------------TAATCATATTATTTTTTGCATTCTAGAA---CGCT--CAC-------------CATATGCCGTTTACTTTGAACAGCCACGCGATGCGCTGCTG |
| droMoj3 | scaffold_6540:24942475-24942652 - | ACCGATTGGATATTCTGATGTCATGCTACAGCTTGGCTCCCGAGAAGCCCGTAAGTGGATTTGCAA----------ACTAAATA-GAATAATGGCAT-CACTATTTATAATTCACTTGATTTGATTTGA---TTGCA---T-TCTAGAA---CGAT--CAC-------------CATATGCCGTCTACTTTGAACAGCCACGTGAATCGCTGCCG | |
| droGri2 | scaffold_15074:5160685-5160843 - | CCCCATTGGCTACTCCGACGTCATGGTACAGCTTGGGTCCAGCGAGCGGGGTAAGTGAATC---TCAGACTGCTAAAC-AAAAATGTG------CATGTTC--------------------AAATAT-T---TAAAT---A-CTTTGCAGAACGTT--CACCCTACATAC----------CCGTTTACT------TGCAACGCGAGAGGCTGTCG | |
| dp5 | 2:25781796-25781955 + | TTCCATGTCGTACGGAAATGTGGGTCAGAATCCGG---ACCAGGAGACCCGTAAGGGGGAGT-CTTAG-TTCCAG-TCCAGTTATGGG-------TAATAC--------------------TAATATTT---TGGAA---C-CCTTGCA---GATGGCGGCCCTGCGTATACTCTATACTCCATA---TATGAACAGCCGCGTGAT--------- | |
| droPer2 | scaffold_6:1142174-1142333 - | TTCCATGTCGTACGGAAATGTGGGTCAGAATCCAG---ACCAGGAGACTCGTAAGGGGGAGT-CTTAG-TTCCAG-TCCAATTATGGG-------TAATAC--------------------TAATATTT---TGGAA---C-CCTTGCA---GATGGCGGCCCTGCGTATACTCTATACTCCATA---TATGAACAGCCGCGTGAT--------- | |
| droFic1 | scf7180000453488:247180-247321 + | A-----------TGGCAGTGTGGCCACGAGTCCGGCCAGCCAGGAAACCCGTAAGGTCGCCT-CTT--------G-TCCAGTGCCAGA-------TAGTAG--------------------TCATTTTT---TCGGT---T-TCTTGCA---GATT--CCC-------------AATATGCCATGTACTATAATCAGCCGCACGACTCGCTTTCG | |
| droEle1 | scf7180000491080:2110646-2110723 + | T-----------------------------------------------------------------------------------------------AGTAC--------------------TAATCCTC---TGGTTTGCC-TCCTGCA---GATT--CCC-------------AGTATGCCATGTACTATAATCAGCAGCGGGACTCGCTTTCA | |
| droRho1 | scf7180000779338:69464-69616 + | CTCGATGGCTTACGGCGGTGTGACCTCGAGTCCGGGCGGCCAGGATACCCGTAAGCTGG----------TCCCTGGCCACGTTTGAGA-------TAGTAC--------------------TAATCCTA---TTGGC---C-TCTTGCA---GATT--CCC-------------AGTATGCCATGTACTATAATCAGCAGCGGGACACGCTTTCC | |
| dm3 | chrX:3064120-3064154 + | C------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CATTTACTTGTTACAGGCTCGCCATCGCCGGCCA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
Generated: 05/16/2015 at 09:59 PM