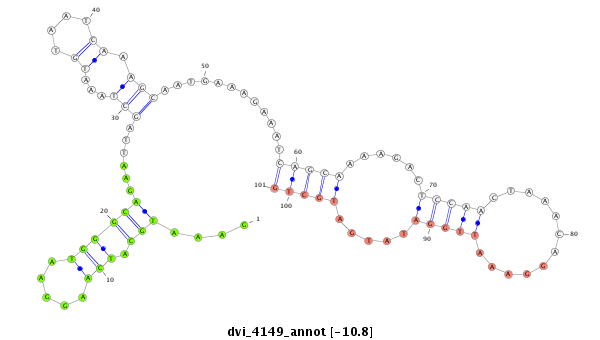
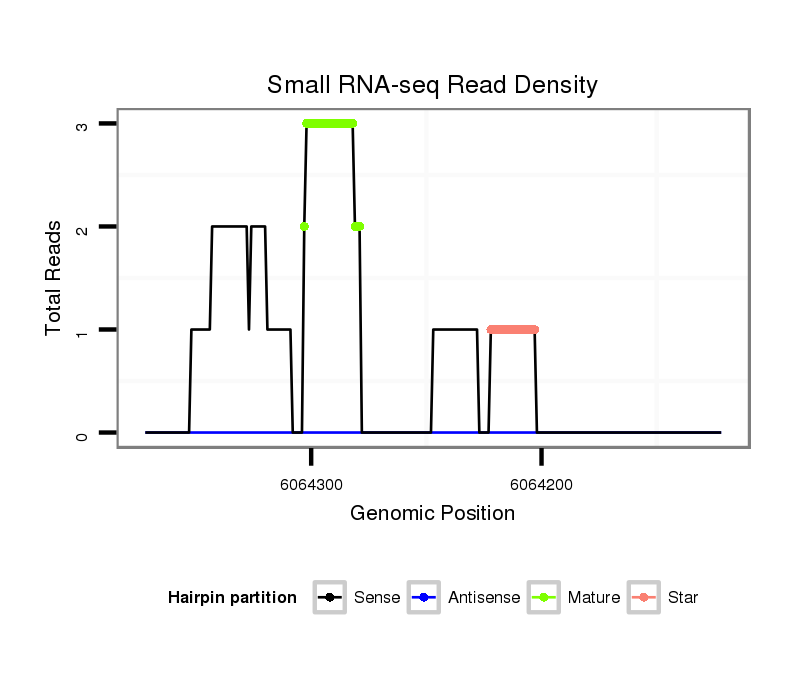
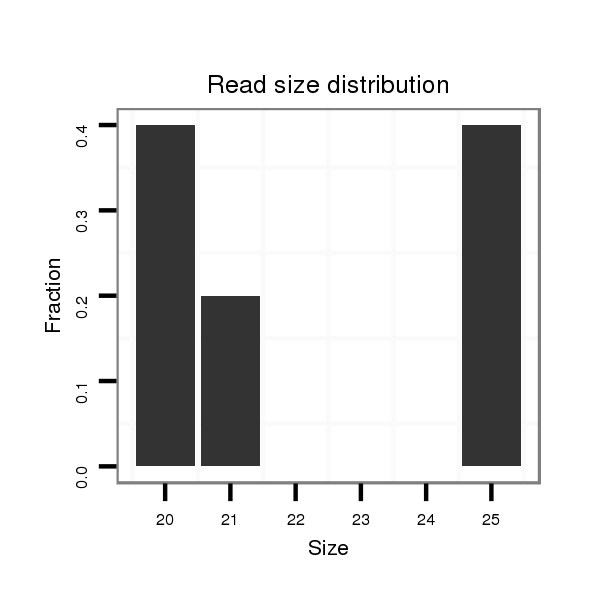
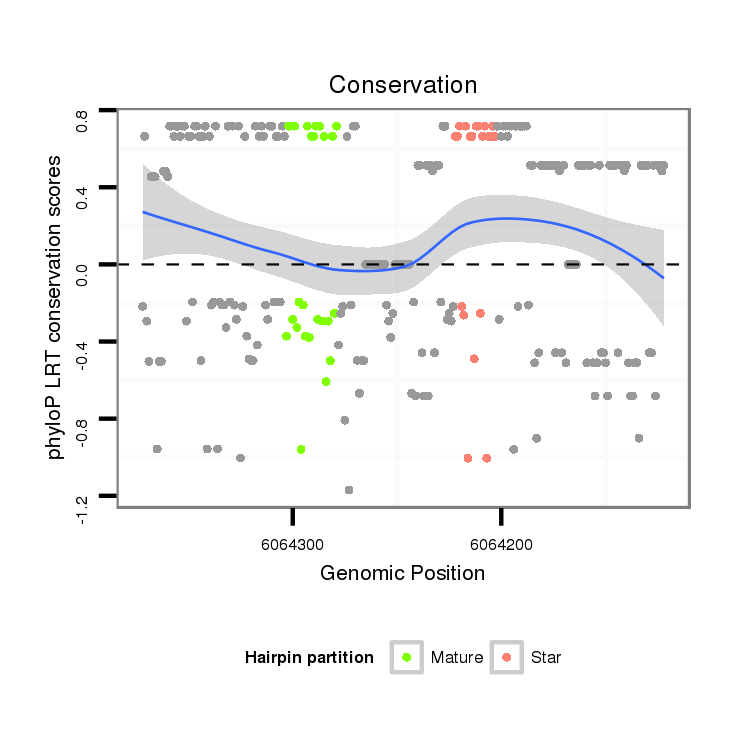
ID:dvi_4149 |
Coordinate:scaffold_12855:6064172-6064322 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -10.6 | -10.4 | -10.3 |
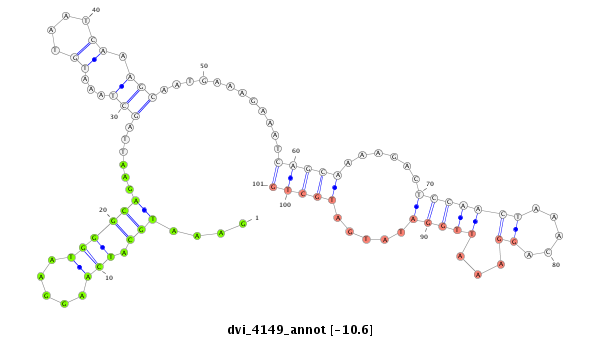 |
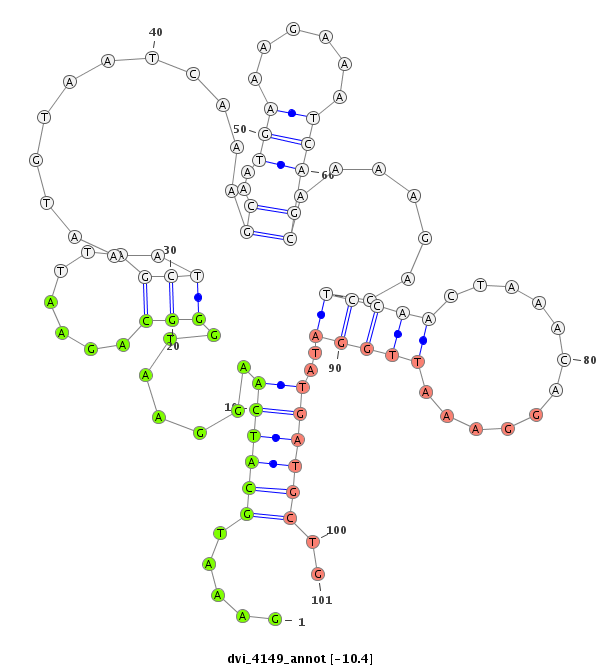 |
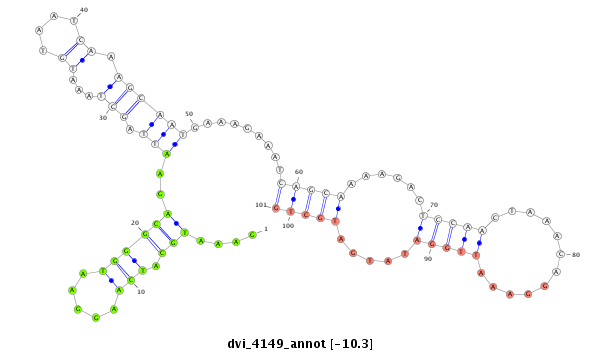 |
CDS [Dvir\GJ10237-cds]; exon [dvir_GLEANR_10175:1]; intron [Dvir\GJ10237-in]
| Name | Class | Family | Strand |
| AT_rich | Low_complexity | Low_complexity | + |
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- GCGGGGCGCCATGAAGAAGAAGCGCAAGAGATGTGTTGATGTGTATAACGGTATAACAGTTAAGCGCAGGAAATGCATCAAGGAATGGGCAGAATTAGCTAAATGTAATCAAAGCAATGAAAGAAATCAGCAAAAGACTCCAACTAAACAGGAAATTGGATATGATGCTGATGATGAATATTAATAAATCCTTTAATACTCTTTAAATTATTATAAAAAAATTGTTTTGTTGATGCATGAAATAAAAATGT *********************************************************************....(((.(((.....))))))......(((...((....)).)))............(((((......(((((............))))).....)))))********************************************************************************* |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060678 9x140_testes_total |
SRR060679 140x9_testes_total |
SRR060680 9xArg_testes_total |
SRR060655 9x160_testes_total |
SRR060670 9_testes_total |
V047 embryo |
SRR060666 160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................................................................GAAATGCATCAAGGAATGGGCAGAA............................................................................................................................................................. | 25 | 0 | 1 | 2.00 | 2 | 0 | 0 | 2 | 0 | 0 | 0 | 0 |
| .............................GATGTGTTGATGTGTATAACGGTA...................................................................................................................................................................................................... | 24 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................AGCGCAAGAGATGTGTTGATGTGTA.............................................................................................................................................................................................................. | 25 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................AACGGTATAACAGTTAAG........................................................................................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................GGAAATTGGATATGATGCTG................................................................................. | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .............................................................................................................................ATCAGCAAAAGACTCCAACT.......................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................AAATGCATCAAGGAATGGGCA................................................................................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .............AAGGGGGAGCGCAAGAGATGT......................................................................................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ............GAAGAAGAAGCGCAAGAG............................................................................................................................................................................................................................. | 18 | 0 | 20 | 0.10 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................ATGAAGCTGATGACGAAT........................................................................ | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
CGCCCCGCGGTACTTCTTCTTCGCGTTCTCTACACAACTACACATATTGCCATATTGTCAATTCGCGTCCTTTACGTAGTTCCTTACCCGTCTTAATCGATTTACATTAGTTTCGTTACTTTCTTTAGTCGTTTTCTGAGGTTGATTTGTCCTTTAACCTATACTACGACTACTACTTATAATTATTTAGGAAATTATGAGAAATTTAATAATATTTTTTTAACAAAACAACTACGTACTTTATTTTTACA
*********************************************************************************....(((.(((.....))))))......(((...((....)).)))............(((((......(((((............))))).....)))))********************************************************************* |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060678 9x140_testes_total |
V047 embryo |
M061 embryo |
SRR060687 9_0-2h_embryos_total |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|
| .............................................................................................................................................................CCTATACAACGACGACTAGTTATA...................................................................... | 24 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..............................................................................................AATTGATCTACATCAGTTTCGT....................................................................................................................................... | 22 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 |
| .................................................................CGTCCTTTCTGTATTTCCTTA..................................................................................................................................................................... | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 |
| ..............................................................................................AATTGATATACATAAGTTTCGT....................................................................................................................................... | 22 | 3 | 9 | 0.11 | 1 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................CAATTCGGGTCCTTAAAGTA............................................................................................................................................................................. | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
| ...............................................................................................................................................................TATACAACGACGACTAGTTATA...................................................................... | 22 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12855:6064122-6064372 - | dvi_4149 | GCGGGGCGCCATGAAGAAGAAGCGCAAGAGATGTGTTGATGTGTATAACGGTATAACAGTTAAGCGCAGGAAATGCATCAAGGAATGGGCAGAATTAGCTAAATGTAATCAAAGCAATGAAAGAAATCAGCAAAAGACTCCAACTAAACAGGAAATTGGATATGATGCTGATGATGAATATTAATAAAT---CCTTTAATACTCTTTAAATTATTATAAAAAAATTGTTTTGTTGATGCATGAAATAAAAATGT |
| droMoj3 | scaffold_6540:6383467-6383622 - | ACGAGGCCTTATGAAGAAGAAGCGCAAGCGACGTGTCGATTTGTACACTGAGCGATCAGTCAAGCGCAGAAAGTTCTTTAAGGAGTGGCCCGGAACACCCAAAGTGC----------TGAG--------TC------------CTAGGCAGGAATTGGGCTACGAGGCTGACGATGAAAATTAATA-------------------------------------------------------------------- | |
| droGri2 | scaffold_14906:8001563-8001783 + | GCT----------AAGAAGAATCGTAAGAGAAGCATAAGCGTATGCATCGAGATATCAGCCAAACGTAGAAAGTGTGCTACGGAGTTGGAAGAAATTCCGGAATTTA----------CTCA--------TTCAAGTAATCTAATTAGAATGGATATAGGCTATGAAGCTGACGATGAACAATAATGAAAGTAGTTTTAATATTCCTA-----ATTAAAATCTAGCATTTTTATTGAATATACAAATGGACATGT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
Generated: 05/19/2015 at 12:51 PM