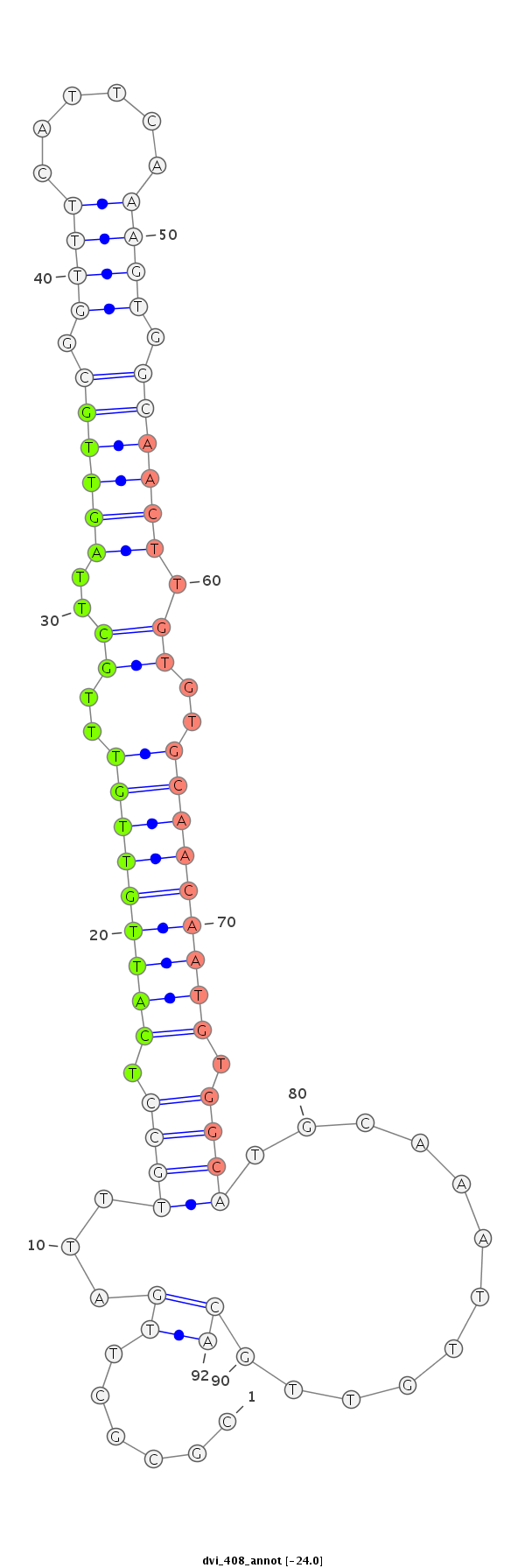


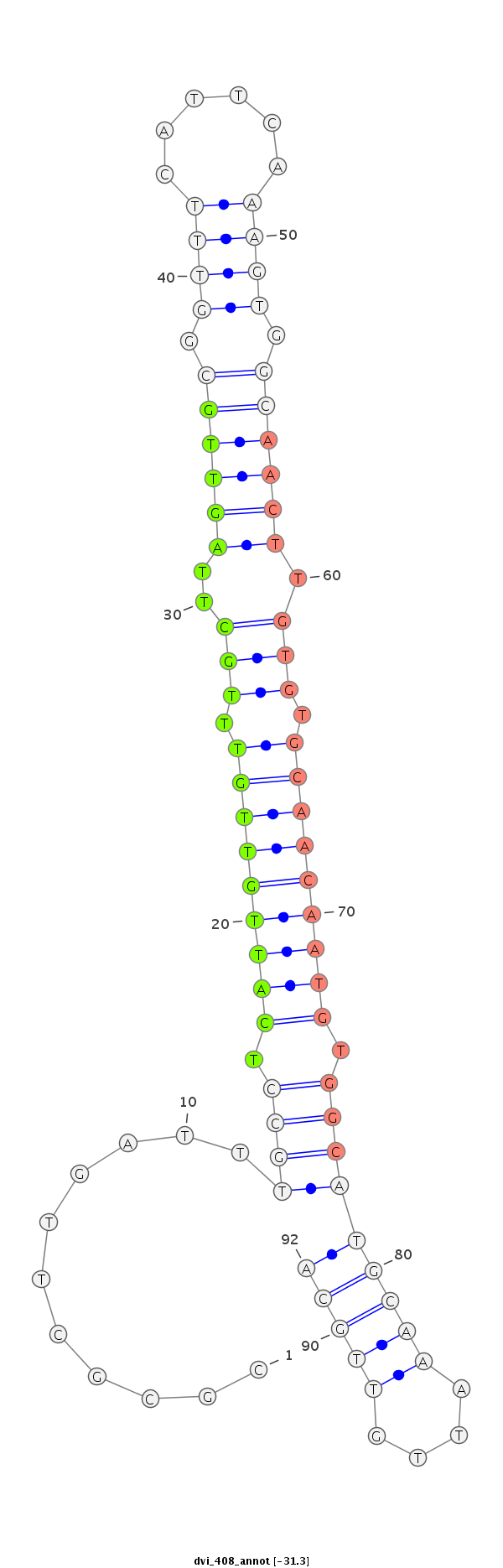
ID:dvi_408 |
Coordinate:scaffold_13049:8158233-8158295 - |
Confidence:candidate |
Class:Canonical miRNA |
Genomic Locale:intergenic |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -31.3 | -30.8 | -30.5 | -30.4 |
 |
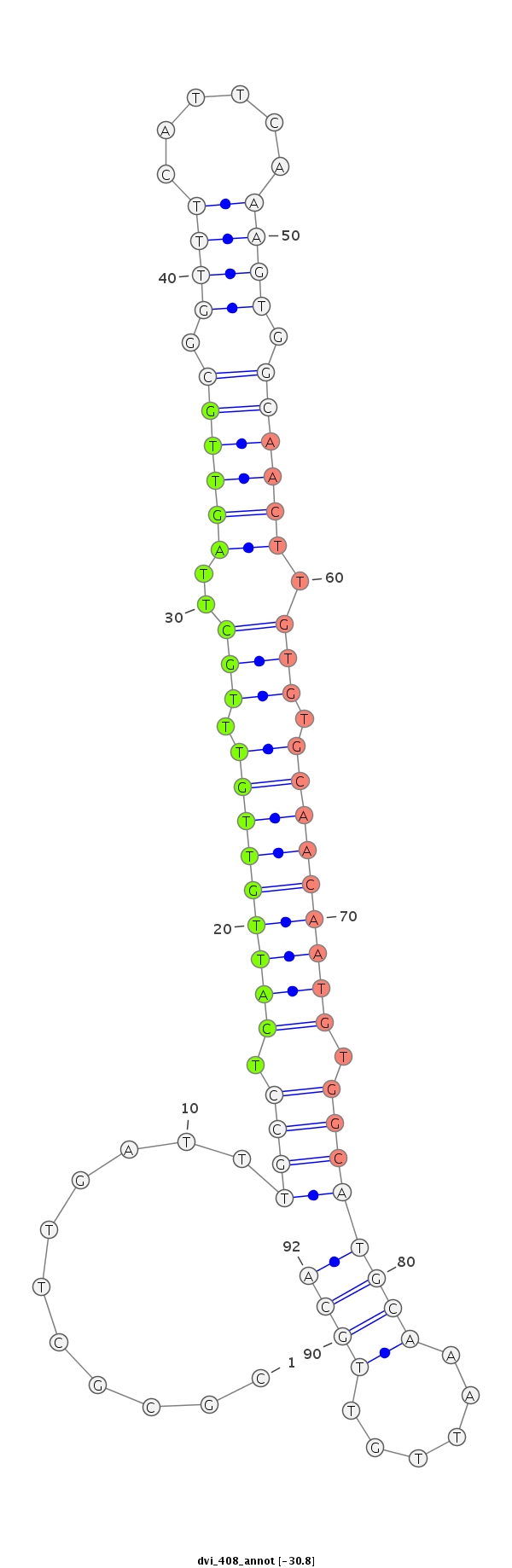 |
 |
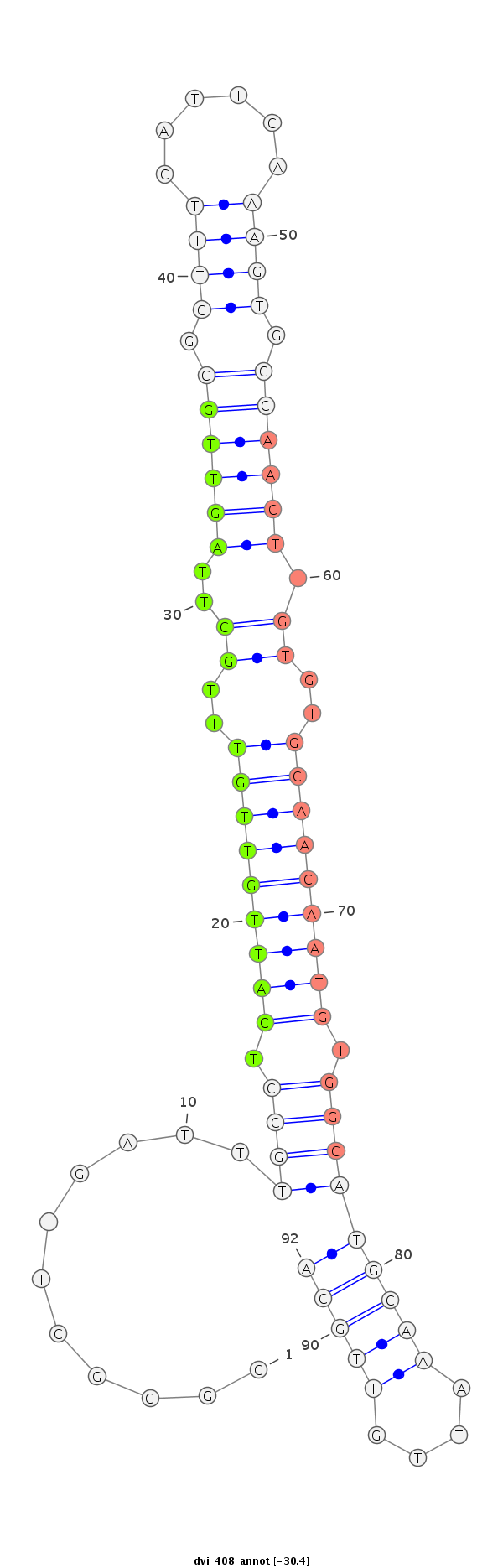 |
intergenic
No Repeatable elements found
|
TTCATGCCGTTTAGTGCACGCGCATTGCATTTGGACGCGCTTGATTTGCCTCATTGTTGTTTGCTTAGTTGCGGTTTCATTCAAAGTGGCAACTTGTGTGCAACAATGTGGCATGCAAATTGTTGCAAATTGTCAACGAGACAAGACCAGCCAGAACTCACGC
***********************************......((...((((.(((((((((..((..((((((.((((......)))).)))))).))..))))))))).))))............))************************************ |
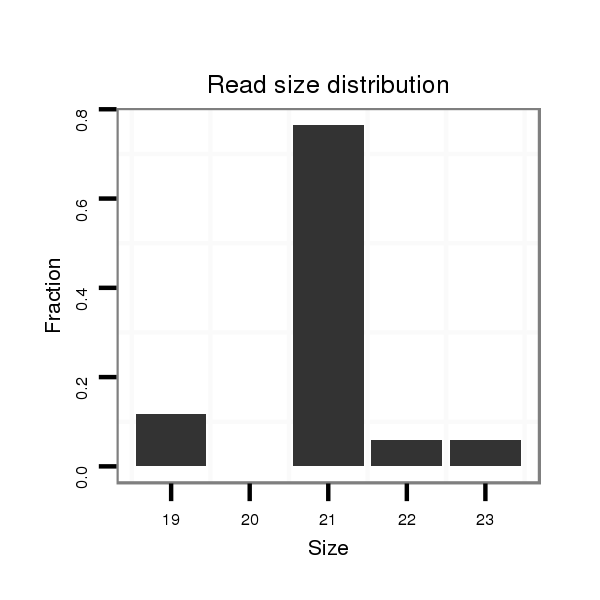
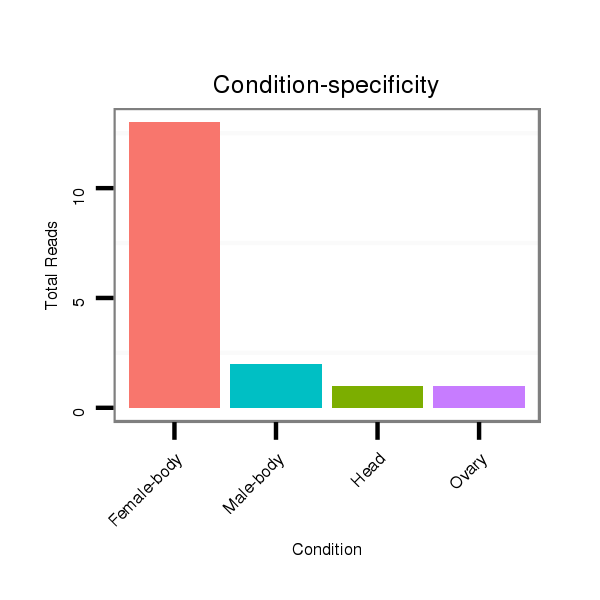
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060667 160_females_carcasses_total |
SRR060668 160x9_males_carcasses_total |
SRR060669 160x9_females_carcasses_total |
SRR060688 160_ovaries_total |
V053 head |
V116 male body |
SRR060686 Argx9_0-2h_embryos_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..................................................TCATTGTTGTTTGCTTAGTTG............................................................................................ | 21 | 0 | 1 | 13.00 | 13 | 6 | 3 | 1 | 1 | 0 | 1 | 1 | 0 | 0 |
| ..........................................................................................AACTTGTGTGCAACAATGTGGC................................................... | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................CATTGTTGTTTGCTTAGTT............................................................................................. | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................TCATTGTTGTTTGCTTAGT.............................................................................................. | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................TCATTGTTGTTTGCTTAGTTGCG.......................................................................................... | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................AGTTGCGGTATCACTCGAAG............................................................................. | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ....................................................................................GGTGGCAACTTGTGTGTATC........................................................... | 20 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
AAGTACGGCAAATCACGTGCGCGTAACGTAAACCTGCGCGAACTAAACGGAGTAACAACAAACGAATCAACGCCAAAGTAAGTTTCACCGTTGAACACACGTTGTTACACCGTACGTTTAACAACGTTTAACAGTTGCTCTGTTCTGGTCGGTCTTGAGTGCG
************************************......((...((((.(((((((((..((..((((((.((((......)))).)))))).))..))))))))).))))............))*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
GSM1528803 follicle cells |
SRR060663 160_0-2h_embryos_total |
SRR060668 160x9_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|
| ..........................................................................................................................................TCTGTGCAGGTCGGTCCTGAG.... | 21 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 |
| ................................................................................................................................................CTGGTCGGTCTCGAATGC. | 18 | 2 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 |
| ..............................................................CGAATCAAGGCAAAAGTTAG................................................................................. | 20 | 3 | 17 | 0.06 | 1 | 0 | 0 | 1 | 0 |
| .......................................GAACTAAACGCAGTAAGTA......................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Type | Alignment |
|---|---|---|---|---|
| droVir3 | scaffold_13049:8158183-8158345 - | dvi_408 | candidate | TTCATGCCGTTTAG---TGCACGC-GCATTGCATTTGGACG-----------CGCTTGATTTGC--CTCATTGTTGTTTGCTTAGTTGCGGTTTCATTCAAAGTGGCAACTT--------------GT-GTGCAACAATGTG--GCA--TGCAAATTGTTGCAAATTGTCAACGAGACAAGACCAGCCA------GAACTCACGC |
| droMoj3 | scaffold_6680:11820152-11820240 - | TGCATG----------------------------------------------CGCTTGATTTGCGGCTCATTGTG--CCGCT-------------------------------------------------GCAACAATGTG--GCAAGCGCAAATTGTGGCAAATTGTCAACGAGACAAGAATA-----------------TGC | ||
| droGri2 | scaffold_15110:7186156-7186344 - | TTCATGGATTTTAG---TGCACAGTTTACAGCAGTTGAGCGTGTTTTTGTGGCGCTTGATTTGC--GGCATTGTG--TTACTAATTTGCGGTTGCATACAATGTAGCAATGTAGCAATGCAGCAATGTAGTGCAACAATGTGCAGCA--TGCACATTGCG-----TTGTCAA-GCAAAACCAACGAACAGCCCAAGAAGCCA-TC | ||
| droWil2 | scf2_1100000004822:3412332-3412471 + | TTACTGCACTTTGACGTTGCATGC-GCAAAACATTTCGACG-----------T-----------GGCTTATTGTA--TTACTAATTTGCGGTCGGA------------------------------------CAACAATGTC--TGC--TGCAAATTGCCACAGCCCGGTGAAGGGAAGGGAACGGAGGGGGGATGGATAGAAGA | ||
| droEle1 | scf7180000490993:419643-419666 - | ------------------------------------------------------------------------------------------------------------------------------------------------------------------AAATTCGCAATGTAACAAGAGTAG------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
Generated: 10/20/2015 at 07:25 PM