
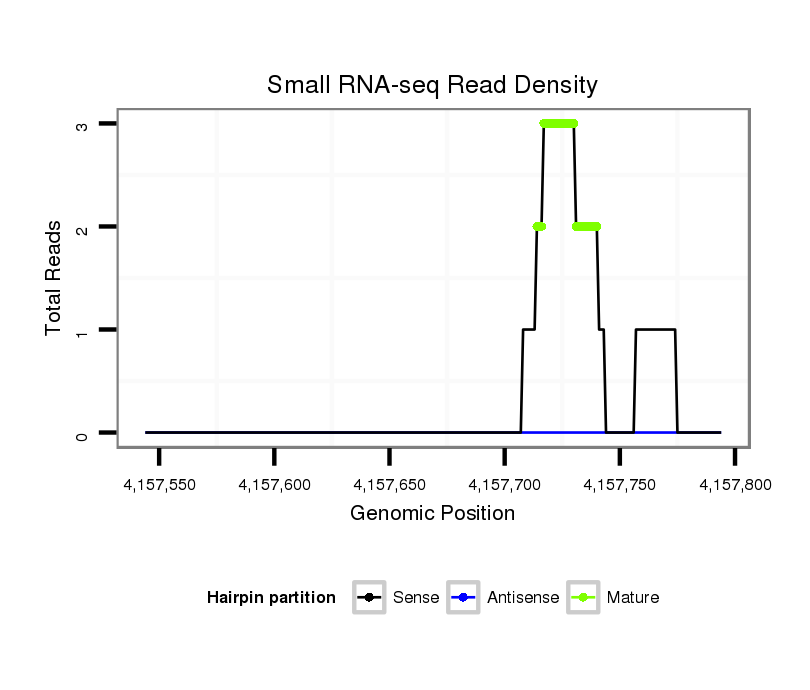
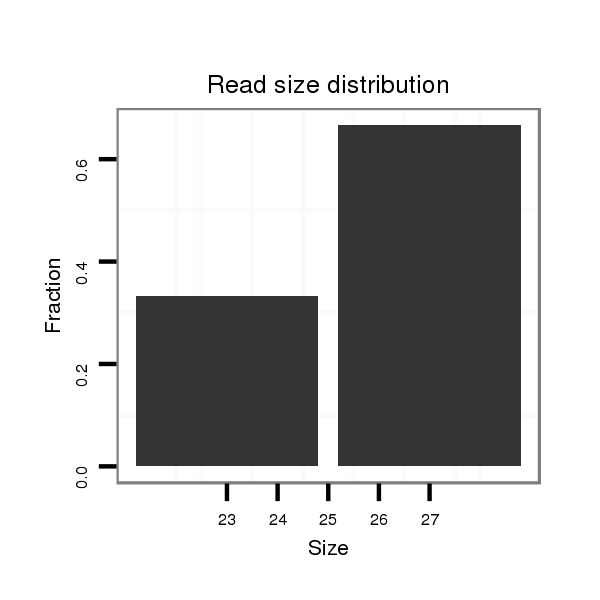
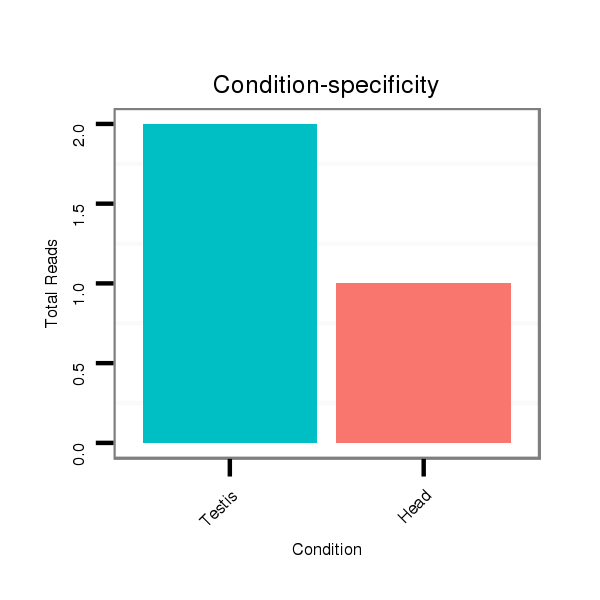
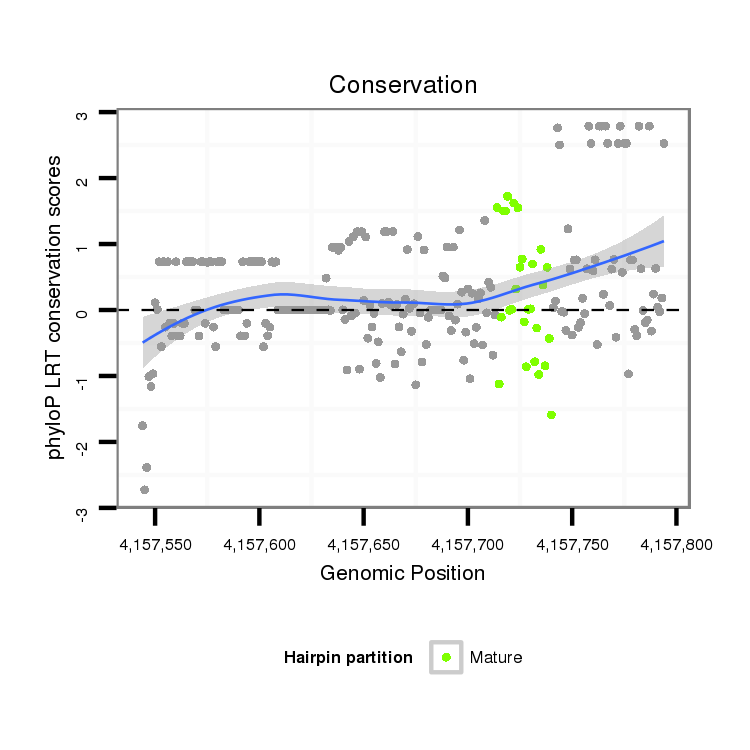
ID:dvi_3858 |
Coordinate:scaffold_12855:4157594-4157744 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -24.6 | -24.6 | -24.4 |
 |
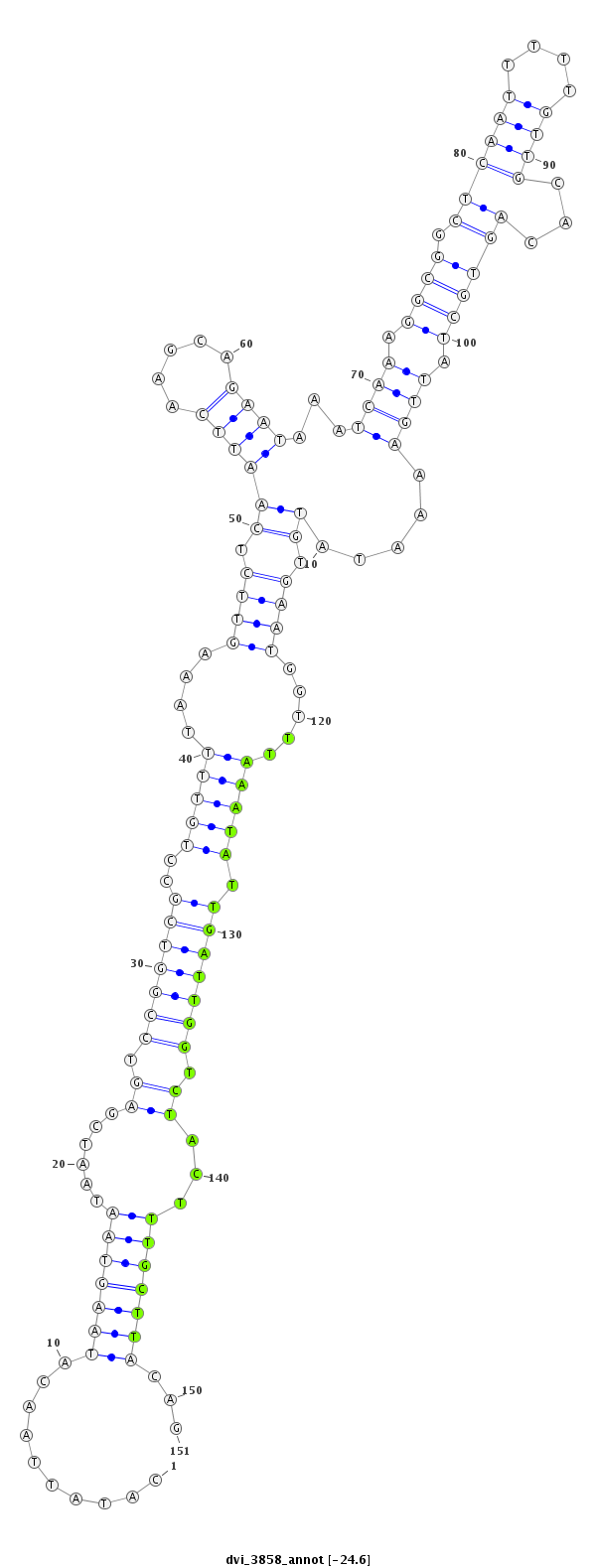 |
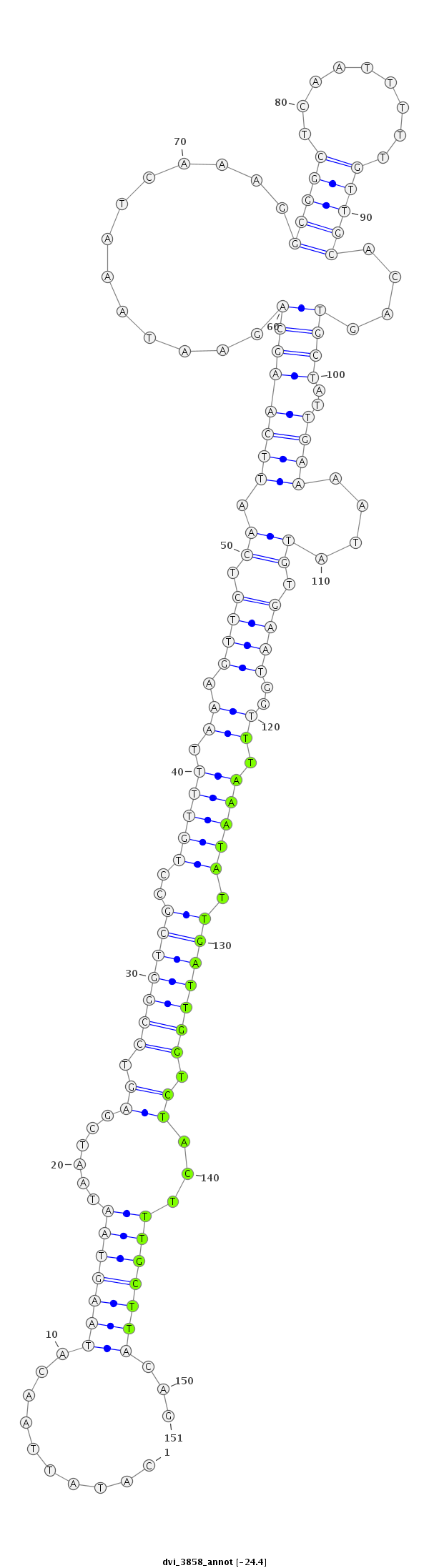 |
CDS [Dvir\GJ10812-cds]; exon [dvir_GLEANR_10712:2]; intron [Dvir\GJ10812-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## ATTATATGTGTAAGCGAACCGTAATAGCTACGTTTGTTAAATTATCAAACCATATTAACATAAGTAATAATCGAGTCCGGTCGCCTGTTTTAAAGTTCTCAATTCAAGCAGAATAAATCAAAGGCGGCTCAATTTTTGTTGCACAGTGCTATTGAAAATATGTGAATGGTTTAAATATTGATTGGTCTACTTTGCTTACAGCCCTCAAATGGAGACCTGTACACGCTGGAAGCCAATTTAGCTAACAAATC **************************************************..........(((((((......((.(((((((..(((((....((((.((.((((((((.............(((((.........)))))....))))..))))....)).)))).....))))).))))))).))...)))))))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060678 9x140_testes_total |
SRR060668 160x9_males_carcasses_total |
SRR060679 140x9_testes_total |
M028 head |
SRR060659 Argentina_testes_total |
SRR060681 Argx9_testes_total |
V053 head |
M047 female body |
SRR1106729 mixed whole adult body |
V116 male body |
SRR060677 Argx9_ovaries_total |
M027 male body |
SRR060666 160_males_carcasses_total |
SRR060676 9xArg_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................................................................................................................................................................................................................GACCTGTACACGCTGGAA.................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................ACATTGATTGGTCTACTTTGCTTACAG.................................................. | 27 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................TTAAATATTGATTGGTCTACTTTGCTT...................................................... | 27 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................AATGGTTTAAATATTGATTGGTC................................................................ | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................CCGGTCGCGTGTTTTGAAG............................................................................................................................................................ | 19 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................AATATTGATTGGTCTACTTTGCTTACA................................................... | 27 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................TCCCTCAAATGGAGACCTGT............................... | 20 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................AGCGTAATAGCTAAGCTTGT...................................................................................................................................................................................................................... | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................TCGAGTCCGGGCGCC...................................................................................................................................................................... | 15 | 1 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................GATGGCGACCTGTTCACGCT........................ | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................TGAATGGTTCAAATATGGTT..................................................................... | 20 | 3 | 20 | 0.10 | 2 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................CAGAAAAAATCATGGGCGGC........................................................................................................................... | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................GCACAGTGCTGTCGAAAAAA........................................................................................... | 20 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...............................................................................................................................................................AGGTGAATGGTTTAAAAA.......................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..................................................................................................................................................................TGAATGGTTCAAATATGGT...................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..................................................................................................................................................................................TCATTGGTCTACTTTCCCT...................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
|
TAATATACACATTCGCTTGGCATTATCGATGCAAACAATTTAATAGTTTGGTATAATTGTATTCATTATTAGCTCAGGCCAGCGGACAAAATTTCAAGAGTTAAGTTCGTCTTATTTAGTTTCCGCCGAGTTAAAAACAACGTGTCACGATAACTTTTATACACTTACCAAATTTATAACTAACCAGATGAAACGAATGTCGGGAGTTTACCTCTGGACATGTGCGACCTTCGGTTAAATCGATTGTTTAG
**************************************************..........(((((((......((.(((((((..(((((....((((.((.((((((((.............(((((.........)))))....))))..))))....)).)))).....))))).))))))).))...)))))))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
SRR060655 9x160_testes_total |
SRR060658 140_ovaries_total |
M027 male body |
M061 embryo |
|---|---|---|---|---|---|---|---|---|---|---|
| ............................................................ATTCATTATTGGCGCAGTCCA.......................................................................................................................................................................... | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ....CTACACAATCGCTTGGGATTA.................................................................................................................................................................................................................................. | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ............TCGCTTGGCACTATCGGT............................................................................................................................................................................................................................. | 18 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 |
| ...........................................................................................................................................................CGTATACAGTTACCAAATTTAT.......................................................................... | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................AAAAGAATGTTGGGAGTT........................................... | 18 | 2 | 11 | 0.36 | 4 | 0 | 0 | 4 | 0 | 0 |
| ....................................................................................................................................................................................................ATGTTGGGAGTTTACATC..................................... | 18 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 |
| ....................................................................................................................................................................................................ATGTTGGGAGTTGACATCT.................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12855:4157544-4157794 + | dvi_3858 | ATTATATGTGTAAGCGAAC---------CGTAATAGCTACGTTTGTTAAATTATCAAACCATATTAACATAAGTAATAATCGAGTCCGGTCGCCTGTTTTAAAGTTCTCAA---------TTCAA-GCAGAATAAATCAAAGGCGGCTCAATTTTTGTTGCACAGTGCTATTGAAAAT-----ATGTGAATGGTTTAAATATTGAT--TG------G-TCTACTTTGCTTACAGCCCTCAAATGGAGACCTGTACACGCTGGAAGCCAATTTAGCTAACAAATC--- |
| droMoj3 | scaffold_6540:8496988-8497130 + | CCCAT--------------------------------------------------------------------------------------------TTTAAAGTTTCCCA---------TTAAAAGTTGGGTCAATCGATAGGGGCGCAGTT-------------------------------AGTTAATGCCTGAAATATTGAT--TG------G-TTTACTTCACGCACAGCCCTCAAATGGAGACCTGTACACGCTGGAAGCGAACCTGGCCAACAGGGC--- | |
| droGri2 | scaffold_14906:6761563-6761682 + | GTTGCAC------------------------------------------------------------------------------------------------------------------------------------------------------------AGTGTTGTTGGAAAA-----TTGTAAATGCTTGAAATATTGAT--TGGTCTAGC-TTTGCATTGCTTACAGCCCTCAAATGGAGACCTGTACACCCTGGAAGCCAAATTGGTTAGCGATTC--- | |
| droWil2 | scf2_1100000004902:11401300-11401369 - | GTTTT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTT--TGGTTTGTATAGTCTCCCAATGGCGACCTCTATACCCTTGAAGCCAATTTAATGAACAAATC--- | |
| dp5 | 2:11502255-11502345 + | AGACAT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTAATTAGCT-------------TGATATTGA-AAAACGTAACTTGCAGTACCCCAACGGCGACCTCTACACGCTCGAGGCGAACCTGATGAACAAGTC--- | |
| droPer2 | scaffold_0:9016484-9016546 - | ACG-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAACTTGCAGTACCCCAACGGCGACCTCTACACGCTCGAGGCGAACCTGATGAACAAGTC--- | |
| droAna3 | scaffold_13340:9481112-9481198 + | TACTA---------------------------------------------------------------------------------------------------------------------------------------------------------------------CTGAAAGT-----------------------ATG-TG-TG------T-TTTTTTTTTCCAACAGTCACCCAATGGCGACCTATATACCCTTGAAGCCAACCTGGTCAGCAAGGC--- | |
| droBip1 | scf7180000396712:3104774-3104849 + | AAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAATGAT--TT--------CT--TGTTACCCACAGTCACCCAATGGCGACCTATACACCCTTGAAGCGAACTTGGTCAGCAAGGC--- | |
| droKik1 | scf7180000302699:380091-380144 - | A--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCCCCCAACAACGACCTGTACACCCTCGAGGCAAACCTGGTGAGCAAGTC--- | |
| droFic1 | scf7180000453800:1135202-1135254 - | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCTCCCAATGGCGACCTTTACACCCTGGAGGCCAATCTGGCGAACAAGGC--- | |
| droEle1 | scf7180000491008:1617198-1617416 + | AATATGTATCTTAAAAACAATTCTCCCCTATAAT--ATATGTTACTTA--------CAATATATTAAGGGTAGT------------------------CAAAAGTCCTCCATTGATTCTACTTAT-GCAGAGGTAATCAATGA-AA-TA----------------TTTTGTTGGCAATCTGGT------TTGATTCAAATGTTAACATTT-------GTTTTTTTT--TTGCAGATGCATCGCGAGAACGCCTATACCAAGGAGGCCGCCCTGAAGAATCTGGCCAG | |
| droRho1 | scf7180000769666:43275-43409 - | TCTAC--------------------------------------------------------------------------------------------------------------------TTAA-GCAAAGGAAATCAAAGGAAA-TTGTTCGTTT---------------GGCACTATGGT------TTGATTTGAATA----TGTTT-------ATTTACTTT--CCGTAGATGCATCGTGAGAACGCCTACACCAAGGAGGCCGCCCTGAAGAATCTGGCCAG | |
| droBia1 | scf7180000299102:1282907-1282958 + | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTCCCCGAACAACGACCTCTACACCCTCGAGGCGAACTTGGCGAGCCAGGC--- | |
| droTak1 | scf7180000415878:185865-186007 + | AAAGTTT----------------------------------------------------------------------------------------------------ACCA---------TTAAA-GTAA-CCACATTAAAGAAGACTTGCTTTTAT---------------TTTCCTAATCT--TCAAAGGATTTGAATG----TGTGC-------TTTTATTTT--CCGCAGATGCAACGCGAGAACGCCTACACCAAGGAGGCCGCCCTGAAGAATCTGGC--- | |
| droEug1 | scf7180000409802:1360611-1360702 + | AATAAATA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AATAATAAT--TTTTTTTGA-AATGCCTTACCGAAAGTCCCCGAACAGCGACCTTTACACTCTGGAGGCTAAACTGGCCAGCCGGGC--- | |
| dm3 | chr3R:8682278-8682325 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCCGAACTGTGACCTCTACACCCTGGAGGCCAACATGGCGAGTCAAGC--- | |
| droSim2 | 3r:12475999-12476053 + | T-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAAGAACCCGAACGGCGACCTCTACACCCTGGAGGCCAACCTGGCGAGTCAAGC--- | |
| droSec2 | scaffold_0:13307099-13307156 + | GC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTAAAGAACCCGAACGGCGACCTCTACACCCTGGAGGCCAACCTGGCGAGTCAAGC--- | |
| droYak3 | 3R:12976443-12976498 - | T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAAAGTCCCCGAACAGCGACCTCTACACCCTGGAGGCCAACCTGGCGAGTCAAGC--- | |
| droEre2 | scaffold_4770:12966426-12966481 + | T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAAAGTCCCCTAATGGCGACCTCTACACCCTGGAGGCCAACCTGGCGAGTCAAGC--- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 11:30 PM