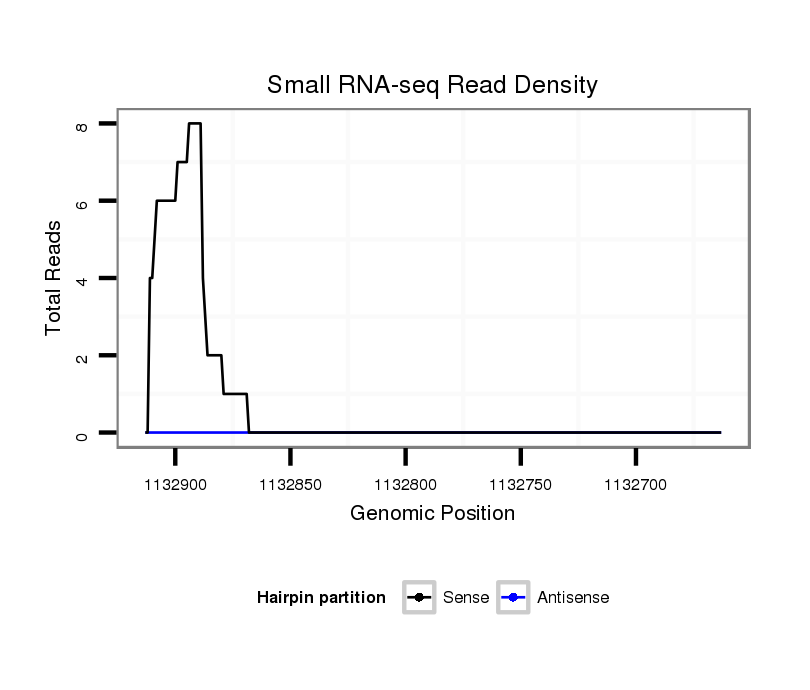
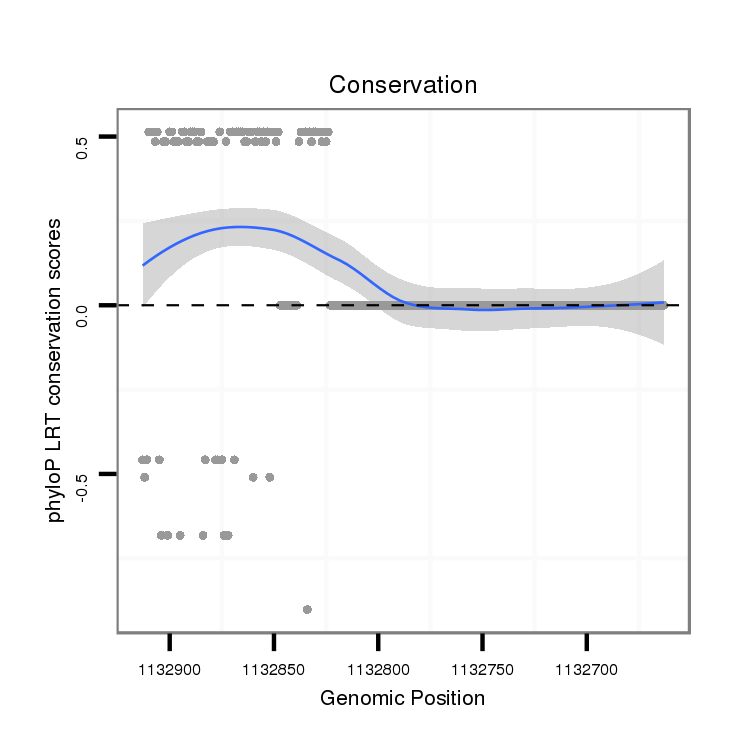
ID:dvi_2931 |
Coordinate:scaffold_12823:1132713-1132863 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ17761-cds]; exon [dvir_GLEANR_233:1]; intron [Dvir\GJ17761-in]
No Repeatable elements found
| -------------#####################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- ATCAATCTCAGCCATGGGTTTCGATAGCAACGCGGACAAGCCAATAAAACGTATGTAGACTTTTGTTGATTTCCAGTTTCTGATTTGTGAATGTAAACTTAATAGGTGAGGTATTTAAGAGACATTGCCCTTTAAAAAAAATTATTTTGAATGAAAACTATGCAAGTGAATTATTCAAGCAACATTTGCATCGATTAGAAAGCCGATAAATTGAAATTTATATATGTTTATTTTAAACAAGAGAATTTTCT **************************************************.......(((....))).........((((((((((((((((((....((((..(((((.((.........)).)))))..))))..........(((((((((..(((.....)))...)))))))))..)))))))))..)))))))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060664 9_males_carcasses_total |
SRR060668 160x9_males_carcasses_total |
M061 embryo |
SRR060671 9x160_males_carcasses_total |
SRR060678 9x140_testes_total |
SRR060666 160_males_carcasses_total |
SRR060685 9xArg_0-2h_embryos_total |
M027 male body |
SRR060682 9x140_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060672 9x160_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..CAATCTCAGCCATGGGTTTCGAT.................................................................................................................................................................................................................................. | 23 | 0 | 1 | 3.00 | 3 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............TGGGTTTCGATAGCAACGCG......................................................................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................TTCGATAGCAACGCGGACAAGCCAAT.............................................................................................................................................................................................................. | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................CATTTGCATTGATTAGAAAG................................................. | 20 | 1 | 2 | 1.00 | 2 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................TAAGAGACATTCCCCTTTAAAA.................................................................................................................. | 22 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....TCTCAGCCATGGGTTTCGATA................................................................................................................................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....ATCTCAGCCATGGGTTTCGAT.................................................................................................................................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................AGGTAGACTTTTGTTGATTTCC................................................................................................................................................................................. | 22 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..CAATCTCAGCCATGGGTTTCGATAG................................................................................................................................................................................................................................ | 25 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................ACATCGCCCGTCAAAAAAAA.............................................................................................................. | 20 | 3 | 16 | 0.25 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 1 | 1 | 0 |
| ............................AACGCGGACAATCCATTAC............................................................................................................................................................................................................ | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................................................................................GAATGCAAACCATGAAAGTG................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TAGTTAGAGTCGGTACCCAAAGCTATCGTTGCGCCTGTTCGGTTATTTTGCATACATCTGAAAACAACTAAAGGTCAAAGACTAAACACTTACATTTGAATTATCCACTCCATAAATTCTCTGTAACGGGAAATTTTTTTTAATAAAACTTACTTTTGATACGTTCACTTAATAAGTTCGTTGTAAACGTAGCTAATCTTTCGGCTATTTAACTTTAAATATATACAAATAAAATTTGTTCTCTTAAAAGA
**************************************************.......(((....))).........((((((((((((((((((....((((..(((((.((.........)).)))))..))))..........(((((((((..(((.....)))...)))))))))..)))))))))..)))))))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR1106727 larvae |
V053 head |
SRR060670 9_testes_total |
SRR060684 140x9_0-2h_embryos_total |
M027 male body |
SRR060687 9_0-2h_embryos_total |
SRR1106729 mixed whole adult body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................................................................................................TTTCGGCTAGTTAACTTTAAA................................ | 21 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................TCACTTAAAAAGGTCGTGGTAA................................................................. | 22 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................TCACTTAATAAGTTCACGGTAA................................................................. | 22 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................CTCTTCGGTTCTGTTGCATA..................................................................................................................................................................................................... | 20 | 3 | 10 | 0.20 | 2 | 0 | 0 | 0 | 0 | 1 | 1 | 0 |
| ...............................................................................................................................................TGATACTTACTTTTGATAC......................................................................................... | 19 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................CGCTATCGTTGCTCCTGATC................................................................................................................................................................................................................... | 20 | 3 | 13 | 0.08 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................AAAGGTCAAAGGCTA....................................................................................................................................................................... | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12823:1132663-1132913 - | dvi_2931 | ATCAATCTCAGCCATGGGTTTCGATAGCAACGCGGACAAGCCAATAAAACGTATGT-----AGACTTTTGTTGATTTCCAGTTTCTGATT-TGTGAATGTAAACTTAATAGGTGAGGTATTTAAGAGACATTGCCCTTTAAAAAAAATTATTTTGAATGAAAACTATGCAAGTGAATTATTCAAGCAACATTTGCATCGATTAGAAAGCCGATAAATTGAAATTTATATATGTTTATTTTAAACAAGAGAATTTTCT |
| droGri2 | scaffold_15245:6082820-6082906 + | GATAATCTTCGCAATGGGGTTCGATAGCACTGCGGGTAGTCAAACAAAACGTAAGTAATCTAGACTATTGT---------GTTTGTGATTATGTGA----------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
Generated: 05/16/2015 at 11:23 PM