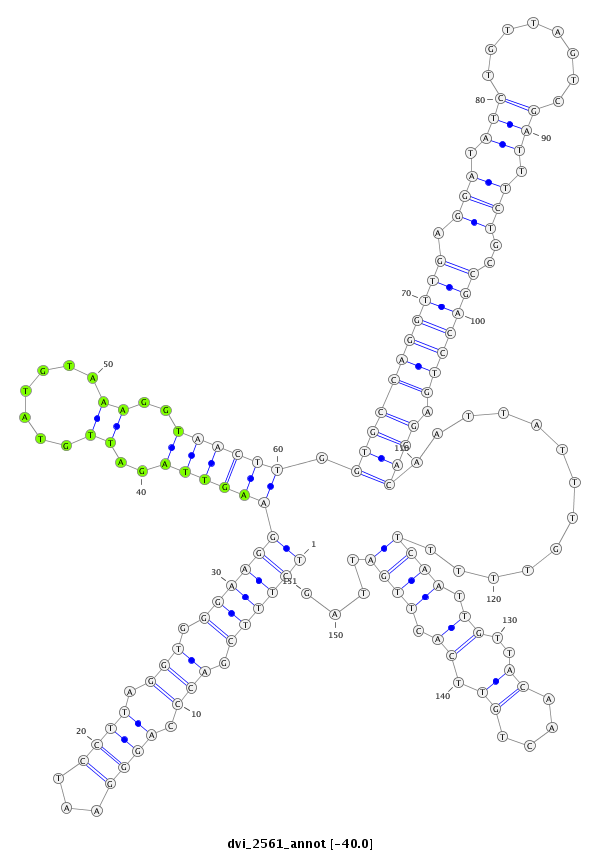
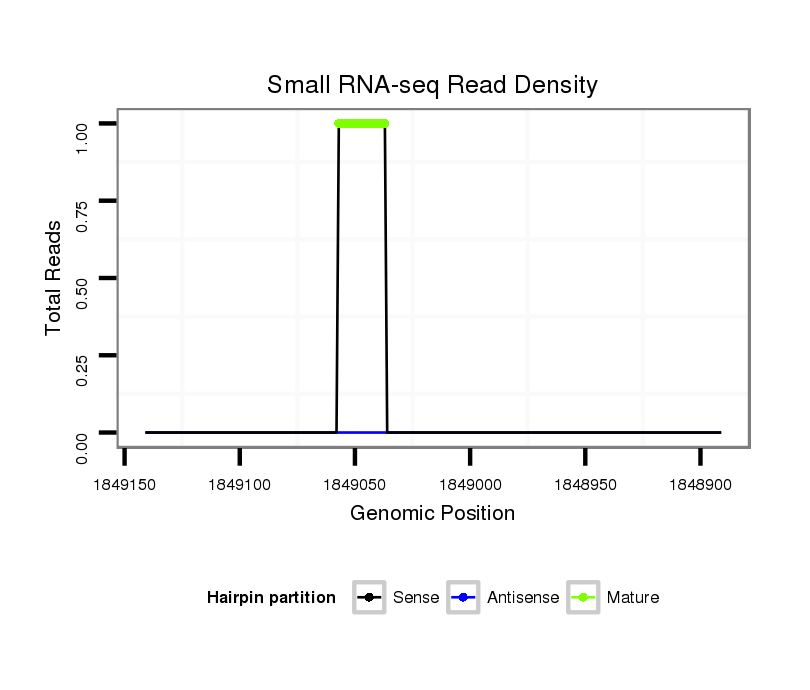
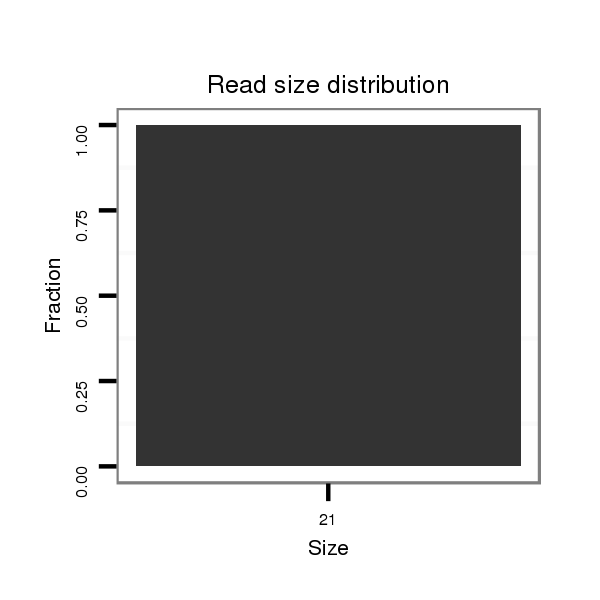
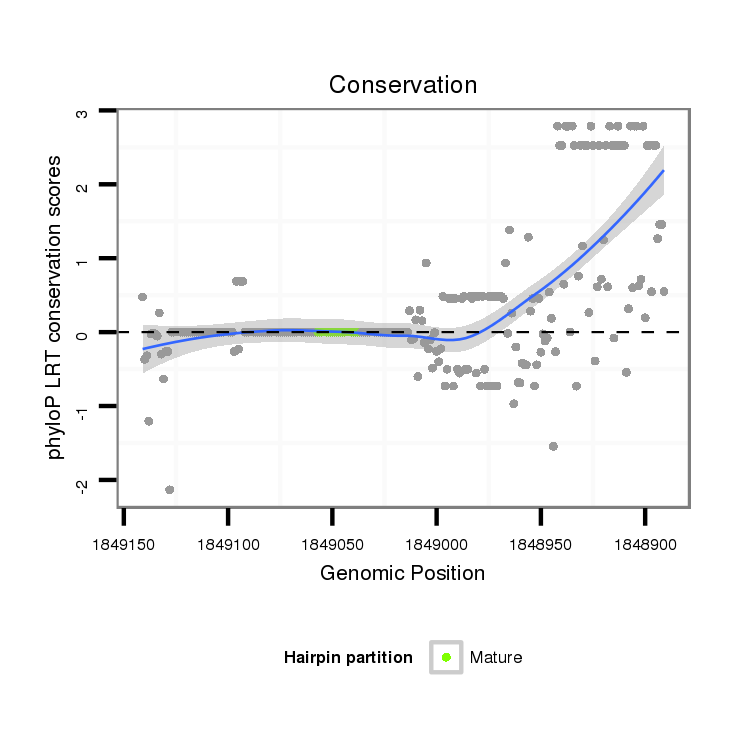
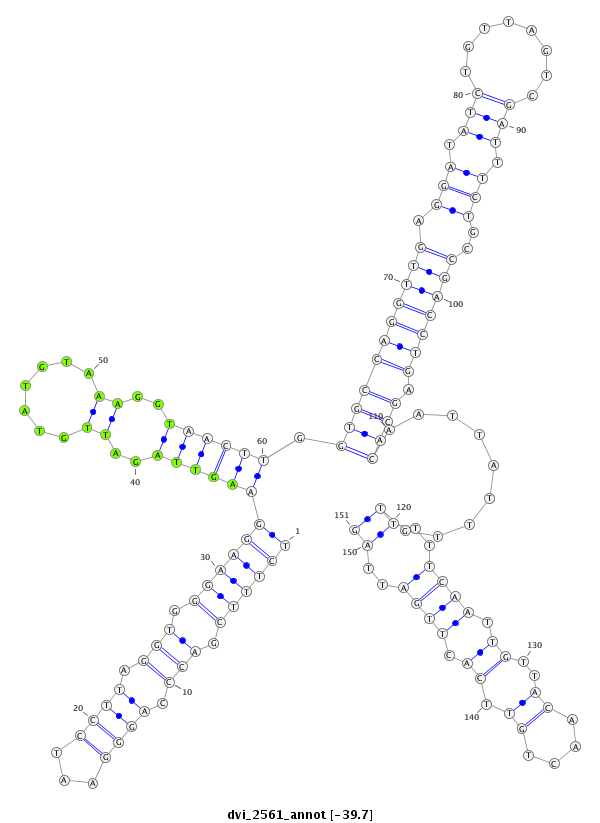
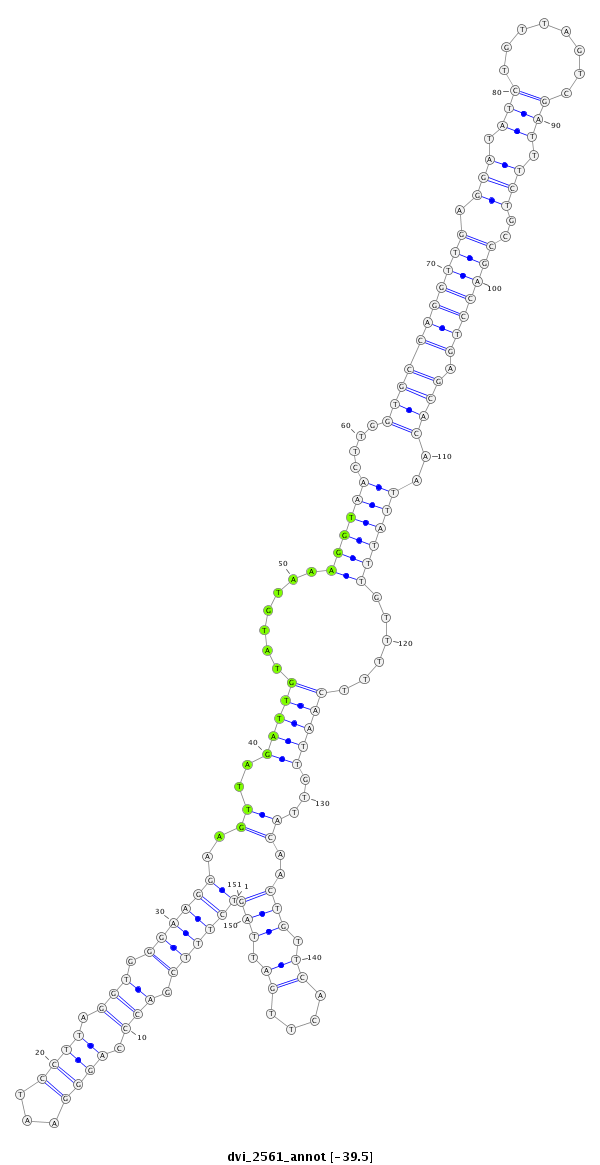
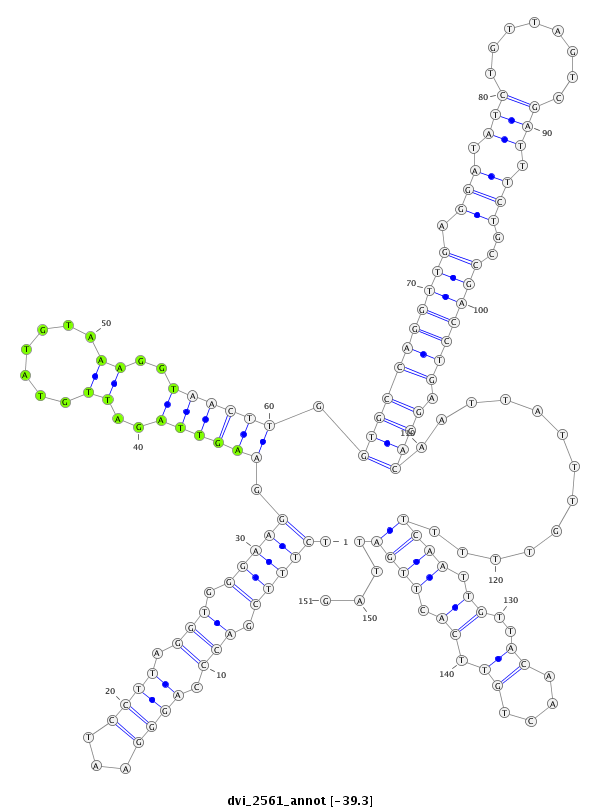
ID:dvi_2561 |
Coordinate:scaffold_12822:1848941-1849091 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -39.7 | -39.5 | -39.3 |
 |
 |
 |
exon [dvir_GLEANR_14203:4]; CDS [Dvir\GJ14246-cds]; intron [Dvir\GJ14246-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TTCCTGATGAGAAGGGCACCTTGAAAGTTGCTGGGGGTTACAATTATATTTCTTTCGACCCAGGGAATCCTTAGGTGGGAAGGAAGTTAGATTGTATGTAAAGGTAACTTGGTGCCAGGTTGAGGATATCTGTTAGTCGATTTCTGCCGACCTGAGCACAATTATTTGTTTTTCAATTGTTACAACTGTTCACTTGATTAGGCATCACCACGGGCTCGTCCAGCTCGGTGCCGTTGTTTGTGCGTCCCATG **************************************************((((((.(((.((((...)))).))).))))))((((((..((.......))..)))))).(((((((((((.(((.(((........))).)))..))))))).)))).............((((.((..((....)).)).))))....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060667 160_females_carcasses_total |
V053 head |
SRR060663 160_0-2h_embryos_total |
GSM1528803 follicle cells |
M061 embryo |
SRR060682 9x140_0-2h_embryos_total |
SRR060665 9_females_carcasses_total |
SRR060679 140x9_testes_total |
SRR060683 160_testes_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060672 9x160_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................AGTTAGATTGTATGTAAAGGT.................................................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................GGGAATACGTAGGTGGCAAG......................................................................................................................................................................... | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........AGAAGGGCTCGTTGAAAGT............................................................................................................................................................................................................................... | 19 | 2 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................CGATGTGAGCACAATGATTT.................................................................................... | 20 | 3 | 18 | 0.22 | 4 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 1 | 0 |
| ....................................................................................................................................TCAGTCGATTTCTGCCTCCC................................................................................................... | 20 | 3 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................................GTCCAGCGCGATGCCGT................. | 17 | 2 | 12 | 0.17 | 2 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................GTAATACTTAGGTGGCAAG......................................................................................................................................................................... | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................GGAATACGTAGGTGGCAAG......................................................................................................................................................................... | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................GATGAGATCTGTTAGTAGA............................................................................................................... | 19 | 3 | 17 | 0.06 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................................GTTGTTTGAGCGCCCCGTG | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ....................................................................................................................................................GACCTGAGCTCAAGTATTC.................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
AAGGACTACTCTTCCCGTGGAACTTTCAACGACCCCCAATGTTAATATAAAGAAAGCTGGGTCCCTTAGGAATCCACCCTTCCTTCAATCTAACATACATTTCCATTGAACCACGGTCCAACTCCTATAGACAATCAGCTAAAGACGGCTGGACTCGTGTTAATAAACAAAAAGTTAACAATGTTGACAAGTGAACTAATCCGTAGTGGTGCCCGAGCAGGTCGAGCCACGGCAACAAACACGCAGGGTAC
**************************************************((((((.(((.((((...)))).))).))))))((((((..((.......))..)))))).(((((((((((.(((.(((........))).)))..))))))).)))).............((((.((..((....)).)).))))....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
SRR060683 160_testes_total |
V047 embryo |
SRR060682 9x140_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060658 140_ovaries_total |
SRR060667 160_females_carcasses_total |
SRR060662 9x160_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..........................................................................................................................................................TCGTGTTAAGAAACAAAA............................................................................... | 18 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................CTTCCATAGAACCACAGTCC.................................................................................................................................... | 20 | 3 | 3 | 0.67 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................GCAGGGTGCCTTTGGAATC................................................................................................................................................................................. | 19 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................................GGTCGAGCCACGGCGAAA.............. | 18 | 2 | 6 | 0.33 | 2 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................AGTGGTCCCCAAGCAGGT............................. | 18 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................GAGTCCTCCCTTGCTTCAA................................................................................................................................................................... | 19 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................AAAGACGGCTTGACTCAT............................................................................................. | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ......................................................................................................CCATTGAACGACGGTCAAGC................................................................................................................................. | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................TGGTCCGTTAGGAATCTAC.............................................................................................................................................................................. | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ................................................................................................................................AGACGATCAGCTAAACGCG........................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................AAAGCTGGGATCCTTGGG..................................................................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...........................................................................................................................................................................................................................GGTCGAGCCACGGCGAAAT............. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12822:1848891-1849141 - | dvi_2561 | TTCC--TGAT---GAGAAGGGCACCTTGAAAGTTGCTGGGGGTTACAATTATATTTCTTTCGACCCAGGGAATCCTTAGGTGGGAAGGAAGTTAGATTGTATGTAAAGGTAACTTGGTGCCAGGTTGAGGATATCTGTTAGTCGATTTCTGCCGACCTGAGCACAATTA----TTTGTTTTTCAATTGTTACAACTGTTC---ACTTGATTAGGCATCACCACGGGCTCGTCCAGCTCGGTGCCGTTGTTTGTGCGTCCCATG |
| droMoj3 | scaffold_6540:11294519-11294650 + | TA-----------------------------------------------------------------------------------------------------------------------------------TCTGCTAATCGTTCACTTTCGCCTAGAATACATTGACAAACGTTTTGTTCAATTTCTGCAACTGGTCCATTCATTAACAGGCATTACCTCGGGCTCGGCCAGCTCCGTGCCGTTGTTTGTGCGTCCCATG | |
| droGri2 | scaffold_14624:2853536-2853623 - | TTCGTT-------TAA---------------------------------------------------------------------------------------------------------------------CACAAA----------------------------------------------TGTTCGATTCA--A---TTCATTAACAGGCATCACGACGGGCTCGGCCAGTTCGGTGCCGTTGTTTGTGCGTCCCATG | |
| droWil2 | scf2_1100000004943:11280934-11281021 - | TTGTCT-------GTA---------------------------------------------------------------------------------------------------------------------TCTAAT----------------------------------------------TTGTGTTCCCT--G---CTGATGGACAGGCATTACATCCGGCTCGGCTAGCTCGGTGCCATTGTTTATTCGTCCTGCC | |
| dp5 | 2:15795999-15796082 - | TTTC--AGTT---GTCTTT------------------------------AACAT-------------------------------------------------------------------------------TTCAACA-----------------------------------------------------------------CCGACCAGGTATTACGTCCGGCTCCGCCAGCTCCGTGCCTTTGTTTGTCCGCCCCATG | |
| droPer2 | scaffold_0:4617550-4617633 + | TTTC--AGTT---GTCTTC------------------------------AACAT-------------------------------------------------------------------------------TTCAACA-----------------------------------------------------------------CCGACCAGGTATTACGTCCGGCTCCGCCAGCTCCGTGCCTTTGTTTGTCCGCCCCATG | |
| droAna3 | scaffold_13340:18125622-18125680 - | CT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGGTTAGGCATCACATCCGGTTCCGCTGGCTCGGTGCCATTGTTCATCCGCCCCATG | |
| droBip1 | scf7180000396708:5248458-5248516 + | CT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGGCTAGGCATCACATCCGGTTCCGCTGGCTCGGTGCCATTGTTCATCCGCCCCATG | |
| droKik1 | scf7180000302255:1998-2090 + | CA-----------------------------------------------------------------------------------------------------------------------------------GCTATTTGTGTTCCTG----------------------------------ATTTACCTTAACT--T---CCGTGGTTTAGGCATCACATCCGGCTCAGCTGGCTCGGTGCCTTTGTTCATTCGCCCCATG | |
| droFic1 | scf7180000453910:376433-376498 + | TG-----------------------------------------------------------------------------------------------------------------------------------TTCATT------------------------------------------------------------------TGGGACAGGCATCACATCCGGCTCGGCTGGCTCAGTGCCACTTTTCATCCGCCCCATG | |
| droEle1 | scf7180000491278:744005-744070 + | TA-----------------------------------------------------------------------------------------------------------------------------------TTCGCT------------------------------------------------------------------TGGGATAGGCATCACATCCGGCTCGTCTGGCTCGGTGCCGTTGTTCATCCGCCCCATG | |
| droRho1 | scf7180000770346:643-720 - | TTTA--AATATTTGCA---------------------------------------------------------------------------------------------------------------------TTCATT------------------------------------------------------------------TGGGTCAGGTATCACATCCGGCTCGGCTGGCTCTGTGCCGTTGTTCATCCGCCCCATG | |
| droBia1 | scf7180000302155:2698757-2698808 - | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGGCATAACTTCCGGCTCTGCTGGCTCGGTGCCGCTCTTCATCCGCCCCATG | |
| droTak1 | scf7180000414778:25115-25170 + | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGCCAGGCATCACTTCCGGTTCGGCTGGCTCGGTGCCCCTGTTCATCCGCCCCATG | |
| droEug1 | scf7180000409766:1020029-1020095 + | TA-----------------------------------------------------------------------------------------------------------------------------------TTCATTT-----------------------------------------------------------------TGGGCCAGGCATCACATCCGGTTCAGCTGGCTCGGTGCCGTTGTTCATCCGCCCCATG | |
| dm3 | chr3R:2031374-2031429 + | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGCTAGGCATCACTTCCGGCTCGGCTGGGTCGGTGCCGTTGTTTATCCGCCCCATT | |
| droSim2 | 3r:1930933-1930988 + | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGCTAGGCATCACATCCGGCTCGGCTGGGTCGGTGCCGTTGTTCATCCGCCCCATG | |
| droSec2 | scaffold_6:2117436-2117491 + | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGCTAGGCATCACATCCGGCTCGGCTGGGTCGGTGCCGTTGTTCATCCGCCCCATG | |
| droYak3 | 3R:17957200-17957255 + | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGCCAGGCATCACATCCGGCTCGGCTGGGTCGGTGCCGTTGTTCATCCGTCCCATG | |
| droEre2 | scaffold_4770:2273369-2273424 + | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGCCAGGCATCACTTCCGGCTCGGCTGGGTCGGTGCCGCTGTTCATCCGCCCCATG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/19/2015 at 11:51 AM