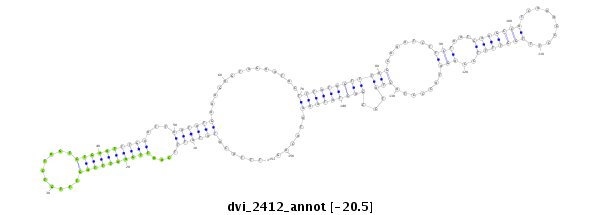
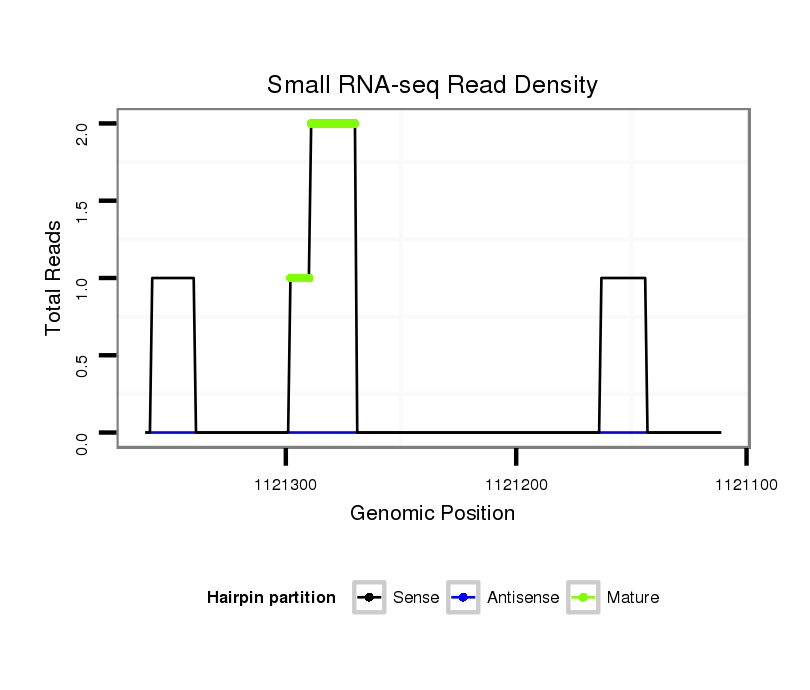
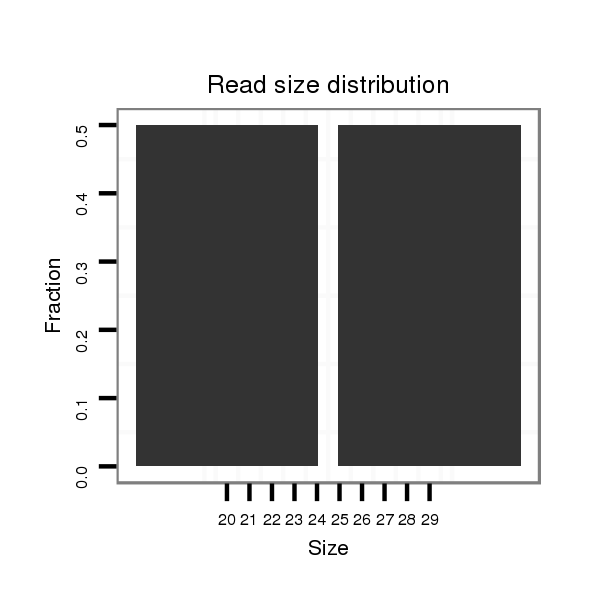
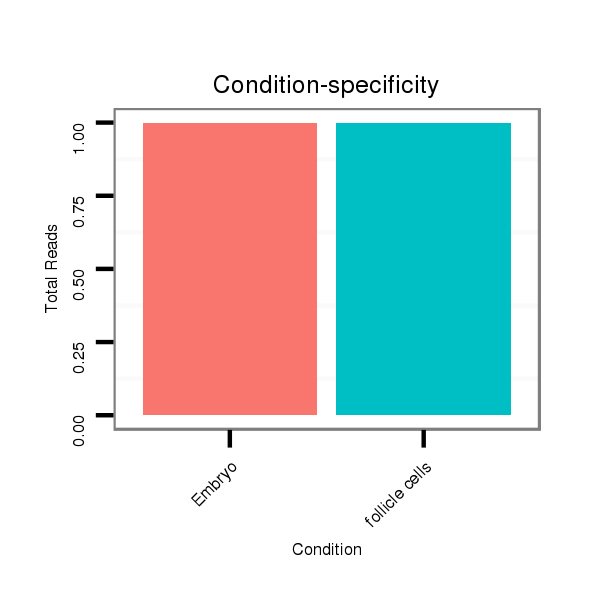
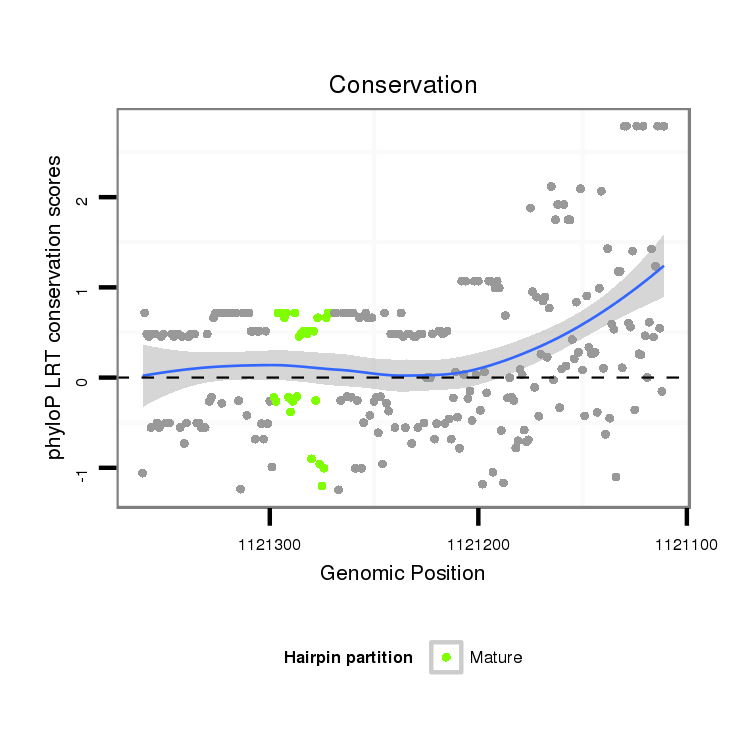
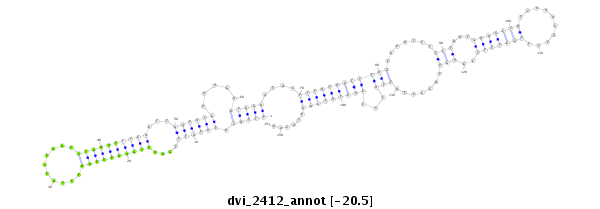
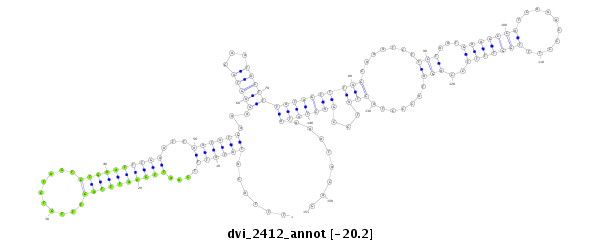
ID:dvi_2412 |
Coordinate:scaffold_12822:1121161-1121311 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -20.5 | -20.5 | -20.2 |
 |
 |
 |
exon [dvir_GLEANR_14258:2]; CDS [Dvir\GJ14305-cds]; intron [Dvir\GJ14305-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TAAGATTCTGAGCTTGTAAGACGAATGCTATATGGAAATAATAAAAAGAATTTACATATATTTCAATTGAATTTAGTTTGTCTAGTCTGAATTTGAATTAATATGAAAGAGTAGAATACTTTATAGTTTAGCAAATCTTCTAATGAAGCCATCAAACCCTTTGGTTTTCCAGATACCCTACTATCCGACTATAAAATACAGTTGCCACCGCTGAAACGAAAATCGGGTAAAAAGGAGAGCTTCGATCATTT **************************************************......((((((....((((((((((..........))))))))))...))))))..............((((((((((((.......(((...(((((((..........)))))))..))).......)))...)))))))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
M061 embryo |
GSM1528803 follicle cells |
SRR060677 Argx9_ovaries_total |
SRR060664 9_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................CAATTGAATTTAGTTTGTCTAGTCTGAAT............................................................................................................................................................... | 29 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................CAGTTGCCACCGCTGAAACG................................. | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................CAAGATACCCTACTATCCGA............................................................... | 20 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...GATTCTGAGCTTGTAAGAC..................................................................................................................................................................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................TTAGTTTGTCTAGTCTGAAT............................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 |
| ......................................TTATAAAAAGAATTTACAT.................................................................................................................................................................................................. | 19 | 1 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 |
| ..........................................................................................................................................................................................................................AGAATCGGGTAAAAAGGA............... | 18 | 1 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 1 |
|
ATTCTAAGACTCGAACATTCTGCTTACGATATACCTTTATTATTTTTCTTAAATGTATATAAAGTTAACTTAAATCAAACAGATCAGACTTAAACTTAATTATACTTTCTCATCTTATGAAATATCAAATCGTTTAGAAGATTACTTCGGTAGTTTGGGAAACCAAAAGGTCTATGGGATGATAGGCTGATATTTTATGTCAACGGTGGCGACTTTGCTTTTAGCCCATTTTTCCTCTCGAAGCTAGTAAA
**************************************************......((((((....((((((((((..........))))))))))...))))))..............((((((((((((.......(((...(((((((..........)))))))..))).......)))...)))))))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060671 9x160_males_carcasses_total |
SRR060666 160_males_carcasses_total |
SRR060687 9_0-2h_embryos_total |
SRR060669 160x9_females_carcasses_total |
SRR060668 160x9_males_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
M027 male body |
SRR060662 9x160_0-2h_embryos_total |
SRR060664 9_males_carcasses_total |
SRR060665 9_females_carcasses_total |
SRR060681 Argx9_testes_total |
V116 male body |
M047 female body |
SRR060674 9x140_ovaries_total |
SRR060679 140x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .............CACATTTTGCTTACGATAT........................................................................................................................................................................................................................... | 19 | 2 | 3 | 4.00 | 12 | 1 | 3 | 0 | 2 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..............ACATTTTGCTTACGATAT........................................................................................................................................................................................................................... | 18 | 1 | 1 | 2.00 | 2 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............ACATTTTGCTTACGATATG.......................................................................................................................................................................................................................... | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................TAGTTTGGGAAACCGGATGGT................................................................................ | 21 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...............CATTTTGCTTACGATATGG......................................................................................................................................................................................................................... | 19 | 3 | 20 | 0.15 | 3 | 0 | 1 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................ATGGTTTAGAAGACTACT......................................................................................................... | 18 | 2 | 7 | 0.14 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................TGGTTTAGAAGACTACTTT....................................................................................................... | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .......................................................................................................................................................................................................................................TTCCTCTGGGAACTAGTAA. | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..................................................AAATGTATAAAAAGTTAAC...................................................................................................................................................................................... | 19 | 1 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................AAAAGATTACTTCGGTTG.................................................................................................. | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12822:1121111-1121361 - | dvi_2412 | TAAGATT---CTGAGCT--TGTAAGA-CGAATGCTATATGGAAA-TAATAAAAAG--AATTTACATATATTTCAATTGAATTTAGTTTGTCTAGTCTGAATTTGAATTAAT-AT----GAAAGAGTAGAATACTTTATAGTTTA---GCAAATCTTCTAATGAAGCCATCAAAC---CCTTTGGTTTTCCAGATACCCTACTATCCGACTATAAAATACAGTTGCCACC---------GCTGAAAC---------GAAAAT-------CGGGT------------------AAAAAGGAGAGCTTCGATCATTT |
| droMoj3 | scaffold_6540:33762263-33762493 - | TAAGTTTATCTAGAATCTATGTATGCTTGAATATATAAAAGAAATCAATAAAAGC--AAAT---------AATTATTGAATATAGT------GGATGGAATTTTGATTAAT-CCTAAGGCCAGTGAACAGTGTTTAATAGATTCAACGCTAAC--------------TTTAAA-----CTTTGCATTTTCAGAC---TCGTTAACGGACTATAAGATCCAGCTGCCATT---------GCCAAAAC---------GTAAACTGGG-------------CC---------GA---AAGGAGAGCTTCGATCATTT | |
| droGri2 | scaffold_15074:2705621-2705822 + | TA------------------------------------TGGAAA-CAATAAAAAATTAAATTAAATTGAATGCAATTGATGTTG--TTGTGTAGCGAGAATTTCAATCATTTTT----GTAAGTGTTGGTTGT-----A-----------------CGTATGTAAA-AAAAAATATATTTCTTTTCTTGCAGCA---CTGCTTAGCGATTTTAAGATCCATTTGCCGCT---------GCTGACGCG---------AAAGTCA----------------G---------CGAAAAAGGAAAGTTTCGATCATCT | |
| droWil2 | scf2_1100000004943:16188311-16188364 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GC--------------------------CACAACTTAAAC---------GGC------G-------------TCCAGGATCGGAGAAAAATCCAAGTTTCGATCATAT | |
| dp5 | 2:5690222-5690342 + | C---------------------------------------------------------------------------------------------------------------------------------------------------------------------TACCACAT---CATTTTGTCTTGCAGGGAT---CGAAGATGAATTCAAAGTGCAGTTGCCGTTGCTGCCCCTGCTCAAGC---------GGAAGTCAGG----CTCTGGCTCAG---------TGAAGAAGCAAAGCTTCGACCACCT | |
| droPer2 | scaffold_19:1419000-1419120 + | C---------------------------------------------------------------------------------------------------------------------------------------------------------------------TACCACAT---CATTTTGTCTTGCAGGGAT---CGAAGATGAATTCAAAGTGCAGTTGCCGTTGCTGCCCCTGCTCAAGC---------GGAAGTCAGGC----TCCGGCTCAG---------TGAAGAAGCAAAGCTTCGACCACCT | |
| droAna3 | scaffold_13340:23489901-23489943 - | G--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAAGCCGGGT-----------------------------------------------------GGAGCCTCCT---------TG---AAGCAAAACTATTCGAACCT | |
| droBip1 | scf7180000396395:494977-494999 - | A------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GG---AAGCACAACTACCAGCACAT | |
| droKik1 | scf7180000302461:318819-318875 + | G----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CT---------GCCAGTGGCGAAGA---AAAAGC-------CGGGCGGCTCCC---------GG---AAGCAGAACTATGACCACTT | |
| droFic1 | scf7180000454096:477586-477665 + | C-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GACTTCAAGGTGCAGCTTCCGCT---------CCTGGTGGCTAAGAAAAA-AAGG-------CGGGCAGCTCTC---------GC---AAGCAGAACTACGACCACCT | |
| droEle1 | scf7180000491104:1735467-1735486 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAGCAGAACTTTGACCACCT | |
| droRho1 | scf7180000767410:12557-12634 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTTCAAGGTGCAGCTTCCGCT---------CCTGGTGGCCAGAAAAAAAAAGG-------CGGGTAACTTCC---------GC---AAACAGAACTTTGACCACCT | |
| droBia1 | scf7180000302155:575266-575285 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAGCAGAGCTTCGACCACCT | |
| droTak1 | scf7180000415417:171568-171650 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GACTTCAAGGTGCATCTGCCGCT---------CCTGGTGGCCAGGAAGAGGAAA----ACGACGGGCGGCTGCC---------GC---AAGCAGAACTTTGACCACCT | |
| droEug1 | scf7180000409760:1219298-1219380 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GACTATAAGGTGCATCTGCCGCT---------CCAGGTGTCCAGGAAAAAGAAG----GCGTCGGGCGTCTCTG---------GC---AAGCAAAACTTTGACCACCT | |
| dm3 | chr3R:17191800-17191819 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAGCAGAGTTTTGACCACCT | |
| droSim2 | 3r:4120093-4120112 - | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAGAAGAGTTTTGACCACCT | |
| droSec2 | scaffold_6:4299016-4299035 - | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAGAAGAGTTTTGACCACCT | |
| droYak3 | 3R:5997525-5997607 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GACTACAAGGTGCACCTGCCGCT---------CCTGGTGGCAAGGAAAAAGAAG----ACTCCCGGTGGCTCAC---------GC---AAGCAGAACTTTGACCACCT | |
| droEre2 | scaffold_4770:4479796-4479878 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GACTACAAGGTGCACCTGCCGCT---------CCTGGTGGCAAGGAAAAAGAAG----ACTCCTGGTGGCTCCC---------GC---AAGCAGAACTTTGACCATCT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 04:29 AM