
ID:dvi_2411 |
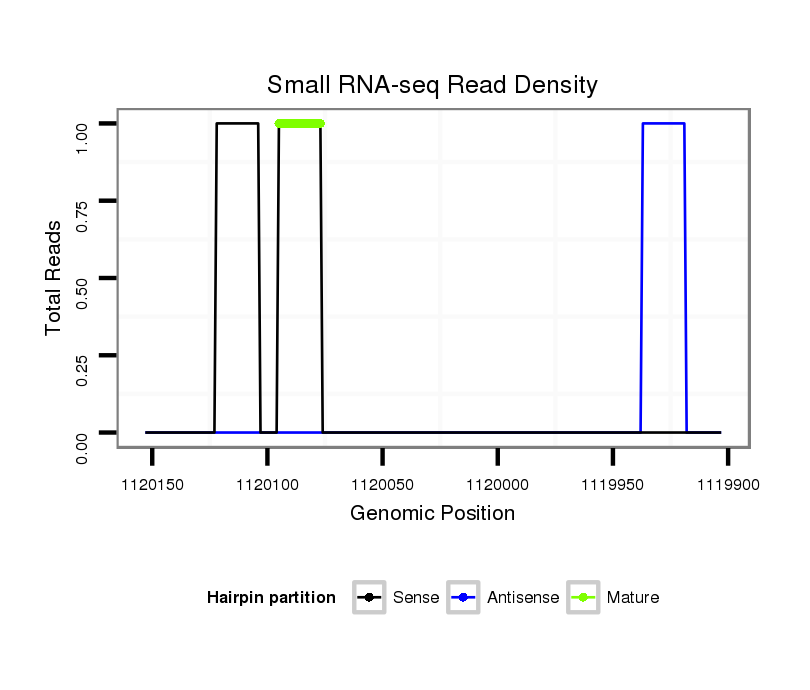
Coordinate:scaffold_12822:1119953-1120103 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
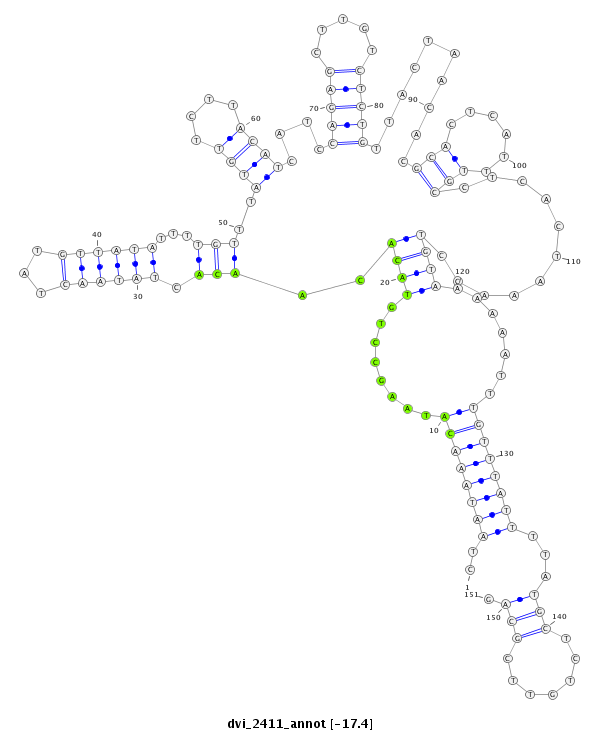
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -17.4 | -17.4 | -17.3 |
 |
 |
 |
CDS [Dvir\GJ14306-cds]; exon [dvir_GLEANR_14259:3]; intron [Dvir\GJ14306-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CTGATTCGCTGCATTGGCAACACTGGCGCCGTGCTGCAGCTAACTAGTCCCTAATAAACATAAGCCTGTACACAACACTATAACTATGTTATATTTTGTTTATGTTCTTACATCATCCAGAGCTTGTCTCTGTTACTAACACGCACTCATTTGCCTCACTAAACCTGTAAAAAATTTGTTTATTTTATGCTCTGTTCGCAGCATCCGCCTCCTCGCCCCGATACCGACGCATCACTTACAGAGAACGCGCC **************************************************..((((((((........((((.((((.((((((...))))))...)))).((((....))))....(((((.....)))))..........(((......)))...........)))).......))))))))...(((.......))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR1106729 mixed whole adult body |
SRR060677 Argx9_ovaries_total |
M047 female body |
SRR060658 140_ovaries_total |
SRR060675 140x9_ovaries_total |
SRR060683 160_testes_total |
SRR1106727 larvae |
M061 embryo |
M027 male body |
SRR1106728 larvae |
SRR060676 9xArg_ovaries_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ............................CAGTGCTGCAGCTAAC............................................................................................................................................................................................................... | 16 | 1 | 5 | 3.40 | 17 | 10 | 0 | 0 | 0 | 0 | 0 | 5 | 0 | 0 | 2 | 0 | 0 |
| .................................GTGCAGCTAACTGGTCCC........................................................................................................................................................................................................ | 18 | 2 | 4 | 1.25 | 5 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 2 | 0 | 0 | 0 |
| ..........................................................CATAAGCCTGTACACAACA.............................................................................................................................................................................. | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................TGCTGCAGCTAACTACTCCCT....................................................................................................................................................................................................... | 21 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................TGCTGCAGCTAACTAGTCC......................................................................................................................................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................TAACACGCACTCGTCTGCCTC.............................................................................................. | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................TGGCGCCGTGCTGCAGCTAAA............................................................................................................................................................................................................... | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................TTATGCTCTGTACACAGCAGC.............................................. | 21 | 3 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................CTAACACGCACTCATTTTTTT............................................................................................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .............TTGGGAACACTGCCGCGGT........................................................................................................................................................................................................................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .................................GGGCAGCTAACTGGTCCC........................................................................................................................................................................................................ | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................GTGCAGCTAACTGGTCCTT....................................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GACTAAGCGACGTAACCGTTGTGACCGCGGCACGACGTCGATTGATCAGGGATTATTTGTATTCGGACATGTGTTGTGATATTGATACAATATAAAACAAATACAAGAATGTAGTAGGTCTCGAACAGAGACAATGATTGTGCGTGAGTAAACGGAGTGATTTGGACATTTTTTAAACAAATAAAATACGAGACAAGCGTCGTAGGCGGAGGAGCGGGGCTATGGCTGCGTAGTGAATGTCTCTTGCGCGG
**************************************************..((((((((........((((.((((.((((((...))))))...)))).((((....))))....(((((.....)))))..........(((......)))...........)))).......))))))))...(((.......))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V116 male body |
SRR060687 9_0-2h_embryos_total |
SRR060679 140x9_testes_total |
SRR060655 9x160_testes_total |
SRR060658 140_ovaries_total |
SRR060680 9xArg_testes_total |
SRR060685 9xArg_0-2h_embryos_total |
M047 female body |
SRR060667 160_females_carcasses_total |
V053 head |
V047 embryo |
M028 head |
SRR060681 Argx9_testes_total |
SRR060657 140_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .............................................................................................................TGTAGTAGGTGGCGAACA............................................................................................................................ | 18 | 2 | 1 | 10.00 | 10 | 6 | 2 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................TGTAGAAGGTTTCGAACAGAG......................................................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................GGGCTATGGCTGCGTAGTG................ | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................GGGCTATGGTTGAGTAGTGGA.............. | 21 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................AGTGGGTTTCGAACAGAG......................................................................................................................... | 18 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................AGCGGGGATATGGGTTCGT.................... | 19 | 3 | 11 | 0.27 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................AGCGGGGATATGGGTTCGTA................... | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................TGTAGTAGGTGGCGAACAC........................................................................................................................... | 19 | 3 | 16 | 0.25 | 4 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...............CCGTTGTGAGTGCGACACGAC....................................................................................................................................................................................................................... | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................................................GTGTAGTAGGTGGCGAACA............................................................................................................................ | 19 | 3 | 18 | 0.11 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................CTTAGCCGGAGGAGCGGAG................................ | 19 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................AGGAGCGGGGATATGGGTT....................... | 19 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................CGGGGCTCTTGTTGCGTAG.................. | 19 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .....................................................................................................ACCAAGAAGGTAGTAGGT.................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
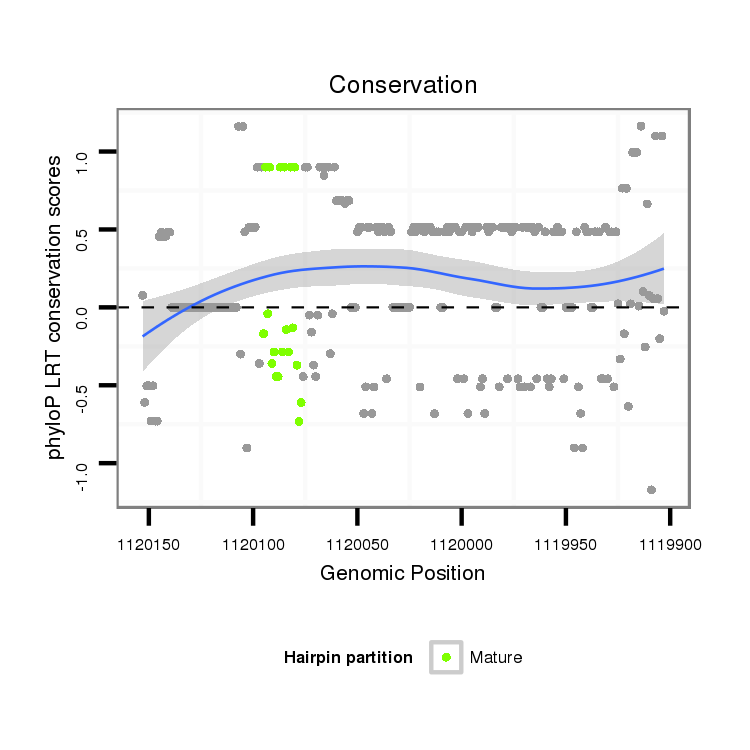
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12822:1119903-1120153 - | dvi_2411 | CTGATTCGCTGCATTGGCAACACTGGCGCCGTGCTGCAGCTAACTAGTCCCTAATA-----------------AACATAAGCCTGTACACAACACTATA--------ACTATGTTATATTTTGTTTATGTTCTTAC-----ATCATCCAGAGCTTGTCTCTGTTACTAACACGCACTCATTTGCCTCACTAAAC----CTGTAAAAAATTTGTTTATTTTATGCTCTGTTCGCAGCATCCGCCTCCTCGCCCCGATACCGACGCATCACTTACAGAGAACGCGCC |
| droMoj3 | scaffold_6540:33761464-33761490 - | CGAGGCATCTGCAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAAGGGGCGCGCC | |
| droGri2 | scaffold_15074:2706469-2706674 + | A---------------------------------------------GCCCGTAATACGCATTAGCTGTTCATATATATATAAATATATACATGCATATGTCTTGTAGTATATGTTGTA----------GTTAATAAGTCGTTTCATCTAG---------CTGTAACTAACCCG--CTCATTCGCTTAACTAATTCATCATGTA--TAATCTGTTCTTATTTTGTT--GCATGCAGCG----GCAAGTCG--CCGGCATCGTCGT---GCTTATAGAGGCCGCACC | |
| droWil2 | scf2_1100000004830:806399-806445 - | AT-----------------------------------------------------A-----------------TACACATACCTATCTAAAAGACTAAG--------ACAATGTTACATTTTGTT---------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | 2:5691138-5691141 + | C---------------------------------------------GCC-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_19:1419917-1419920 + | C---------------------------------------------GCC-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000302155:576229-576229 + | C-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409760:1218336-1218360 - | C---------------------------------------------GTC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATACCTTTTATAG--GGAGAACG | |
| droEre2 | scaffold_4770:4478883-4478883 - | C-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 04:29 AM