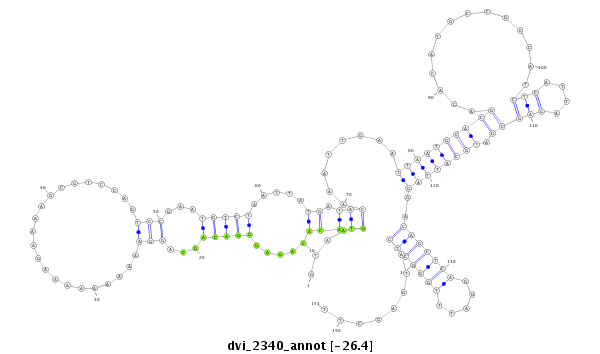
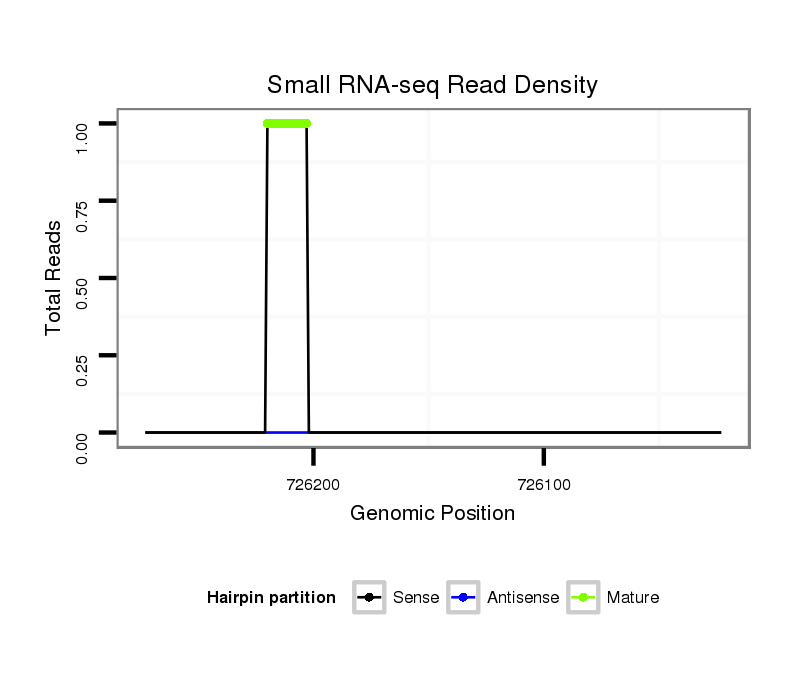
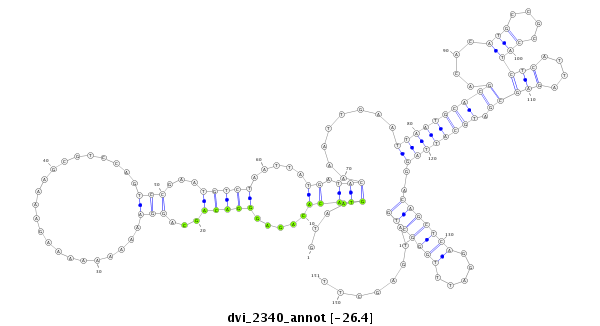
ID:dvi_2340 |
Coordinate:scaffold_12822:726073-726223 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
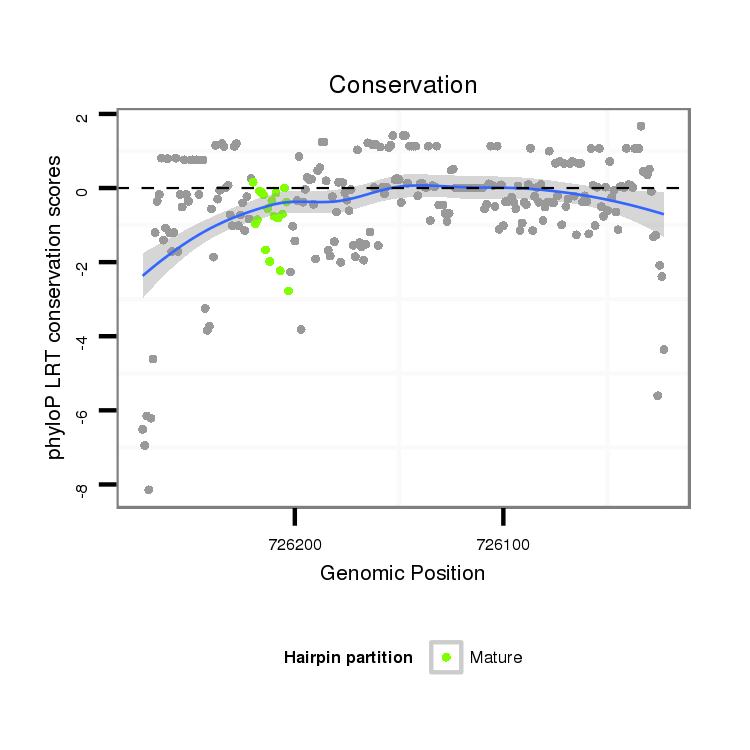
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
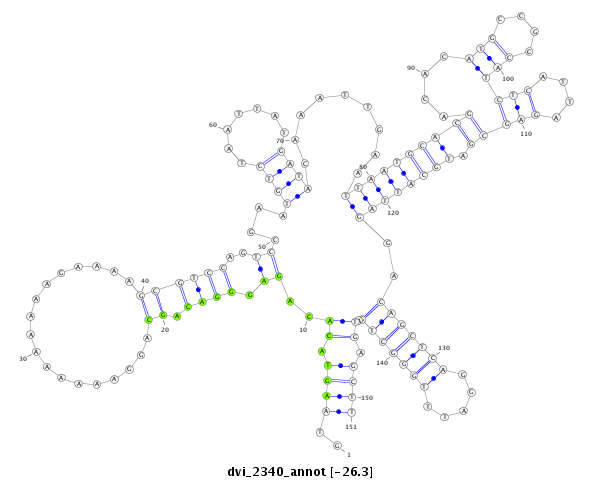
| -26.4 | -26.3 | -26.3 |
 |
 |
 |
CDS [Dvir\GJ14331-cds]; exon [dvir_GLEANR_14281:1]; intron [Dvir\GJ14331-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CCTCTTTCCTCCGAGTACGGACCCAGTCTGGCCTACAATCTGAGCAGGGTGTAAGTACACAGAGGGACAGCAGGAAAAAAAAAAGAAAAGCGTCCAGTCCGAATGTCTAATTATGATACAAATTGAATTAATGCACGACACATGCCGCCATCTCATTAGAGCGATGCATTAGGACAGCTCAGGATTTGGGCTGATGAGCTTACTCCTCCGCCTCCTGATTTCTGTACTGGCCATGCATATCTAATGACGCA **************************************************....(((((.....(((((...(((......................)))...))))).....)).)))........((((((((((..............(((....))))).))))))))..(((((((.....)))))))........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060655 9x160_testes_total |
SRR060681 Argx9_testes_total |
V053 head |
SRR1106730 embryo_16-30h |
SRR060679 140x9_testes_total |
SRR060682 9x140_0-2h_embryos_total |
M047 female body |
SRR1106727 larvae |
SRR1106728 larvae |
V047 embryo |
GSM1528803 follicle cells |
SRR060664 9_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................GGTGTCAGTACACACAGGGACAGC.................................................................................................................................................................................... | 24 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................AGTACACAGAGGGACAGC.................................................................................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................GACAGCAGGAAAAAAAAACAAAAGGC................................................................................................................................................................ | 26 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................ATCCGACTGTCTAATTATG........................................................................................................................................ | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................GGACAGAAGGAAAAAAAAA........................................................................................................................................................................ | 19 | 1 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................AGTCCGACTGTCTAATTA.......................................................................................................................................... | 18 | 1 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................GTCCGACTGTCTAATT........................................................................................................................................... | 16 | 1 | 13 | 0.23 | 3 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................TCCGCCGCCTGATTTCGGGA......................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................CAGGGTGTAGATACAGAGAG........................................................................................................................................................................................... | 20 | 3 | 14 | 0.07 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................TAAGTACACGGTGGGAAAG..................................................................................................................................................................................... | 19 | 3 | 16 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................GTCCGACTGTCTAAT............................................................................................................................................ | 15 | 1 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................................GCCATGCATTTCTAATTAC... | 19 | 2 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .............................................................GAGGTACAGGGGGAAAAAA........................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ....................................................AAGTACACACAGGGACGGCA................................................................................................................................................................................... | 20 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
GGAGAAAGGAGGCTCATGCCTGGGTCAGACCGGATGTTAGACTCGTCCCACATTCATGTGTCTCCCTGTCGTCCTTTTTTTTTTCTTTTCGCAGGTCAGGCTTACAGATTAATACTATGTTTAACTTAATTACGTGCTGTGTACGGCGGTAGAGTAATCTCGCTACGTAATCCTGTCGAGTCCTAAACCCGACTACTCGAATGAGGAGGCGGAGGACTAAAGACATGACCGGTACGTATAGATTACTGCGT
**************************************************....(((((.....(((((...(((......................)))...))))).....)).)))........((((((((((..............(((....))))).))))))))..(((((((.....)))))))........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
SRR060657 140_testes_total |
SRR1106723 embryo_12-14h |
SRR060679 140x9_testes_total |
SRR060681 Argx9_testes_total |
GSM1528803 follicle cells |
V053 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ................................................................................................................................................GGCGTTAGAGTAATCACGCTC...................................................................................... | 21 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................GAGGGCTGAAGACATGTCCGGT.................. | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................................CCGGTACGTGTAGATTACC.... | 19 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................GAGGTCTGAAGACATGTCCGGT.................. | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................ACGTGGTGTGTAGGGCGTTAG................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..GGAAAGGAGGCCCATGACT...................................................................................................................................................................................................................................... | 19 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ............................................................TCTCCCTGTCGTCCTTAA............................................................................................................................................................................. | 18 | 2 | 9 | 0.22 | 2 | 0 | 0 | 0 | 0 | 0 | 2 | 0 |
| ...GAAAGGAGGCCCATGACTC..................................................................................................................................................................................................................................... | 19 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ............................................................TCTCCCTGTCATCCTTTA............................................................................................................................................................................. | 18 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..........GGCTCATACCTGGGTCGGT.............................................................................................................................................................................................................................. | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..GGAAAGGAGGCCCATGAC....................................................................................................................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12822:726023-726273 - | dvi_2340 | CCTCTTTC---CTCCGAGTACGGACCCAGTCTGGCCTACAATCTGAGCAGGGTGTAAGTACACAG-------------------------AGGGACAGCAGGAAAAAAAAAAGAAAAGCGTCCAGTCCGAATGTCTAATTATGATACAAATTGAATTAATGCACGACACATGCCGCCA--TCTCATTAGAGCGATGCATTAGGACA-------------------------------------------------------------GCTCAGGATTTGGGCTGATGAG-------------------CTTACTCCTCCGCCTCCTGATTTCTGTACTGGCCATGCATATCTAAT-GACGCA |
| droMoj3 | scaffold_6540:33382473-33382651 - | AGTGTC---------------------------TAA------------------------------------------------------------------------------------------------------TTATGATACAAATTGAATTAATGCACAAGGCA------------------------CGCACAACATTGGAGTCCAA----ATGAGGGGGATGT-AGC----------------------TGCAGCCCACTCCTTCAGATTCGGTTTGCGGATTCGAGCTTCAGGTCGG--CTTTCGCCTTGGCTTTGTGTGTGCGCTGCCAGCCATGCATATCTAAT-GACGCC | |
| droGri2 | scaffold_15074:3061386-3061609 + | TCCAGT---------------------------TGA---------------------------------------------------------------AAGGCCT------------------------------AATTACGGCTCAAATTCAATTAATACACAACACACATACACACA--------------CACACAACGCTGTCATTAAGTCACAGGAGGCG-ATGTTTGCCAAAAACATTATGGCTTGGTATCATCGACCATGCTCATTATTTTAGCTGATGAACTCG--CTCCAGGCTAGATCCTCCTACTCAGCTCTTTGCTTTGTGTACTCATCATGCATATCTAAT-GACGCA | |
| droWil2 | scf2_1100000004943:8973136-8973259 + | CATCTT---------------------------TGA-------------------------TCTCAATCAGTCAATGAGTGGCTACAA-CTCAACTAAACT----------------------------------------------------------------------------------------------------------------------------GTAAGT-AGA----------------------TCAATCTAACTGCAATAGAATTATTCCTCGGGTC----------------------------------TAATT-AT---GGGGCAAAAAATATTTAATTAACGCA | |
| dp5 | Unknown_singleton_4019:239-249 + | CTACCG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACAAT | |
| droPer2 | scaffold_5:6142694-6142740 - | TGAGGA---------------------------GGG----------------CGGGAGTACATA---------------------------AAAGAACCAAAAACAAAAAA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAAAAA | |
| droAna3 | scaffold_13099:1508641-1508707 + | AGCCAG---------------------------GCTATCGATCTGGACAAAATATAAAAAAAAGA-------------------------GAAAGCAACAAAAAACAAGAAAGA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAAGC | |
| droBip1 | scf7180000392274:7565-7632 + | TTAAAC---------------------------TAA-------------------------------------------------------------AGTGCGAGAAA----------------------------------------AGCCAAAATAATATTAAAAAAA------------------------ATTACTAGGACA-------------------------------------------------------------GA--------------------------------------------------------------------------------TCAGAA-TTCGAG | |
| droKik1 | scf7180000302259:2293-2347 + | CAAAGA---------------------------GTT------------------------------------------------------------CGAGGGAAAGAAAAAGGAAAAGCGGCCAGTAGGAGTAATTA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTCGT | |
| droEle1 | scf7180000491212:1548100-1548112 + | ATCTAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T-GGCCTA | |
| droRho1 | scf7180000779367:4314-4385 - | AGGCTG---------------------------GGCAGCGGTCAAAGCAGAGGGAACCGTTAGAG-------------------------AGGGACGGCAGTAAAACAAAACGAAATGCG----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCTA | |
| droBia1 | scf7180000300013:12987-13029 + | TGTGAG---------------------------TAA-------------------------CCAC-------------------------AGACGCAGCAAGAAAAAAAGGAAA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAATCG | |
| droTak1 | scf7180000412859:1286-1331 - | CAGATG---------------------------ACC------------------------------------------------------------AGGTGGAAGAAAAAAAGTAAAGTTTTCAG-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTACAATG | |
| droEug1 | scf7180000408801:3817-3862 + | AGGCCG---------------------------TGA---------------------ATACACTG-------------------------AGAAAAAATAAGAACAAAAAAAGA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATAGC | |
| dm3 | chr2R:1328492-1328531 - | TCTATTT-----TATGAGTACACATACAGTTTGGCCCACA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATCTG | |
| droSim2 | 2l:21868873-21868932 + | AGGAAT---------------------------AAC---------------------------------------------------------------AGGAAAAAAATGAGAAAAAAGATCGGACCAATTAAAGATTCAAG-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------A-GACAAA | |
| droSec2 | scaffold_4:4633146-4633263 - | CCCCTGCAGGATTTCGGCCAG------------GAGCTCGGTCTGTCCACCA--------------------------GTTCCTTCAATCACAGCCAGACGTGAGTTGAACC-----CGGATCCGACCGCATGTCTAATTATGACACAAACTAAA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTGATG | |
| droYak3 | 3L:20498805-20498864 - | GCTGAA---------------------------GA----------------GATTACCGTCGGTC-------------------------TCCGTTTGCCGAGATATGTATC-----TGAGATAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GC-GGCGCA | |
| droEre2 | scaffold_4517:2185-2203 - | ATACGT---------------------------A---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCTGAT-AATGCA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 11:40 PM