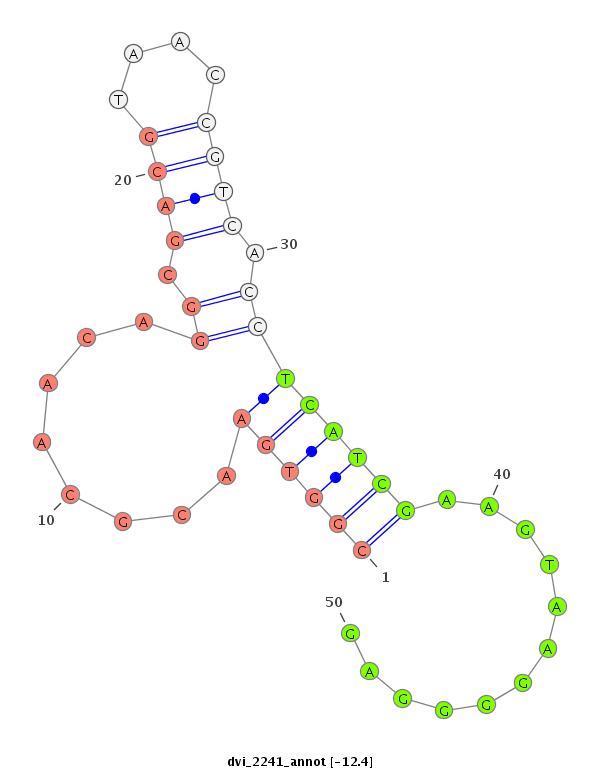
ID:dvi_2241 |
Coordinate:scaffold_12799:250403-250553 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
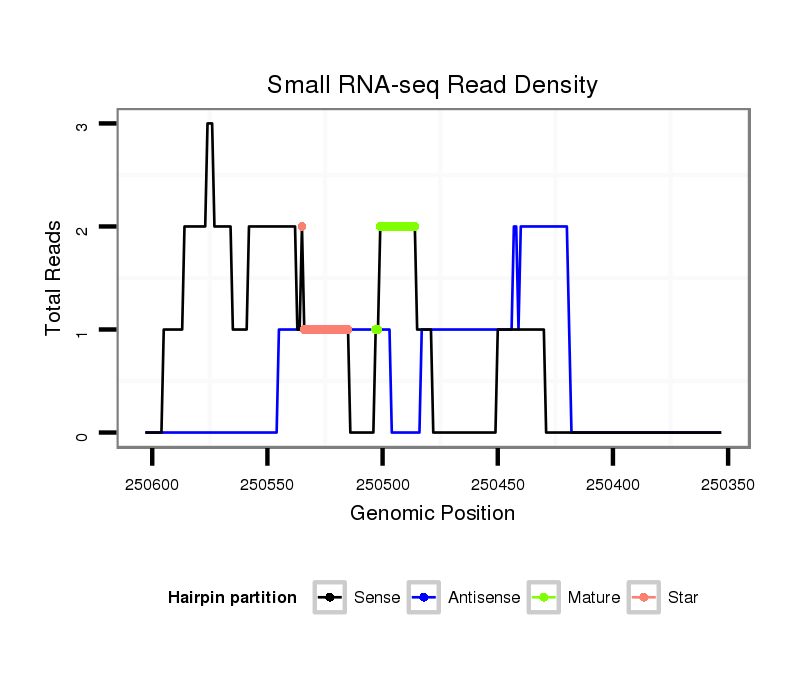
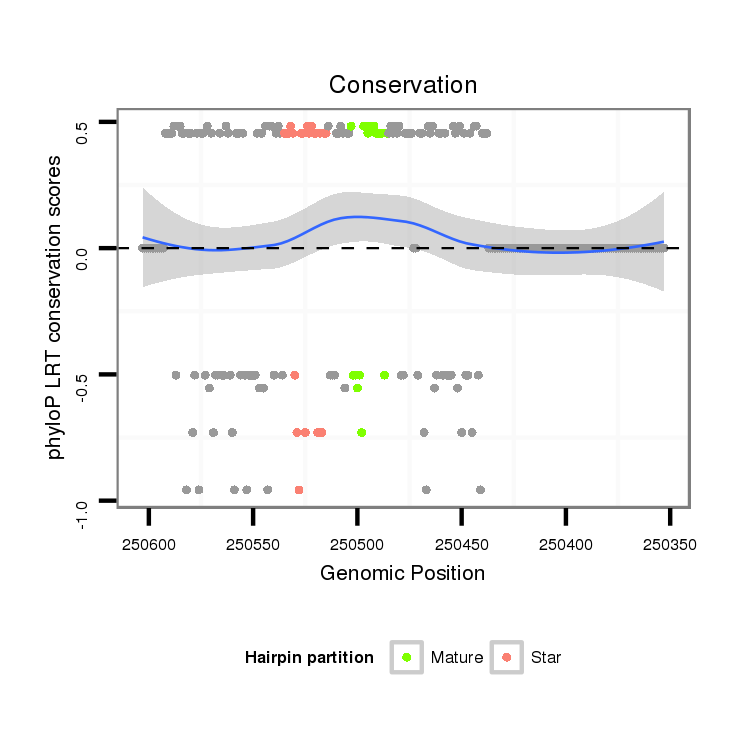
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -12.1 |
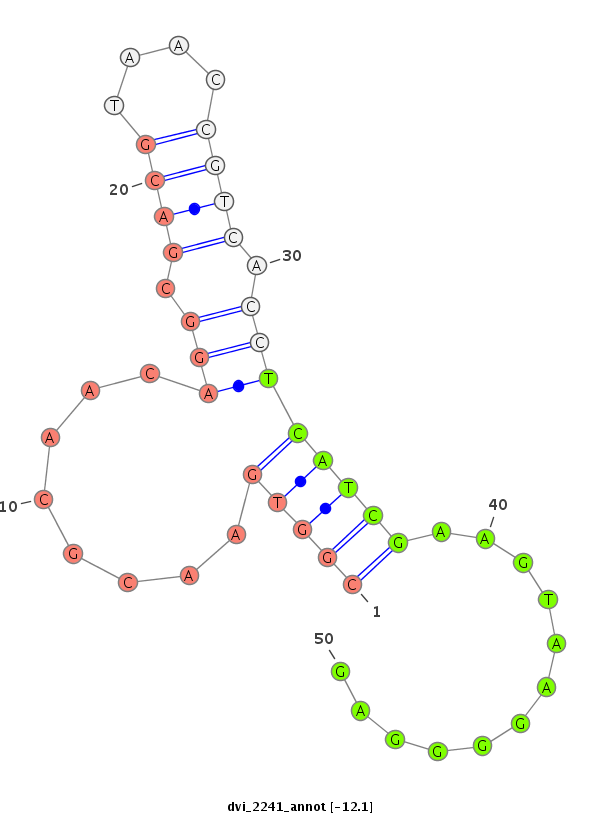 |
CDS [Dvir\GJ19790-cds]; exon [dvir_GLEANR_5057:1]; intron [Dvir\GJ19790-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- AATCCGCCTGGGCCGATATCGGCGAGCCCGTTACCGGGCAACAGGGGGCAGTAAGGACAAGAACGTCGCGGTGAACGCAACAGGCGACGTAACCGTCACCTCATCGAAGTAAGGGGAGGTGTGAAACGGCTAACCAGAGTAGCCACTCTACAATCCTGTTATGCGGCAAGAAGGCGCTAGATCTATAATTTCATTGTTTTGTTAGGTTAAACGTAGCACAAGTATTATTATTAAGAAAGGCAAGGAGCCAC ********************************************************************((((((........((.((((....)))).))))))))............************************************************************************************************************************************* |
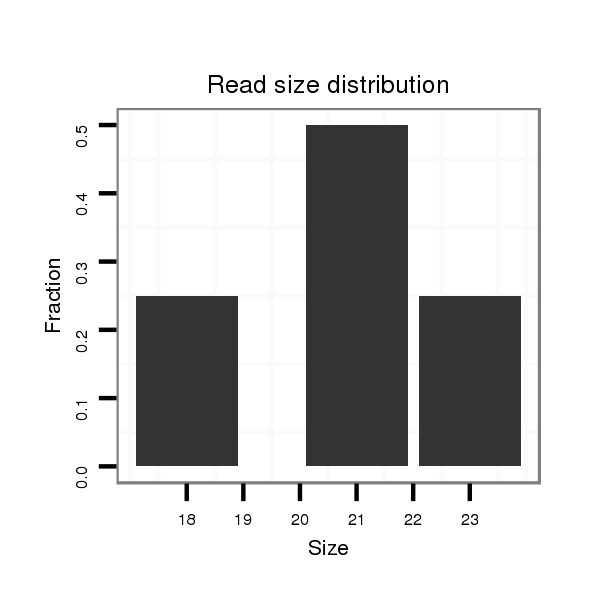
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060673 9_ovaries_total |
SRR060658 140_ovaries_total |
M047 female body |
SRR060656 9x160_ovaries_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060674 9x140_ovaries_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060681 Argx9_testes_total |
SRR060660 Argentina_ovaries_total |
SRR060672 9x160_females_carcasses_total |
V047 embryo |
V053 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................TCATCGAAGTAAGGGGAG..................................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........TGGGCCGATATCGGCGAGCCCG............................................................................................................................................................................................................................. | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................CCGTTACCGGGCAACAGGGGG........................................................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................CAGTAAGGACAAGAACGTCGC...................................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................AAGGGGAGGTGTGAAACGGCTT....................................................................................................................... | 22 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................GGGCAGTAAGGACAAGAACGT......................................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................CGGTGAACGCAACAGGCGACG.................................................................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................TCCTGTTATGCGGCAAGAAGG............................................................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................ATCGAAGTAAGGGGAGGTGTGAA.............................................................................................................................. | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................ATCGGCGAGCCCGTTACCGGG..................................................................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................ACCTCAAGTATTATTACTAAG................ | 21 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................AGAATGTCGCGGTGAATAC............................................................................................................................................................................. | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................TGAACGCAACTGGGGGCGT................................................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .............................................................................................................TAAGAGGAGGTGTGAAAGC........................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ........................................................ATCAGAATGTCGCGGTGAA................................................................................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
|
TTAGGCGGACCCGGCTATAGCCGCTCGGGCAATGGCCCGTTGTCCCCCGTCATTCCTGTTCTTGCAGCGCCACTTGCGTTGTCCGCTGCATTGGCAGTGGAGTAGCTTCATTCCCCTCCACACTTTGCCGATTGGTCTCATCGGTGAGATGTTAGGACAATACGCCGTTCTTCCGCGATCTAGATATTAAAGTAACAAAACAATCCAATTTGCATCGTGTTCATAATAATAATTCTTTCCGTTCCTCGGTG
*************************************************************************************************************************************((((((........((.((((....)))).))))))))............******************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060661 160x9_0-2h_embryos_total |
GSM1528803 follicle cells |
SRR060656 9x160_ovaries_total |
SRR060663 160_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060688 160_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................................................TTTGCTGCATTGGCAGTGGAGTA................................................................................................................................................... | 23 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................................................................CACTTTGCCGATTGGTCTCAT.............................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...................................................................................................................................................................GCCGTTCTTCCGCGATCTAGA................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................TGGCAGTGGAATAGCTTC.............................................................................................................................................. | 18 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................TACGCCGTTCTTCCGCGATCTAGAT.................................................................. | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .............................................................................................................................................CGGTGAGATGTTAGGACAATA......................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................TTCTTGCAGCGCCACTTGCGTTGTCCGCTG................................................................................................................................................................... | 30 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................................CAGCGCGGCTTGCGTTGTCCA...................................................................................................................................................................... | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................CATTGGCAGTGGAGTAGCT................................................................................................................................................ | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .....................................................................CCACTTGCGTTGGCCTTTGC.................................................................................................................................................................. | 20 | 3 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................GCAGCGACACTTGCG............................................................................................................................................................................. | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12799:250353-250603 - | dvi_2241 | AATCCGCCTGGGCCGATATCGGCGAGCCCGTTACCGGGCAACAGGGGGCAGTAAGGACAAGAACGTCGCGGTGAACGCAACAGGCGACGTAACCGTCACCTCATCGAAGTAAGGGGAGGTGTGAAACGGCTAACCAGAGTAGCCACTCTACAATCCTGTTATGCGGCAAGAAGGCGCTAGATCTATAATTTCATTGTTTTGTTAGGTTAAACGTAGCACAAGTATTATTATTAAGAAAGGCAAGGAGCCAC |
| droMoj3 | scaffold_3753:138-290 - | -----------GCCGACATCGCCGCACGCGCTTCAAAGTGACGTCGGACGCCGGAGTCTACAATGTCACGGTGGCGGCCACAGGAGCCGTGGTCGTCTCCTTGATTAAGTAAGGGGGGGTGTGAGGCGGC--GCCCCAGTTACCGCCTCACTAGCTCGGTACCCGG------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
Generated: 05/16/2015 at 09:43 PM