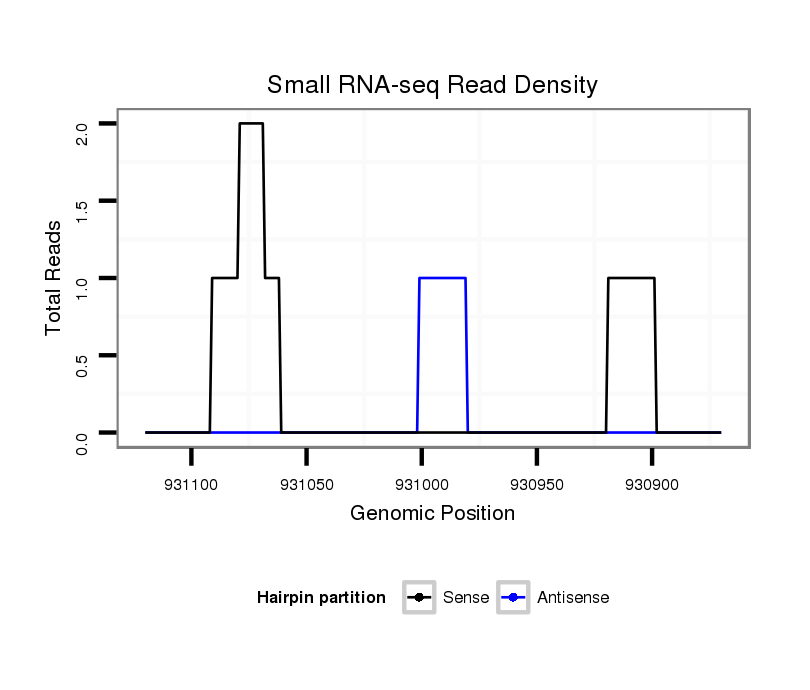
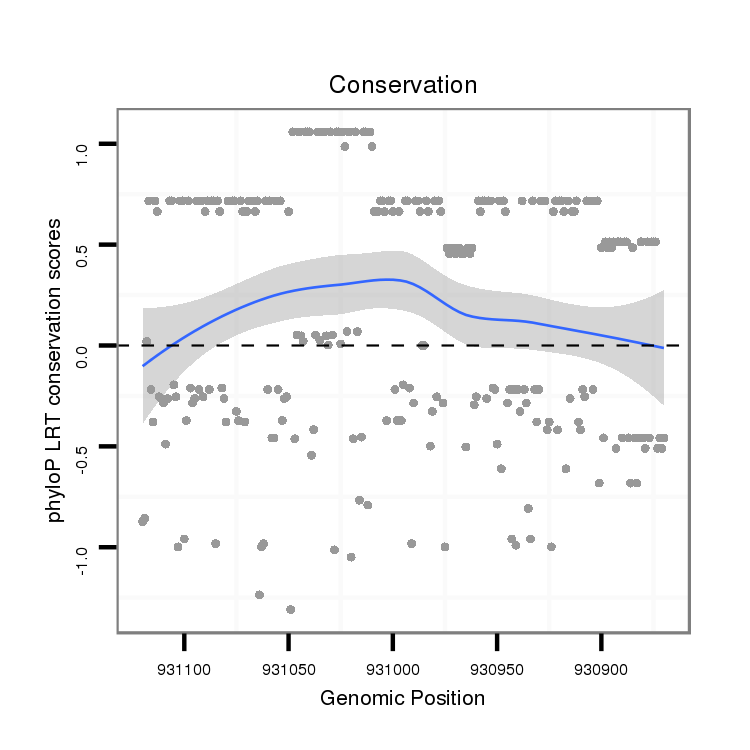
ID:dvi_22258 |
Coordinate:scaffold_13052:930920-931070 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_60:1]; CDS [Dvir\GJ20569-cds]; intron [Dvir\GJ20569-in]
| Name | Class | Family | Strand |
| Galileo_DB | DNA | P | + |
| -------------------------#########################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CTTTTTTGTATATATCTCTTACAATATGAACATAAGACATTATTACGAGTGTAAGTGGGTATTTTTTGTTGAATAAAAAAAGAAAAAAACAAAAACTGCAAATGAAAAGTGCGCATGCATGAGCGCATACATACATACACTATGTGTGTACATCCGTCAGATGTATATTACTGTGTTTAACGAAACAATAGAATAACGATTTCGTACGATAATTAACAAGCCAGTGTAATGAACCGACGTTATCAAAACAT **************************************************....((..(((.((((((((.................)))))))).)))..)).....((((((........)))))).((((((((...))))))))((((((.....))))))((((.((((((((....)))).))))))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060658 140_ovaries_total |
SRR060663 160_0-2h_embryos_total |
SRR060679 140x9_testes_total |
SRR060684 140x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|
| .............................ACATAAGACATTATTACGAGTGT....................................................................................................................................................................................................... | 23 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 |
| .........................................................................................................................................................................................................TCGTACGATAATTAACAAGCC............................. | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 |
| .........................................ATTACGAGTGTAAGTGGG................................................................................................................................................................................................ | 18 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| .............................................................TTTGTTGTTGAATAAAAAA........................................................................................................................................................................... | 19 | 1 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 |
| .............................................................TGTCTTGTTGAATAAAAAAAAAAAAAA................................................................................................................................................................... | 27 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 |
|
GAAAAAACATATATAGAGAATGTTATACTTGTATTCTGTAATAATGCTCACATTCACCCATAAAAAACAACTTATTTTTTTCTTTTTTTGTTTTTGACGTTTACTTTTCACGCGTACGTACTCGCGTATGTATGTATGTGATACACACATGTAGGCAGTCTACATATAATGACACAAATTGCTTTGTTATCTTATTGCTAAAGCATGCTATTAATTGTTCGGTCACATTACTTGGCTGCAATAGTTTTGTA
**************************************************....((..(((.((((((((.................)))))))).)))..)).....((((((........)))))).((((((((...))))))))((((((.....))))))((((.((((((((....)))).))))))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060679 140x9_testes_total |
SRR060680 9xArg_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................................................ACTCGCGTATGTATGTATGTG............................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| .................................................ACATTCACCCTTAACAAAC....................................................................................................................................................................................... | 19 | 2 | 8 | 0.25 | 2 | 0 | 1 | 1 | 0 |
| .............................................................................................................................................................................................................................GTCACATTCCTTAGCTTC............ | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13052:930870-931120 - | dvi_22258 | CTTTTTTGTATATATCT-----------------------CTTACAATATGAACATAAGACATTAT-TACGAGTGTAAGTGGGTATTTTTTGTTGAATAAAAAAAGAAAA-----AAAC-AAAAACTGCAAATGAAAAGTGCGCATGCATGAGCGCATAC--ATACATACA-------------------------------------------------------------CTATGTG------------------TGTACATCCGTCAGATGTATATTACTGTGTTTAACG-AAACAATA-GAATAACGATTTCGTACGATAATTAACAA---GCCAGTGTAA---TGAACCGACGTTATCAAAACAT |
| droMoj3 | scaffold_6498:1290418-1290719 + | AAGTTTTGCTCCAATCC-----------------------ATTTTAACTTAAGCATAAAACAATAT-TAAAAGTGTAAGTCTATAT--TTTAACGTATTAGCAAATGTAAAAATGAAGCAATTAACTGCATATGCTAACTGCGCATGTATGAACACATAT--GTAC--ACCCGTGCGCATACACACCAAGCACACATACACGCAAAGTATAACTCACCCACTTGATCCGAATATGTGCTTGTATGTATATACGTACTTGTACATCCATCAGGTGTAAATTACTCTGCTAATTTAAAGGTATAGAAATTAAGAATTCCTTCGATTACTAATAA-------------------------------------- | |
| droGri2 | scaffold_14822:85084-85390 + | TCTTAGTGTACCTATTTAAAGATGTTGCATATTGCAAACATTTGTAGCATGAACAAAATACGTGATCTACAAGGGTAAGTAATTTTCCTATATTGCAAAAAAAAAGGAAAGAAAAAAAAAAACAACTGCAGTTGCAAAGTGCGCATGTATGTACATATGAATGTAC--ACATGAATACATA----T----------------------------------------CGATATCTATGCATATGCATATATATAAA---------------TATGTATATCTGTGTGCACTCCG-ATGGGAAC-GAATTCTGAATTCGTACGAGTTTTAACAACTGTCTAGTGTTAAGATAAATAGGAATCTTTAAATTTC | |
| droPer2 | scaffold_26:379143-379185 + | TTT--------------------------------------------------------------------------------------------GACAAAAAAAAAAAC-----AAAC-AAAAAATGTATGTAATAAATG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
Generated: 05/17/2015 at 05:25 AM