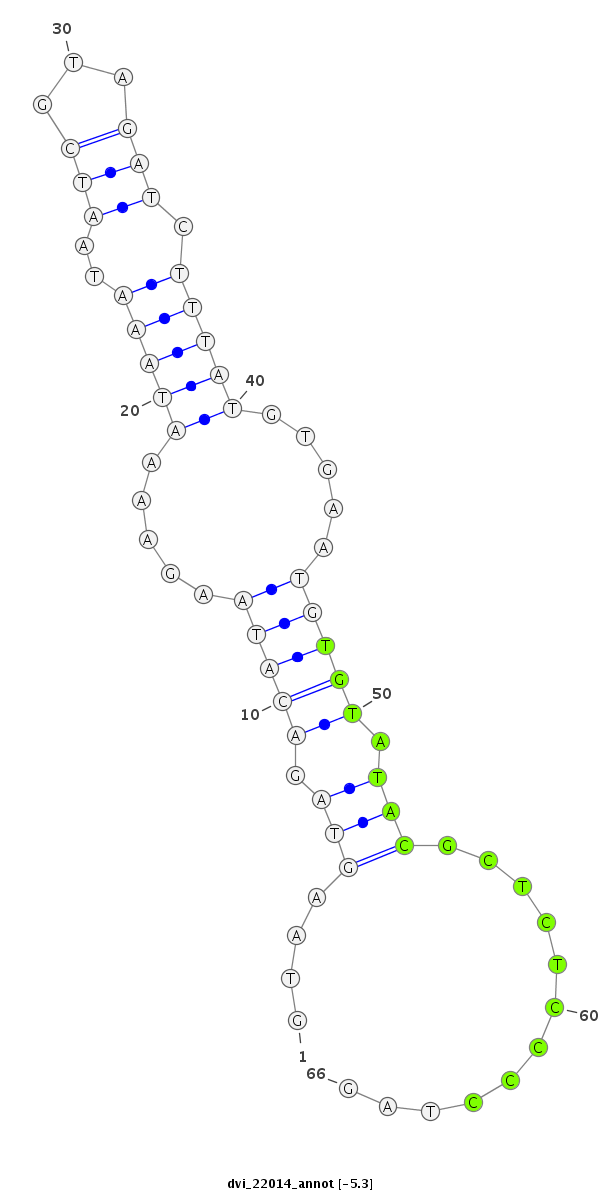
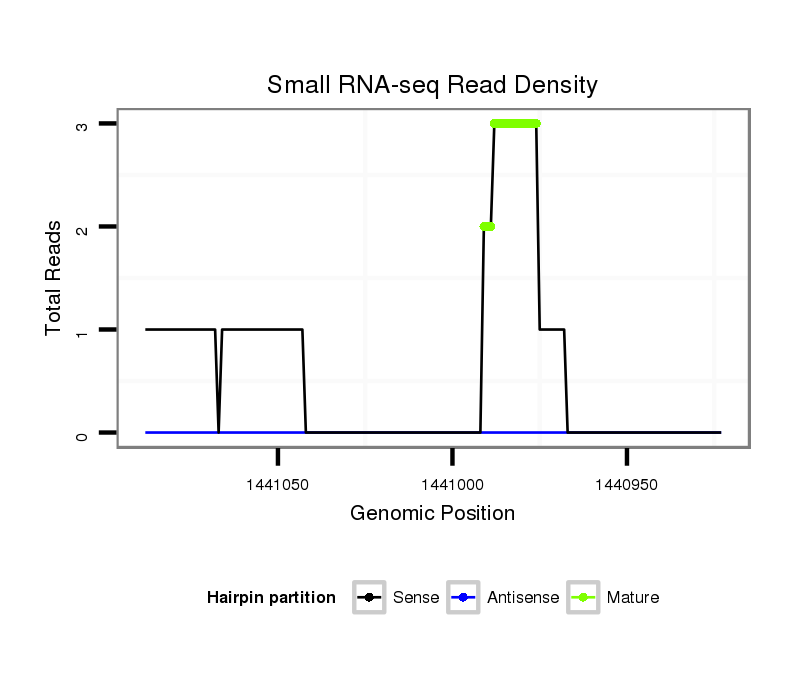
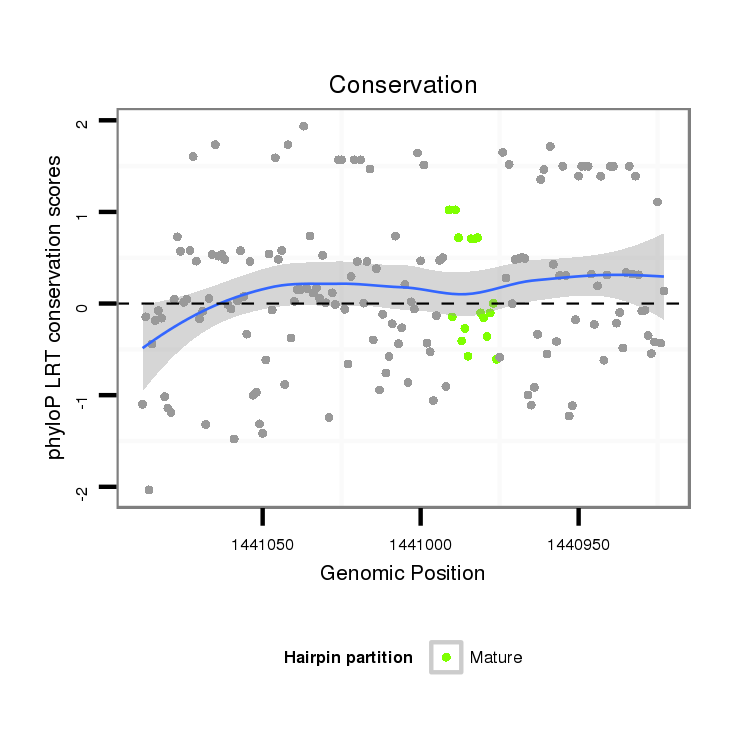
ID:dvi_22014 |
Coordinate:scaffold_13052:1440973-1441038 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -4.5 | -4.4 |
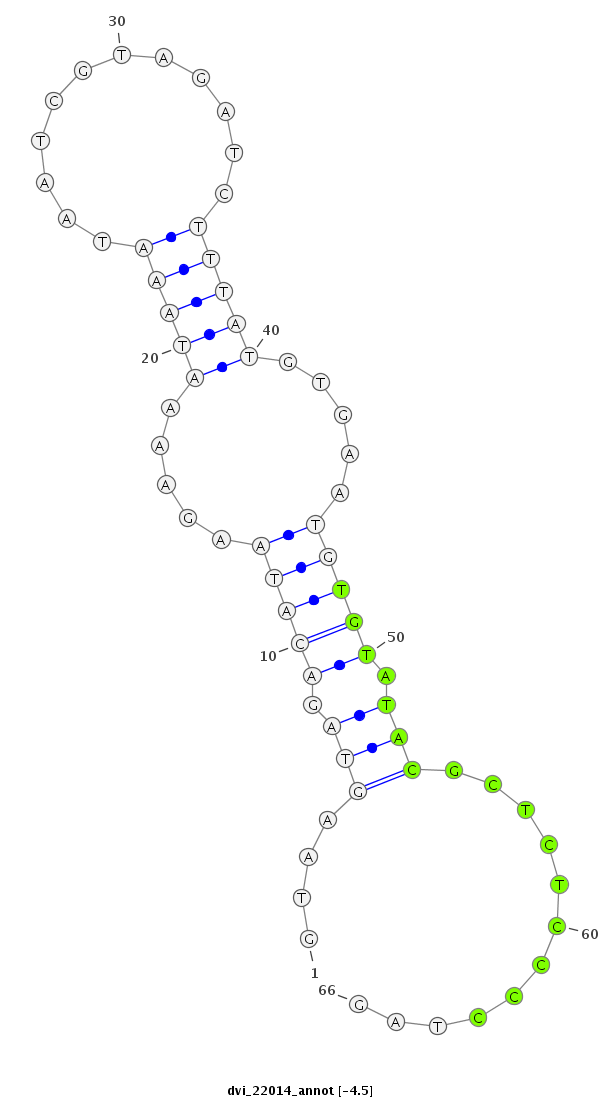 |
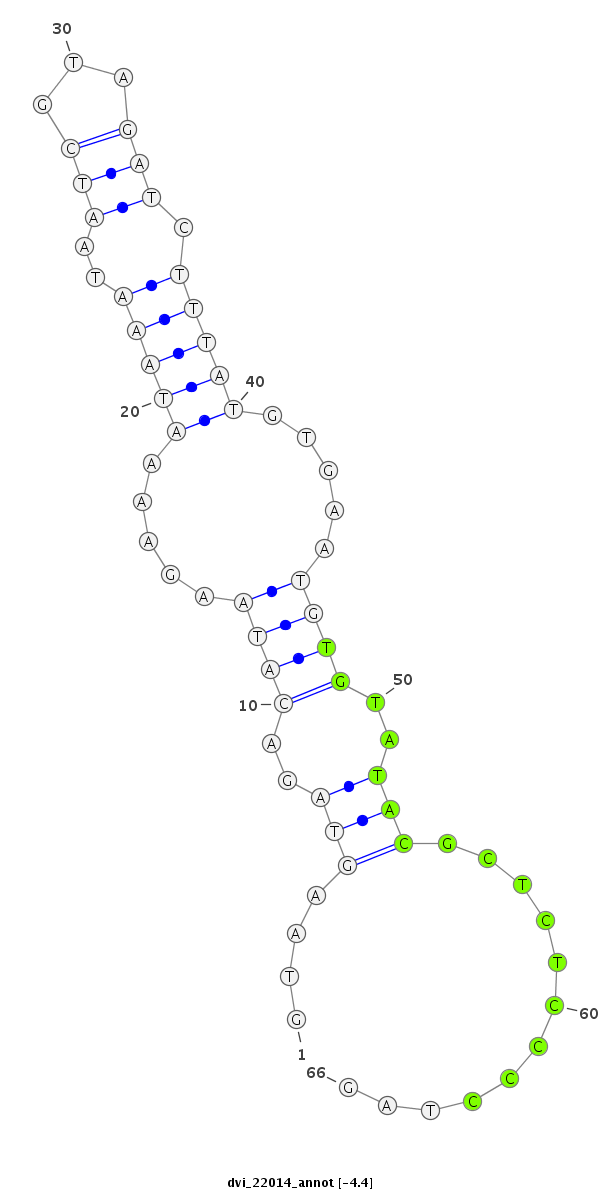 |
CDS [Dvir\GJ18882-cds]; CDS [Dvir\GJ18882-cds]; exon [dvir_GLEANR_37:5]; exon [dvir_GLEANR_37:6]; intron [Dvir\GJ18882-in]
No Repeatable elements found
| ##################################################------------------------------------------------------------------################################################## TTACGTGGATCTTGTTATGTACAAATGGGCAAACCCTCGTTGGCTCTTCAGTAAGTAGACATAAGAAAATAAATAATCGTAGATCTTTATGTGAATGTGTATACGCTCTCCCCTAGGGATTTTTGTGAGTGCATTGCACAAAGTGGCATTGTCGAGGATCACTTTA **************************************************....(((.(((((.....(((((..(((...))).))))).....))))).)))............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
GSM1528803 follicle cells |
M027 male body |
SRR060685 9xArg_0-2h_embryos_total |
V116 male body |
SRR060687 9_0-2h_embryos_total |
SRR060662 9x160_0-2h_embryos_total |
SRR1106726 embryo_14-16h |
SRR060666 160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................................................................TGTATACGCTCTCCCC..................................................... | 16 | 0 | 1 | 2.00 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................ATACGCTCTCCCCTAGGGATT............................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| TTACGTGGATCTTGTTATGTA................................................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................AAATGGGCAAACCCTCGTTGGCTC........................................................................................................................ | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................GTAAGGAGACATAAGAACAAA............................................................................................... | 21 | 3 | 3 | 0.67 | 2 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................GTAAGGAGACATAAGAAAC................................................................................................. | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...CGTGGGTCCTGTTATCTAC................................................................................................................................................ | 19 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................ATACCCTGGTTGGCTCCTCA.................................................................................................................... | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ................................ACCCTGGTTGGCTCCTCA.................................................................................................................... | 18 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...................................................TAAGGAGACATAAGAGACTA............................................................................................... | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ............................................................................................................TCCTTTGGGGATTTTTGTGA...................................... | 20 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .................................................................................................................................................GCACTGTTGAGGATCACGT.. | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
AATGCACCTAGAACAATACATGTTTACCCGTTTGGGAGCAACCGAGAAGTCATTCATCTGTATTCTTTTATTTATTAGCATCTAGAAATACACTTACACATATGCGAGAGGGGATCCCTAAAAACACTCACGTAACGTGTTTCACCGTAACAGCTCCTAGTGAAAT
**************************************************....(((.(((((.....(((((..(((...))).))))).....))))).)))............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060677 Argx9_ovaries_total |
V116 male body |
SRR060663 160_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|
| ........TAGAACAATACACGTTTACCCGT....................................................................................................................................... | 23 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ................................TGGGAGCTACAGAGAAGTG................................................................................................................... | 19 | 3 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 |
| ..............................TTTGGGAGATACAGAGAAG..................................................................................................................... | 19 | 3 | 19 | 0.11 | 2 | 0 | 1 | 0 | 1 |
| ................................TGGGAGATACCGAGAAGTG................................................................................................................... | 19 | 3 | 11 | 0.09 | 1 | 0 | 1 | 0 | 0 |
| ..............................TGTGGGAGCTACAGAGAAG..................................................................................................................... | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13052:1440923-1441088 - | dvi_22014 | TTACGTGGATCTTGTTATGTACAAATGGGCAAACCCTCGTTGGCTCTTCAGTAAGTAGAC---ATAAGAAAATAAATAATC----------------------------------------------GTAGATCTTTATGTGA----------------------ATGTGTATACGCTCTCCCCTAGGGATTTTTGTGAGTGC------ATTGCACAAAGTG------GCATTGTCGAGGATCACTTTA |
| droMoj3 | scaffold_6498:414278-414442 - | CTACGAGGATCGTGCTATGTCCAAATGGGTGAATCTGTATTAGCTTTACAGTAAGTATC-TCT---------------------CAA--------------------TCCCCATAAATGTACATTA-------------TATGAAAACAATGTTGAAATCTG--TCAT---------------ACAGGGATTTCACTGAATGC------ATTCATCAAAATG------GAATTGTCGAAGATCACTTTA | |
| droGri2 | scaffold_14822:460436-460498 - | CTGCGTGGGGCTTGCTATGTTCAAATGGGCAAATCCAGCATGGCTTTGCAGTGAGTATAT---A---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TA | |
| droWil2 | scf2_1100000004943:1148484-1148647 + | TTGCGTGGATCTTGCTATCGTCAAATGTCAATGCCGTCTTTGGCCCTTCAGTAAGT----------------------------------------------------------------------TACAACTACTTATTATGAAGACATTTTTATATTTCGCATATT---------------AAAGGGATCTTGAGGAGTGTATAGCCATTGGTCAAAGCGGACCAGGACTTAAGGAGGACTCATTCC | |
| dp5 | Unknown_group_398:14331-14488 - | CTGCGTGGAGCTTTCTATAGGCAAATGGAGCAGCCGAATCTAGCTCTTCAGTAAGCGTA-TCT-----------------------------------------CGAGCAGAGAAAATTATCCAATTACAATTACCTATTTCG----------------------ATT---------------TCAGGGACTTGCACGATTGC------TTAACACAAAGAG------GAATTAAGGAGGACTTGTTTT | |
| droPer2 | scaffold_42:62477-62634 - | CTGCGTGGAGCTTTCTATAGGCAAATGGAGCAGCCGAATCTAGCTCTTCAGTAAGCGTA-TCT-----------------------------------------CGAGCAGAGAAAATTATCCAATTATAATTACCTATTTCG----------------------ATT---------------TCAGGGACTTGCACGATTGC------TTAACACAAAGAG------GAATTAAGGAGGACTTGTTTT | |
| droAna3 | scaffold_13230:2100782-2100837 + | AGGTTCTC--T------------------------------------TGGATATGCGTC-CGT---------------------CTT-------------------------------------------------------------------------------------------CCCCTCTAAGTATTCTTGTGAGTGG------------------------------------------ATTG | |
| droBip1 | scf7180000394775:7269-7303 - | AATAACTAGTC----------------------------------------------------ATAAAATAATAAATAGTC----------------------------------------------GTAAAT-------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302383:1099588-1099653 + | AAGAAAATAAATAATT---------------------------------------------------------------------------------------------------------------GTAATTGTATCTAATA----------------------ATATATAGTGGCTTTATTCCAAGAGTTTA--------------------------------------------------TATAA | |
| droFic1 | scf7180000453932:180882-180923 - | AAATACAC-------------------------------------------------------ATAAACAAATAAAAAATT----------------------------------------------GTATACATTTA----------------------------------------------------------------------------------------------------------TATAA | |
| droRho1 | scf7180000771378:3413-3495 - | TTTCATGGCGCTTACCAA---AAAGTAAGCAAATT---AATAGTTTTTAAGTGACTATAC---GTAAAAAAATAAAGGTTT----------------------------------------------ATCGAT---------------------------------------------------------------------------------------------------------------TTTTA | |
| droTak1 | scf7180000414050:211838-211901 + | TACTAT------------GTATGAACAGGGAAACCTTATTAAGTTGTACCATAAATATAG---ATAAATAAATAA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATAA | |
| droEug1 | scf7180000409791:67332-67385 + | AATACTTAGAC----------------------------------------------------AAAAGAAAATTAAGAAGA----------------------------------------------TGATAATTTTATATAA----------------------ATGTGT---------------------------------------------------------------------------GTA | |
| dm3 | chr2R:12038066-12038080 + | TTGT--TGTTCT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGTCA | |
| droSim2 | 2r:12707958-12707972 + | TTGT--TGTTCT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGTCA | |
| droYak3 | 3R:1828039-1828135 + | AAATGTTAATT----------------------------------------------------TGAA---AGTGTAGATCCTCTTTCTCCTATTTAATATACTTCAGACGGCGG-------------------------------------------------------------------------------------ATTT------ATTCTACAAAGTC------GCATTTTCAAGGCTCCCCTTA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/16/2015 at 08:35 PM