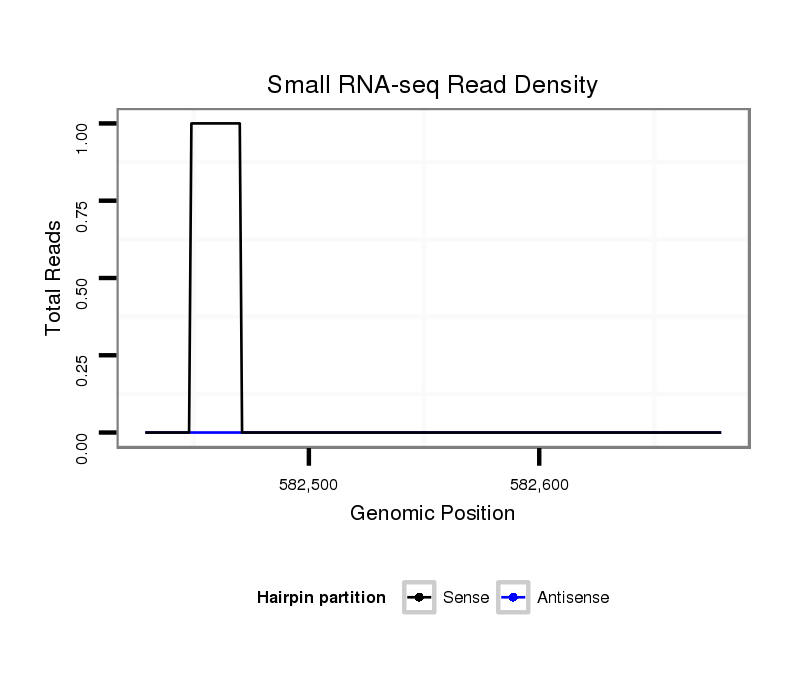
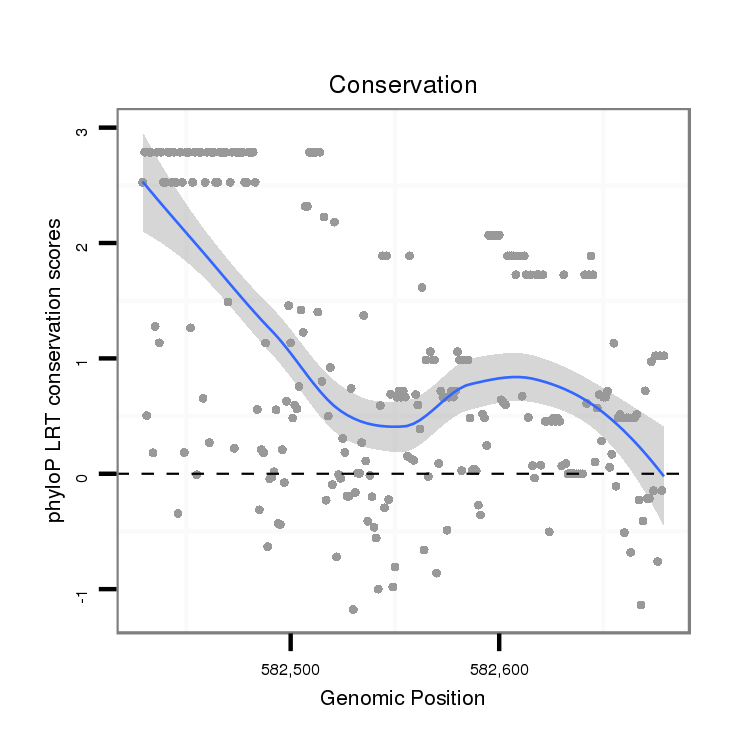
ID:dvi_2185 |
Coordinate:scaffold_12758:582479-582629 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_15942:17]; CDS [Dvir\GJ15520-cds]; intron [Dvir\GJ15520-in]
| Name | Class | Family | Strand |
| (CAGG)n | Simple_repeat | Simple_repeat | + |
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- GAAAAACTATGGAAGACATCGTACGAATTCGAAAAGGATTATCTCATTTGGTAAGCAATGGCAACGTTCAAAACGATTGCAATTATTTTAGCCAACGGCAACGACAACGACATGGTTTCAGCCTGAGTACCTCTTAGCTCCTTTGGGCACTTGTCGCAAATGACAAAATATTTGAAATTGAAGTGCCAGCCAGGCAGACAGCCAAACAGCCAGGCAGTCATGCTGGCAGTCAGGCCGAGCATTGCCTGACT **************************************************..........((((((((...)))).))))..........(((...(((...((.(((......)))))(((.((((.....)))))))......(((((((....((((((......))))))......))))))).))).)))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060679 140x9_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
M047 female body |
SRR1106729 mixed whole adult body |
SRR060663 160_0-2h_embryos_total |
M028 head |
SRR060662 9x160_0-2h_embryos_total |
M027 male body |
SRR1106727 larvae |
SRR060661 160x9_0-2h_embryos_total |
SRR060689 160x9_testes_total |
SRR060671 9x160_males_carcasses_total |
SRR060669 160x9_females_carcasses_total |
SRR060687 9_0-2h_embryos_total |
V047 embryo |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..................................AGGATTAGCTCAGTTGGGA...................................................................................................................................................................................................... | 19 | 3 | 20 | 1.50 | 30 | 0 | 1 | 4 | 5 | 5 | 4 | 4 | 1 | 0 | 2 | 2 | 0 | 0 | 1 | 0 | 1 |
| ....................GTACGAATTCGAAAAGGATTAT................................................................................................................................................................................................................. | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....AACTAAGGAAGACACCGT..................................................................................................................................................................................................................................... | 18 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................GGATTAGCTCAGTTGGTA...................................................................................................................................................................................................... | 18 | 2 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................................AAGCTGGCAGTCGGGCCG.............. | 18 | 2 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................AGTCATGTTGGCAGT..................... | 15 | 1 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................TTTAGCGAACCGCAAGGAC.................................................................................................................................................. | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...................................................................................................AACGAAAACCACATGGT....................................................................................................................................... | 17 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................TTCGAAAAGGCTTAT................................................................................................................................................................................................................. | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................GGGAACGTGCAGAACGATT............................................................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..ACAACTAAGGAAGACACCG...................................................................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
|
CTTTTTGATACCTTCTGTAGCATGCTTAAGCTTTTCCTAATAGAGTAAACCATTCGTTACCGTTGCAAGTTTTGCTAACGTTAATAAAATCGGTTGCCGTTGCTGTTGCTGTACCAAAGTCGGACTCATGGAGAATCGAGGAAACCCGTGAACAGCGTTTACTGTTTTATAAACTTTAACTTCACGGTCGGTCCGTCTGTCGGTTTGTCGGTCCGTCAGTACGACCGTCAGTCCGGCTCGTAACGGACTGA
**************************************************..........((((((((...)))).))))..........(((...(((...((.(((......)))))(((.((((.....)))))))......(((((((....((((((......))))))......))))))).))).)))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060684 140x9_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060663 160_0-2h_embryos_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
M061 embryo |
SRR060661 160x9_0-2h_embryos_total |
SRR060689 160x9_testes_total |
SRR060687 9_0-2h_embryos_total |
V116 male body |
SRR060679 140x9_testes_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060673 9_ovaries_total |
SRR1106730 embryo_16-30h |
M047 female body |
SRR060657 140_testes_total |
SRR060668 160x9_males_carcasses_total |
V053 head |
SRR060680 9xArg_testes_total |
SRR060658 140_ovaries_total |
SRR060670 9_testes_total |
SRR060677 Argx9_ovaries_total |
SRR060681 Argx9_testes_total |
SRR1106729 mixed whole adult body |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................................................................................................................................................................................................CGGCTCGGAAGGGTCTGA | 18 | 3 | 20 | 6.60 | 132 | 28 | 21 | 18 | 12 | 12 | 11 | 6 | 4 | 4 | 4 | 3 | 3 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 1 | 1 |
| .........................................................................................................................................................................................................................................CGGCTCGTAAGGGTCTGA | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................TCGGTACAGTTGCAAGTT.................................................................................................................................................................................... | 18 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................CAGCATGCTGAAGCTTCTC....................................................................................................................................................................................................................... | 19 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................ATCCGGCTCGTAACG...... | 15 | 1 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................AAATAGGTTGCCGTTGCATT................................................................................................................................................. | 20 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................ATGGAGAATTTACGAAACCC........................................................................................................ | 20 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................TTCGTGGGTCCGACAGTAC............................. | 19 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................CACTGTGGGTCCGTCTGT................................................... | 18 | 2 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................CGGCAGTGCGAGCGTCAGT................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .....................................................................................................................................................................................ACACGGTCGGTCGGTCTG.................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12758:582429-582679 + | dvi_2185 | GAAAAACTATGGAAGACATCGTACGAATTCGAAAAGGATTATCTCATTTGGTAAGCAA----------TG--GCAAC-------------------------GTTCAAAACGATTGCAATTAT------TTTAGCC---AACGG----------CAAC---GACA---AC--------------GACA--TGGTTTCAGCCTGAGTACCTCTT-AGCTCCTTT-------GGGCACTTGTCGCAAATG----ACAAAATATTTGAAATTGAAGTGCCAGCCAGGCAGACAGCCAAACAGCCAGGCAGTCAT----------------GCTG--------------------------G-------------------------CAGTCAGGCCGAGCATTGCCTGACT |
| droMoj3 | scaffold_6680:1660416-1660673 + | GAAAAACTATGGAAGACCTCGTACGAATTTGAAAAGGATTATCTCATTTGGTAAGCAA----------TG--TTAACTGCCAAAAAAAAACAAAAAAGGAAAACGAAAATTGTTTGCAATTAT------TTGAGAC---AACGA----------------------------------------GACA--TAGTCTCAAGCTGAGCACCTCTTTTGGTCCCTTCATTTTGGGGCACTTGTCGCAAATG----ACAAAATATTTGAAATTGAAGTGCCTGCCGGGCGGACAGTCT--------GACAGGCACAACTAAGCAACCTGCTGCTG--------------------------G--------------------------------------G----------- | |
| droGri2 | scaffold_15110:894437-894703 - | GAGAAACTATGGAAGACATCGTACGAATTCGAAAAGGATTATCTAATTTGGTAAGCAA----------TG--GCAACGC-----------------------TTTTATAACGATTGCAATTAT------TTAAGAC---AACAA-------------GCACAACACGAACAAATC---------------GAGTCTGTTGCTGAGTATGCTCTTAGCTCCATTC------GGCCACTTGTCGCAAATG----ACAAAATATTTGAAATTGAAGTGCCAGCCGG--------GCC--------GGCAGCAGT-------TAACCTGCTGCTGCCGTTGCTGTTGCTCCTGGGCATTTTC-------------------------CAGTCTGGACGAGG----------- | |
| droWil2 | scf2_1100000004511:10446005-10446099 - | GAGAAACTATGGAAGACGTCATATGAATTCGAGAAGGATTATCTCATTTGGTAAGACT----------GC--TACTC------A-----------------------AAAAAAT---AATTCT------TTGGTTCGACAACAA-------------C---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | XR_group8:3022597-3022683 + | GAGAAGCTGTGGAAGACATCCTACGAATTCGAAAAGGATTATCTTATTTGGTAAGCGA-C--------TG--GCGAC-------------------------ATTTGAGCCCATTGCAATTAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_24:28616-28771 - | GAGAAGCTGTGGAAGACATCCTACGAATTCGAAAAGGATTATCTTATTTGGTAAGCGA-C--------TG--GCGAC-------------------------ATTTGAGCCCATTGCAATTATACTTTC---GGAC---TG----------------CAAA---ACGGAGGACT----------TCACCAGG----------------------CGCTCCTC---------------TGGCGCAAGTGTGGCGCAAAATATTTG------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droAna3 | scaffold_13337:2964607-2964769 + | GAGAAGCTGTGGAAGACGTCATACGAGTTCGAAAAGGATTATCTTATTTGGTAAGGCT----------GG--GAGTC------A--A---------------TGAGAAGCCCAGGGCAATTATACTTTTGTCAGCC---AGGGG----------------------------------------GCGA--TGGTATGA-------CACTTCC-------------------------------------------AAATATTCGAAATTGAAATGGCC-CCGG--------TCC--------GGCAGTCA-------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000396378:271449-271611 + | GAGAAGCTGTGGAAGACATCATACGAGTTCGAAAAGGATTATCTTATTTGGTAAGCCT----------GGGAGTGAA-------------------------TGGGAAGCCCAGGGCAATTATACTTTTGTCAGCC---CGGGG----------------------------------------ACGA--TGGTATGA-------CACTTCC-------------------------------------------AAATATTCGAAATTGAAATGGCC-CCGG--------TCC--------GGCAGTCA-------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302351:495095-495357 + | GAGAAGCTGTGGAAGACGTCGTACGAATTCGAAAAGGATTATCTCATTTGGTAAGCAGACCAGGGGGAGG--CCATG-------------------------GCTTAAGCCCCTTGCAATTATACTTCTGTCAGCC---TGAGGGGA----GAG---GGAA---AGGGAGGAGTGGGTGGGTGGTCAGCAGTGTGTGA-------CATTTCCT----------------G-------------GGTTA----AAGGAATATTCCAAATTGAAATGGCC-CCGG--------TCC--------GGCAGTCAT------------------------------TGCTGCAC--------GCAAATCCCAATACCAATCGTAGTGGCC----------CCCTCACTTAATT | |
| droFic1 | scf7180000454065:692086-692259 - | GAGAAGCTGTGGAAGACCTCGTACGAGTTCGAGAAGGATTATCTCATTTGGTAAGCTG----------GGGGGCCCC-------------------------GCTTAAGCCCATTGCAATTATACTTGTGTCAGCC---GAAGGG---------------------------------------GCGG--CAGTGTCA-------CACTTCCT----------------G-------------GGGGC----CCGTAATATTCCGAATTGAAATGGCC-CCAG--------TCC--------GGCAGTCA-------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491249:5829398-5829498 + | GAGAAGCTGTGGAAGACCTCGTACGAGTTCGAGAAGGATTATCTCATTTGGTAAGCTG----------GG-----AC-------------------------GTTTAAGCCCATTGCAATTATACTTCTGTCAGCC---ATCGG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droRho1 | scf7180000778890:8083-8168 + | GAGAAGCTGTGGAAGACCTCGTACGAGTTCGAGAAGGATTATCTCATTTGGTAAGCTG----------GG--ACAAC-------------------------GTTTAAGCCCATTGCAATTAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000302428:2104440-2104525 - | GAGAAGCTGTGGAAGACCTCGTACGAATTCGAGAAGGATTATCTCATTTGGTAAGCAG----------GG--ACAAC-------------------------GTTTAAGCCCATTGCAATTAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415941:854698-854889 + | GAGAAACTGTGGAAGACCTCGTACGAATTCGAGAAGGATTATCTCATTTGGTAAGCAG----------GG--ACAAC-------------------------GTTTAAGCCCATTGCAATTATACTTCTGTCAGCC---GGAGGGGGTTGATGGTGAT---GGTG---AG--------------GGGA--CGTTGTCA-------CACTTCCT----------------G-------------GAGCT----ACGTAATATTCCGAATTGAAATGGCCCCCAG--------TCC--------GGCAGTCA-------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409466:2653786-2654010 + | GAAAAGCTGTGGAAGACCTCGTACGAATTCGAGAAGGATTATCTCATTTGGTAAGCAG----------GG--ACAAC-------------------------GTTTAAGCCCATTGCAATTATACTTCTGTCAGCC---GGAGGG-----------------------------------------GG--CTGTGTCA-------CACTTCCT----------------G-------------GGGCT----CCGTAATATTCCGAATTGAAATGGCC-CCAG--------TCC--------GGCAGTCAT-------T-----------TCCATTGACATTGCTGCAC--------GCAAATCCCAATACCAATCGTAGTGGC-----------------CCTCACT | |
| dm3 | chr3L:4870731-4870816 + | GAGAAGTTGTGGAAGACCTCGTACGAGTTCGAAAAGGATTATCTCATTTGGTAAGCAG----------CG--GCAAC-------------------------ATTTAAGCCCATTGCAATTAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | 3l:4816046-4816131 + | GAGAAGTTGTGGAAGACCTCGTACGAGTTCGAAAAGGATTATCTCATTTGGTAAGCAG----------CG--GCAAC-------------------------ATTTAAGCCCATTGCAATTAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_2:4857905-4857990 + | GAGAAGTTGTGGAAGACCTCGTACGAGTTCGAAAAGGATTATCTCATTTGGTAAGCAG----------CG--GCAAC-------------------------ATTTAAGCCCATTGCAATTAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | 3L:5455921-5456006 + | GAGAAACTGTGGAAGACCTCGTACGAGTTTGAAAAGGATTATCTCATTTGGTAAGCAG----------CG--GCAAC-------------------------ACTCAAGCCCATTGCAATTAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4784:7588689-7588774 + | GAGAAGCTGTGGAAGACCTCGTACGAGTTCGAAAAGGATTACCTCATTTGGTAAGCAG----------CG--GCAAC-------------------------ACTTGAGCCCATTGCAATTAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 08:56 PM