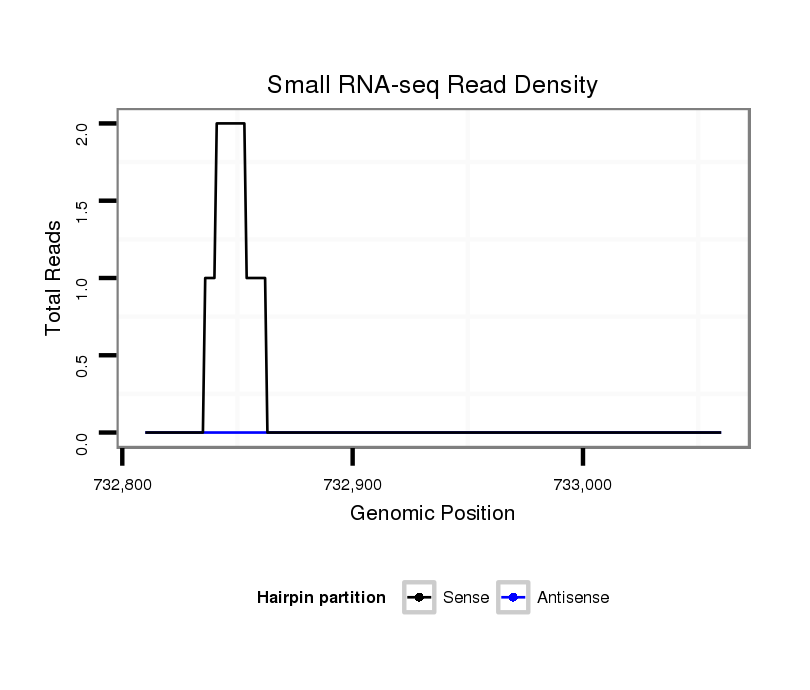
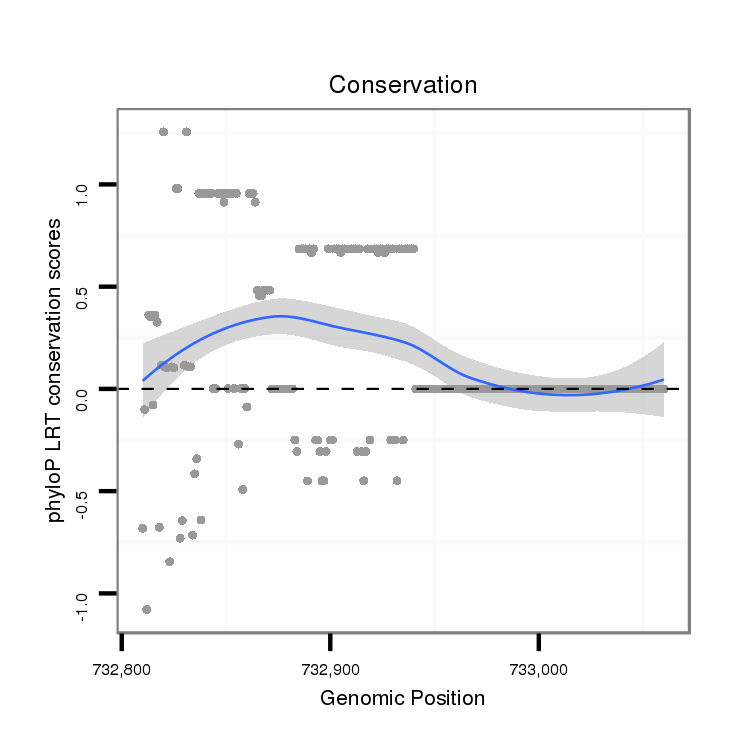
ID:dvi_21810 |
Coordinate:scaffold_13052:732860-733010 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_126:1]; CDS [Dvir\GJ12646-cds]; intron [Dvir\GJ12646-in]
No Repeatable elements found
| --------------------------------##################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CTTTAGATAAACTGTTTTGAATAACTTTTAAAATGGATTCTGAAGTTGTGGTAAGTGCAAAAGCTGAAAATTTGTATTTTTCATCAAGTTCATTACAATTTATATAATTCAAACTTCATCTACTTCATTTTATGTCGTGCATTCTTTAGATATACAAAGTACTTGACAATTAATTGCTATATATAATTTGAACTCTGCCCACCGGGAAAAAGGTATATATTGGTTTCGATACCAATGAAGACTAAAAATTT **************************************************..(((((((((.........))))))))).....((((((((........(((((((((......(((..(((((.....((((((............))))))..)))))..))).....))))).)))).......)))))).))....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060665 9_females_carcasses_total |
SRR060667 160_females_carcasses_total |
SRR060666 160_males_carcasses_total |
V053 head |
M028 head |
SRR060662 9x160_0-2h_embryos_total |
M027 male body |
SRR060671 9x160_males_carcasses_total |
V047 embryo |
M047 female body |
SRR060661 160x9_0-2h_embryos_total |
SRR060668 160x9_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................AATGGATTCTGAAGTTGTGGTA...................................................................................................................................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................TTTAAAATGGATTCTGAA............................................................................................................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................TTAACAATGGATTCTGAGGTT............................................................................................................................................................................................................ | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................AAATGGATTGTGAAGTTGTGA........................................................................................................................................................................................................ | 21 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................ATGGATTCTGAAGTTGTGAA....................................................................................................................................................................................................... | 20 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................AATGGATTCTGAGGTTGC.......................................................................................................................................................................................................... | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................AATGGATTCTGAGGTTGCT......................................................................................................................................................................................................... | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 |
| ....................ATAATTTTGAAAATGGATTC................................................................................................................................................................................................................... | 20 | 2 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................GAACTCAGCCCACCAAGAA........................................... | 19 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................................................AGTGCAAAGGCTGAAATTCT.................................................................................................................................................................................. | 20 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...................AATAATTTTGAAAATGGATT.................................................................................................................................................................................................................... | 20 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................TTATGGTAAGTGCAAAAGGTGTA....................................................................................................................................................................................... | 23 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................................................CTATACCACTGAAGACTC....... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................................................GATAGTGCAAAAGCTGAAT...................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
|
GAAATCTATTTGACAAAACTTATTGAAAATTTTACCTAAGACTTCAACACCATTCACGTTTTCGACTTTTAAACATAAAAAGTAGTTCAAGTAATGTTAAATATATTAAGTTTGAAGTAGATGAAGTAAAATACAGCACGTAAGAAATCTATATGTTTCATGAACTGTTAATTAACGATATATATTAAACTTGAGACGGGTGGCCCTTTTTCCATATATAACCAAAGCTATGGTTACTTCTGATTTTTAAA
**************************************************..(((((((((.........))))))))).....((((((((........(((((((((......(((..(((((.....((((((............))))))..)))))..))).....))))).)))).......)))))).))....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060687 9_0-2h_embryos_total |
V116 male body |
SRR060682 9x140_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060660 Argentina_ovaries_total |
SRR060679 140x9_testes_total |
SRR060669 160x9_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................TAAGACATCAACACCATTTA................................................................................................................................................................................................... | 20 | 2 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................TAGGTTTGAAGTAGCTAAAGT............................................................................................................................ | 21 | 3 | 7 | 0.14 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................AAATATATTGAGTTGGAAGTAGAA................................................................................................................................. | 24 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..................................................................................................AAATATATTGAGTTGGAAGTAG................................................................................................................................... | 22 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................TTGCCCTAAGACTTCAAGA.......................................................................................................................................................................................................... | 19 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..................................................................................................................................................ATCTGTATGTTGCATGGAC...................................................................................... | 19 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ....................................................................TTAAACTTAAAAGGGAGTTCAA................................................................................................................................................................. | 22 | 3 | 15 | 0.07 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................AAGATTGTACCTAAGACT................................................................................................................................................................................................................ | 18 | 2 | 16 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13052:732810-733060 + | dvi_21810 | CTTTAGATAAACTGTTTTGAATAACTTTTAAAATGGATTCTGAAGTTGTGGTAAGTGCAAAAGCTGAAAATTTGTATTTTTCATCAAGTTCATTACAATTTATATAATTCAAACTTCATCTACTTCATTTTATGTCGTGCATTCTTTAGATATACAAAGTACTTGACAATTAATTGCTATATATAATTTGAACTCTGCCCACCGGGAAAAAGGTATATATTGGTTTCGATACCAATGAAGACTAAAAATTT |
| droMoj3 | scaffold_6498:1468102-1468163 - | CCATAGATCAACCTACTTTGATTTTACTGAAAATGGATTCTGAAGTCGAGGTAAGTGCAAAA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004511:11364332-11364398 + | AAAAAATTA----------------------------------------------------------------AAATTTGTCACTTCTATTGTTACAATTTATTTTCATTAAACTTCATTTGATTTATTTT------------------------------------------------------------------------------------------------------------------------ | |
| droAna3 | scaffold_13077:109522-109524 + | ATT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000396733:469837-469888 - | TTCTAAAATAATTGTT---AATAAATTTTAAAATAAATTCTAAAATCAAATTAAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4770:3789046-3789070 - | CTTTTGATAGACTTTTTTTTGTAAC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 04:50 AM