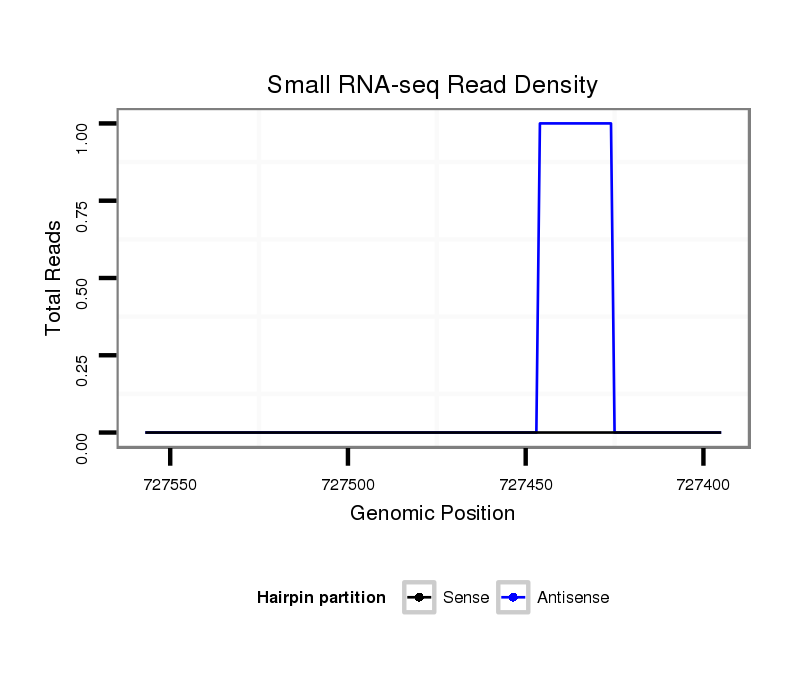
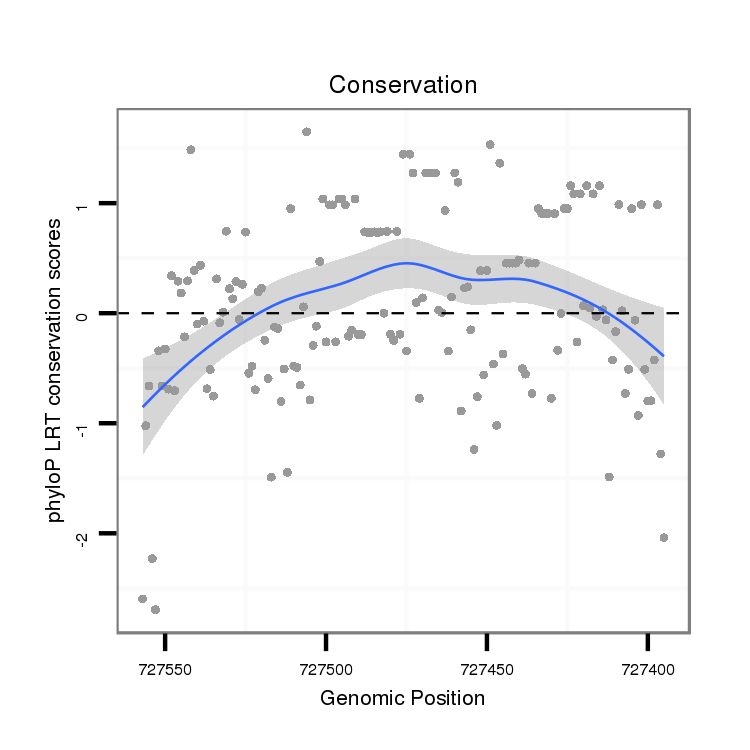
ID:dvi_21809 |
Coordinate:scaffold_13052:727445-727507 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ21551-cds]; exon [dvir_GLEANR_69:1]; exon [dvir_GLEANR_69:2]; CDS [Dvir\GJ21551-cds]; intron [Dvir\GJ21551-in]
No Repeatable elements found
| ##################################################---------------------------------------------------------------################################################## GTGACACGTCTGCAAGCCACCAAGCAGGCTGTCAAACAGAAAATAGAGATGTGAGTTCCCCAAGAATTTTGGAGTGTCAATTCTTGATATTTGCCGAACGTAGCCTTTTGTAGCCCCAAACGCTGGCCCAGAAAGGCGAAGAAAACGGAGCCCACAGCTGTAC **************************************************.((.((((..((((.(((..(((((....))))).))).))))..)))).))...........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060679 140x9_testes_total |
SRR060677 Argx9_ovaries_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060671 9x160_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|
| ..............................GTCAAACAGAAAATAGAGATGTGAA............................................................................................................ | 25 | 1 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 |
| ...............................TCAAACAGAAAATAGAGATGTGAGT........................................................................................................... | 25 | 0 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 |
| ...............................................................................................GAACGTAGCCTTTTGTAGCCC............................................... | 21 | 0 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 |
| ......................AGCACGCTGTCAAACAGA........................................................................................................................... | 18 | 1 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 |
| ............CAAGCCACCAAGGATGCT..................................................................................................................................... | 18 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 | 0 |
| ..........................................................................TTTCAATTCTTGATCTTTG...................................................................... | 19 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 1 |
|
CACTGTGCAGACGTTCGGTGGTTCGTCCGACAGTTTGTCTTTTATCTCTACACTCAAGGGGTTCTTAAAACCTCACAGTTAAGAACTATAAACGGCTTGCATCGGAAAACATCGGGGTTTGCGACCGGGTCTTTCCGCTTCTTTTGCCTCGGGTGTCGACATG
**************************************************.((.((((..((((.(((..(((((....))))).))).))))..)))).))...........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
SRR060659 Argentina_testes_total |
SRR060683 160_testes_total |
V047 embryo |
GSM1528803 follicle cells |
SRR060671 9x160_males_carcasses_total |
SRR060685 9xArg_0-2h_embryos_total |
M047 female body |
SRR1106730 embryo_16-30h |
M027 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................................................................TCGGGGTTTGCGACCGGGTCT............................... | 21 | 0 | 2 | 1.00 | 2 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............GCGGTGTTTCGTCCGACAGTAT............................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............TCGGTGTTTCGTCCGACCGTAT............................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............CGGTGGTTCGACCTACAGTAT............................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............AGGTGGTTCGTCCGACACTGT............................................................................................................................... | 21 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................GTCCGACAATTTGTCTTTTCT...................................................................................................................... | 21 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...............................................................................................................................CGTCTTTCCGCTTATTTTG................. | 19 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..CAGTGCCGTCGTTCGGTGG.............................................................................................................................................. | 19 | 3 | 9 | 0.22 | 2 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................GGGTCTATCAGCTTCTTTT.................. | 19 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........................GTCCGACAATTTGTCTTTTC....................................................................................................................... | 20 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................GTCCGACAATTTGTCTTTTCTA..................................................................................................................... | 22 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................TCCGACAATTTGTCTTT......................................................................................................................... | 17 | 1 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .....................................................................................................TCGGTAAACATCGGGGTCTT.......................................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........CTCGTTCGGTGGTTCGTC........................................................................................................................................ | 18 | 2 | 17 | 0.12 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| ......................................TTTTTAACTCTACACTCAAAG........................................................................................................ | 21 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..CCGTGCCGTCGTTCGGTGG.............................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................CAGTTTGTGTTTTATCT.................................................................................................................... | 17 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13052:727395-727557 - | dvi_21809 | GTGACACGTCTGCAAGCCACCAAGCAGGCTGTCAAACA-----GAAAATAGAG-----------------------------------A---------------TG-------------------TGAGTTCCCCAAGAATTTTGGAGTGTCAATTCTTGATATTTGCCGAACGTAGCCTTTTGTAGCCCCAAACGCTGGCCCAGAA--------------------------------------AGGCGAAG---------------------AAAACGGAGCCCACAGCTGTAC |
| droMoj3 | scaffold_6540:4986729-4986976 - | TTGAAACGTCTGCAAGCCACCAAGCAGGCCATC-----AAGCATGCTATGCATATGCTCATCCCGGAGACCGCCAGCAATCACGTAAGTTGTGGATTTTGGATTATGTACTCGACTGTCTAATTCTTGGC-------------------------TCT------------------GGGTCTTGCAGCCCCAGTCTCTGGCGCGGAAAATATCTCCTCAGCCGCCGGATAACACCAAGTCCGCAAAGGCGAAGCAGCGATCCACAAACAACAAACAGAAAAAGAACAAATTGATTC | |
| droGri2 | scaffold_14624:474878-474918 + | TTTGAAA--------GACACAAAGCAGATTGTCAAACA-----AAAAAAAA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------A---------------AA | |
| droWil2 | scf2_1100000004888:617996-618073 - | GGATAT--------------AGCGAGGGGTGTTTCTCA-----GCT---------------------------------------------GTAAATATTG-------------------GAGTATCAAT-------------------------TTTTTATATTTG--GGACTCAATTTTTTTCAA------------------------------------------------------------------------------------------------------------C | |
| droPer2 | scaffold_25:795770-795807 + | GTGGCAGGCAGGCAGGCAGGCAGGCAGGCTGTC-----AGAC-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------A | |
| droAna3 | scaffold_13337:389432-389460 - | AATGGAG---------------------------------------------------------------------------------T---------------TG-------------------TAAATTCCCCAAGAGT-----------------------------------------------------------------------------------------------------------------------------------------T---------------TT | |
| droBip1 | scf7180000396730:508460-508503 + | GTGACAGCGCCAGGAGCAACTGGACAGACTGAC-----AAACAGACAG-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T | |
| droKik1 | scf7180000301757:4085-4151 - | AAAAAT--------------TCCAGA------------------------------------------------------------------------------AGTTAGTTGGCTATGAAAATATCGAT-------------------------TCTTGGTATTTCTCAAACATATTCTCTT----------------------------------------------------------------------------------------------------------------G | |
| droEle1 | scf7180000491047:1155922-1155956 - | CCACCGACTC-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGGCCCAAAA--------------------------------------AGCCGAAG---------------------AAAGT---------------GG | |
| droRho1 | scf7180000779385:26133-26167 + | CCACCA----------------AGCCGGCAATCAAGCA-----GGAAAGGGAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------C---------------TG | |
| droBia1 | scf7180000300713:10031-10090 + | GTCCTTC---------------G--------------------------------------------------------------------AGACTCTTTG-------------------AAAC--------------------------TTTACTGTTGCCATTTGTCTAACGTATCTGCTTGTAG------------------------------------------------------------------------------------------------------------C | |
| droTak1 | scf7180000415785:372790-372865 - | AAAGCA-------------CTAAGAAGGCTGTCCTTAG-----AGGGTTAA---------------------------------------------------------------------GGATATCGGTTACCAAAGTATCCTGGAGT------------------------ATG-TTTTATTAAT------------------------------------------------------------------------------------------------------------A | |
| droEug1 | scf7180000409774:2295-2331 + | CCAAGAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGGCAAGG---------------------AAAAAAGAAACCCGAGGAATAC | |
| droYak3 | v2_chr3L_random_069:35966-36007 - | GCAATCAGCCAGCCAGCCAACTAGCCAGCTAGC-----AAACAG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T---------------AA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/17/2015 at 04:50 AM