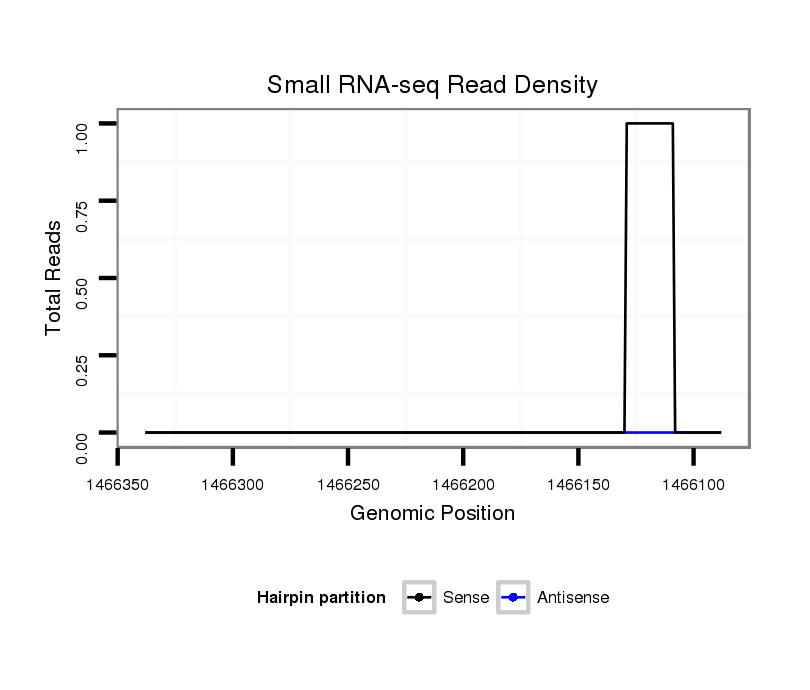
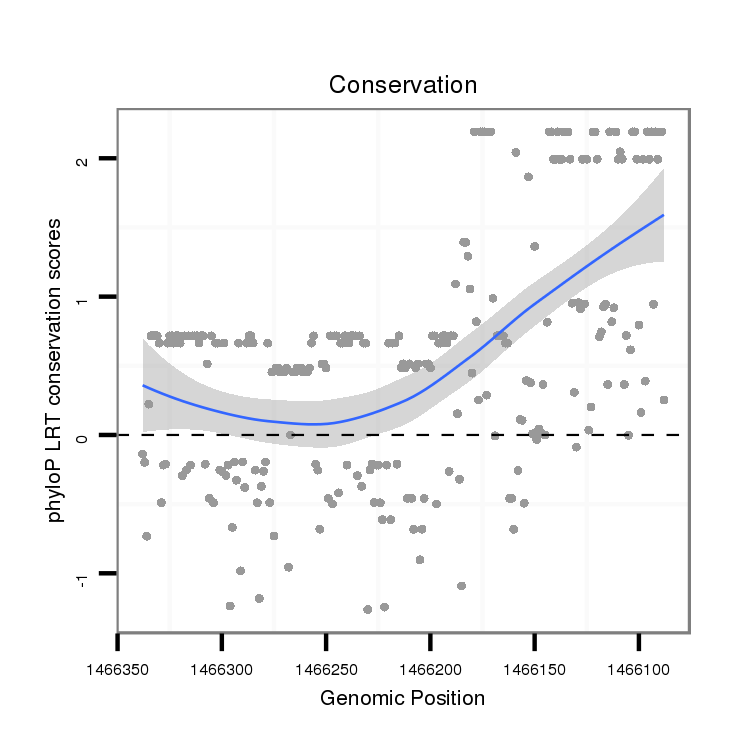
ID:dvi_21708 |
Coordinate:scaffold_13246:1466138-1466288 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
intron [Dvir\GJ16164-in]; CDS [Dvir\GJ16164-cds]; exon [dvir_GLEANR_16610:2]; intron [Dvir\GJ16164-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------##########################################-------- GTACTTTACCAAGAACTTGCAATTATTGTTATATGCCTTGCTGGCGGGCAGTTGAATGTGGCCGAAGGGACCCGAATGCCTCTAAATTCCAAATACCAGCTAACAGGCGTTGCCACGCCCCCTTCTCCATGCTGACAACTTTGCAACTTTGTTCAACTTTGAAATGATTCTAATGCCCCTTTTCTCTCTCTTTCATTGCAGGTAAGTGCAGCTGGAAAGTCATTAGAAGACACCAATGGCCATGTAAGTAA **************************************************(((((((.(((..((((((....((((........))))................(((((....))))))))))).)))(((..........))).....))))))).(((.(((((.........................))))).)))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060657 140_testes_total |
SRR060679 140x9_testes_total |
V116 male body |
SRR060659 Argentina_testes_total |
|---|---|---|---|---|---|---|---|---|---|
| .................................................................................................................................................................................................................AGCTGGAAAGTCATTAGAAGA..................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................................GAAGACACCATTCGCCATGT...... | 20 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 |
| .........................................................GTGGCCGTTGGGACCCGAA............................................................................................................................................................................... | 19 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 |
| ......................................................................................................................................................................................................................................CACCAATGGCCAAGGAAGTC. | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 |
|
CATGAAATGGTTCTTGAACGTTAATAACAATATACGGAACGACCGCCCGTCAACTTACACCGGCTTCCCTGGGCTTACGGAGATTTAAGGTTTATGGTCGATTGTCCGCAACGGTGCGGGGGAAGAGGTACGACTGTTGAAACGTTGAAACAAGTTGAAACTTTACTAAGATTACGGGGAAAAGAGAGAGAAAGTAACGTCCATTCACGTCGACCTTTCAGTAATCTTCTGTGGTTACCGGTACATTCATT
**************************************************(((((((.(((..((((((....((((........))))................(((((....))))))))))).)))(((..........))).....))))))).(((.(((((.........................))))).)))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
V116 male body |
SRR060679 140x9_testes_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060669 160x9_females_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060665 9_females_carcasses_total |
SRR060677 Argx9_ovaries_total |
GSM1528803 follicle cells |
M028 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..................................................................................................................................................................................................................................TTCTGGGCTTACCGGGACAT..... | 20 | 3 | 1 | 6.00 | 6 | 6 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................AAAAATATACGGAACGAC................................................................................................................................................................................................................ | 18 | 1 | 1 | 2.00 | 2 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................................................TTCTGGGCTTACCGGGACA...... | 19 | 3 | 9 | 1.22 | 11 | 11 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................GCGGGGGCAGAGGTACGTC..................................................................................................................... | 19 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................AAAAAAAATATACGGAACGAC................................................................................................................................................................................................................ | 21 | 2 | 5 | 0.40 | 2 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 0 |
| ...................................................................................................................................................................................................................................TCTGGGCTTACCGGGACAT..... | 19 | 3 | 11 | 0.36 | 4 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................GGGGGAAGAGATTCGACT.................................................................................................................... | 18 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................AGCGGGGGCAGAGGTACG....................................................................................................................... | 18 | 2 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................CGGGGGAAGAGATACGTC..................................................................................................................... | 18 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................................................CGGGGGAAGAGGTTCGTC..................................................................................................................... | 18 | 2 | 9 | 0.11 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................CGGGGGAAGAGATTCGAC..................................................................................................................... | 18 | 2 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ....................................................................CTGGGATTACGGAGATT...................................................................................................................................................................... | 17 | 1 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..............................................................................................................................................CGTTGAAAAAAGTTGACAC.......................................................................................... | 19 | 2 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................................................................GGGGGAAGAGATTCGACTA................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................ATGTTCTGTGCTTACC............ | 16 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ....................................................................................................................CGGGGGAAGAGATCCGTCT.................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13246:1466088-1466338 - | dvi_21708 | GTACTTTACCAAGAACTTGCAATTATTGTTA-----------------------TATGCCTTGCTGGCGGGCAGTTGAATGTGGCCGAAGGGACCCGAATGC------------------------------------CTCTAAATTCCAAATACCAGCTAACAGGCGTTGCCACGCCCCCTTCTCC--ATGCTGACAACT--------TTGCAACTTTGTT-CA----------ACT-----------TTGAAATGATTCTAATGCCCCT-----TTTCT----------------CTCTCTTTCATTGCAGGTAAGTGCAGCTGGAAAGTCATTAGAAGA---CACCAATGGCCATGTAAGTAA---- |
| droMoj3 | scaffold_6500:4437782-4438058 + | GCACTTTACCAAGAACTTGCAGTTATTGTTA-------------------------TGCCCAGCTCTCTGACAGTTGGCGAAGGCCTAAGGGAG-CGAATGCACATATACAAAATAACTCTGCCATGGCCTTTAACATCTCTAG-----ACATTCCAACTAACAAGCCCTGTTCGTTCGCCTT-----------------T--------TGGCAACATTGTCTGA----------AC-------TACAGCTGAAATAATTCTAATGC------------CT--T-TTTCAATTTT--CTTTACTCTGTTGCAGGTAAGTGCAGCTGCGAAGTGCTCAGAAGA---CGCCAATGTGCATGTAAGTAAACAG | |
| droGri2 | scaffold_15252:15757361-15757636 + | GCGCTTTACGTGGAACTTGAAATAATTGTTGGGTCTCGAGGCATAAAAACTTGCTGTCCCTTGAAATTGGTTCGTTGACCATAGG-------------------------------------------------------CTGACTTCTAAATTCCAGCTAAAAAGCTTCCCCCCCCCCCCCTCTCTCTACTCTCCTAACTGTCTTCAGTTGCAACTTTGTTTGAGCCCACAGCAAC-------TACAACTATAATTATTCTAATGCTTAT-----TTTTT-------CAA-------CTTACTCTGTTGCAGGTAAGTG---CAG--AAGACACCAACAG----CGATGATGGGCATGTGAGTAA---- | |
| dp5 | 4_group2:743024-743144 - | GC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------A----------ACTTTTTGGTACAACTGCAATAATTT---------TGTCTC---CT--CTCCTCCGCCCTCTCTCTCTCTCGTTGCAGGTAAGTG---CTGCAAAGACATTAGAAGACGGCAACGATGGCCATGTAAGTACTCAA | |
| droPer2 | scaffold_8:3153970-3154088 + | GC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------A----------ACTTTTTGGTACAACTGCAATAATTT---------TGTCTC---CT--CTCCTCCGCCCT--CTCTCTCTCGTTGCAGGTAAGTG---CTGCAAAGACATTAGAAGACGGCAACGATGGCCATGTAAGTACTCAA | |
| droBip1 | scf7180000395973:294292-294396 + | CTCT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTTGCAACTGCAATAATTA---------TTTAACCCCTA--C-------------CTCTTTTTCGTTGCAGGTAAGAAAAGCTGGCAAGACATTAGAAGA---CAATGATGGCCATGTAAGTACGCCA | |
| droFic1 | scf7180000454111:907332-907412 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCAATAATTT---------TC----CTCTT--C-------------CTC-TTTTCGTTGCAGGTAAGTG---CTGCAAAGACATTAGAAGA---CAACGATGGCCGTGTAAGTAC---- | |
| droBia1 | scf7180000302422:8780176-8780257 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCAATAATTT---------TTC---TCCTT--C-------------CTC-TTCTCGTTGCAGGTAAGTG---CTGCAAAGACATTAGAAGA---CAACGATGGCCATGTAAGTAC---- | |
| droTak1 | scf7180000415604:508158-508238 + | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCAATAATTT---------TC----CCCCT--C-------------CTC-TTTTCGTTGCAGGTAAGTG---CTGCAAAGACATTAGAAGA---CAACGATGGCCATGTAAGTAC---- | |
| droEug1 | scf7180000409463:259672-259753 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCAATAATTC---------TCC---TCTTT--C-------------CTC-TTCTCGTTGCAGGTAAGTG---CTGCAAAGACATTAGAAGA---CAACGATGGCCGTGTAAGTAA---- | |
| dm3 | chr2L:7118877-7118958 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCAATAATTT---------TCA---CCCCT--C-------------CTC-TTTTCGTTGCAGGTAAGTG---CTGCAAAGACATTAGAAGA---CAATGATGGCCATGTAAGTAC---- | |
| droSim2 | 2l:6879657-6879738 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCAATAATTT---------TCT---CCCCT--C-------------CTC-TTTTTGTTGCAGGTAAGTG---CTGCAAAGACATTAGAAGA---CAACGATGGCCATGTAAGTAC---- | |
| droSec2 | scaffold_3:2642623-2642704 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCAATAATTT---------TCT---CCCCT--C-------------CTC-TTTTTGTTGCAGGTAAGTT---CTGCAAAGACATTAGAAGA---CAACGATGGCCATGTAAGTAC---- | |
| droYak3 | 2L:5787578-5787674 + | G---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCAACTGCAATAATAT---------TC-----CCCACCT-TCTC--TTCT--CTT-TTTTCGTTGCAGGTAAGTG---CTGCAAAGACATTAGAAGA---CAACGATGGCCATGTAAGTAC---- | |
| droEre2 | scaffold_4929:16034463-16034564 - | CTGC-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCC-CA----------ACTT--GAGTGCAACTGCAATAATTT---------TC-----CCCT--C-------------CGC-TCTTCGTTGCAGGTAAGTG---CTGCAAAGACATTAGAAGA---CAACGATGGCCATGTAAGTAC---- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 04:41 AM