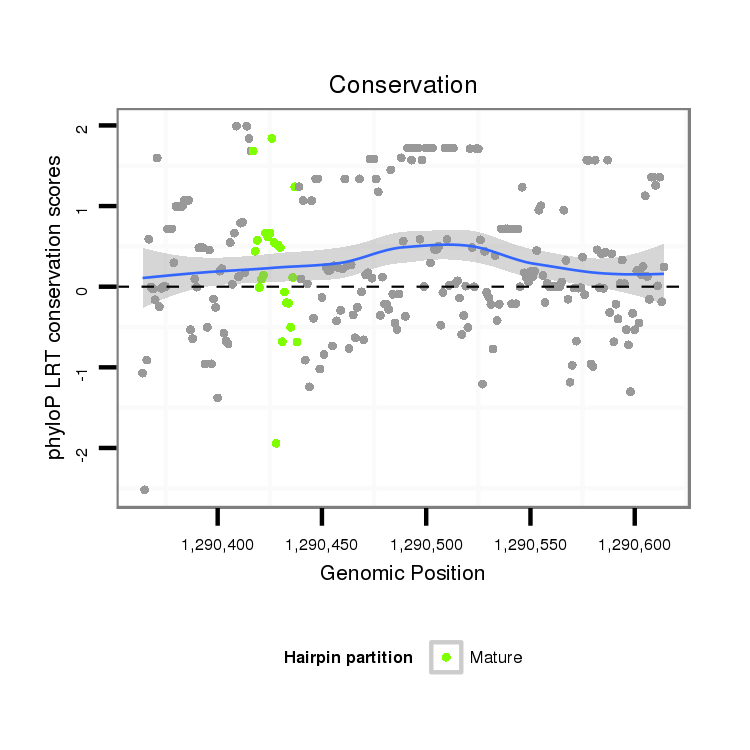
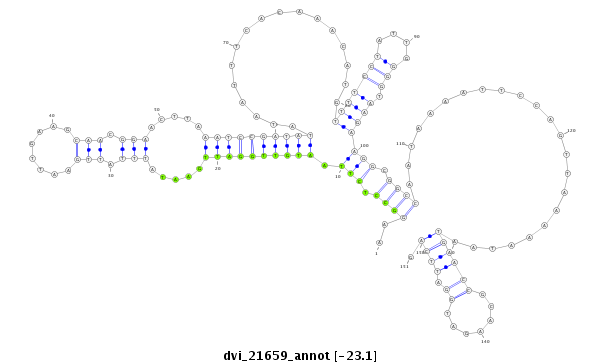
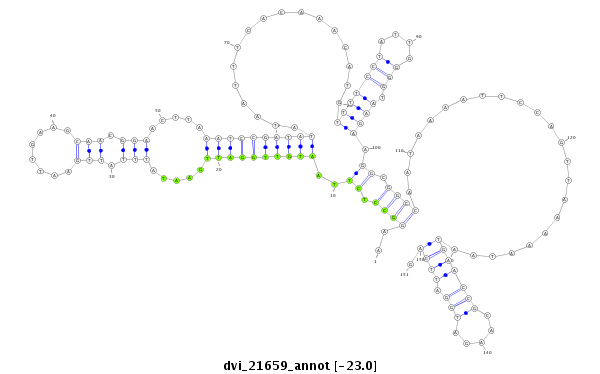
ID:dvi_21659 |
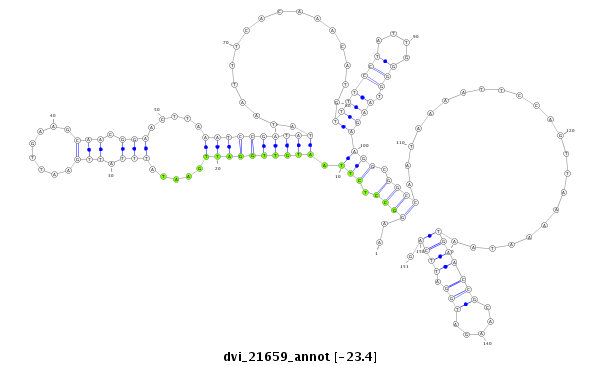
Coordinate:scaffold_13052:1290414-1290564 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -23.1 | -23.1 | -23.0 |
 |
 |
 |
exon [dvir_GLEANR_146:2]; CDS [Dvir\GJ14595-cds]; intron [Dvir\GJ14595-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TGAAAGATCAGTTTTTCCGCATTCATATTTCGCCTTGCATACACAAGATCAAGGCCTCTTAATGTTGGATTGAATATTTATTGAATTGAAGCAACGGAACTTAAATCCGATATATAATTTCACAAACATGTTTTCCTATTGGGGTAAGAAGGCGGCCAATAAAATTCCAGTTAAAAATAATGAACCGCAAGATGGATTCAGTGTTGATCAATCAATTAATAATTTCGAGTTGGAGAGTCACATGTTGGAGA **************************************************..((((.(((.((((((((((.....(((.(((........))).))).....)))))))))).................(((((((....))).))))))).)))).......................(((((((.....))).)))).************************************************** |
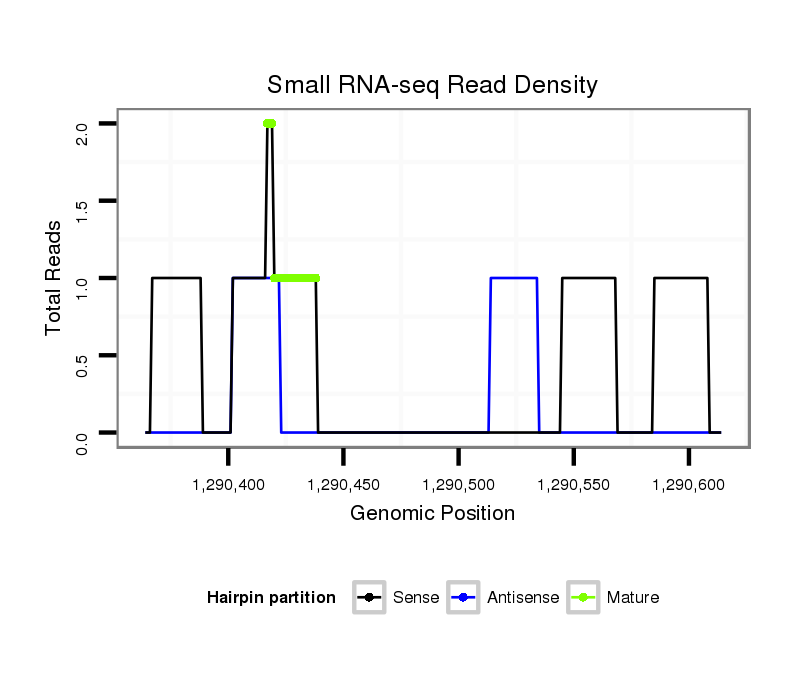
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
M027 male body |
SRR060667 160_females_carcasses_total |
SRR060668 160x9_males_carcasses_total |
SRR060671 9x160_males_carcasses_total |
SRR060681 Argx9_testes_total |
SRR060687 9_0-2h_embryos_total |
V053 head |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................................................GCCTCTTAATGTTGGATTGAAT................................................................................................................................................................................ | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...AAGATCAGTTTTTCCGCATTCA.................................................................................................................................................................................................................................. | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................................ATTTCGAGTTGGAGAGTCACATGT...... | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................ATACACAAGATCAAGGCC................................................................................................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................GAACCGCAAGATGGATTCAGTGTT.............................................. | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................TGAAGCAAGCGGACTTAAATCC............................................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................GTTTCGCCTGGCATACCCAA............................................................................................................................................................................................................. | 20 | 3 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................TTGTTGATTTAAAGCAACGGA......................................................................................................................................................... | 21 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................TATTTCGCCCAGCATACCCA.............................................................................................................................................................................................................. | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................................................................TGAATTGAAGGAACGTAAAT...................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
ACTTTCTAGTCAAAAAGGCGTAAGTATAAAGCGGAACGTATGTGTTCTAGTTCCGGAGAATTACAACCTAACTTATAAATAACTTAACTTCGTTGCCTTGAATTTAGGCTATATATTAAAGTGTTTGTACAAAAGGATAACCCCATTCTTCCGCCGGTTATTTTAAGGTCAATTTTTATTACTTGGCGTTCTACCTAAGTCACAACTAGTTAGTTAATTATTAAAGCTCAACCTCTCAGTGTACAACCTCT
**************************************************..((((.(((.((((((((((.....(((.(((........))).))).....)))))))))).................(((((((....))).))))))).)))).......................(((((((.....))).)))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060671 9x160_males_carcasses_total |
SRR060675 140x9_ovaries_total |
SRR060676 9xArg_ovaries_total |
SRR060682 9x140_0-2h_embryos_total |
M047 female body |
SRR060667 160_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................................................CCGCCGGTTATTTTAAGGTCA................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ................................................AGTTCCGGAGACCTACAAC........................................................................................................................................................................................ | 19 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................TATGTGTTCTAGTTCCGGAGA................................................................................................................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................ACGAGTTCTAGATCCGGA.................................................................................................................................................................................................. | 18 | 3 | 20 | 0.25 | 5 | 2 | 0 | 0 | 0 | 2 | 1 |
| .............................................................................................................................................................................................TCTACCAAAGTCGCAAATAG.......................................... | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13052:1290364-1290614 + | dvi_21659 | TGAAAGATCAGTTTTTCCGCATTCATATTTCG-CCTTGCATAC----------ACAAGATCAAGGCCT-CT-TA-ATG-----TTGGATTGAATA-----TTTATTGAATTGAAGCAACGGAACTTAAAT-----------------------------CCGATATATAATT---T--CACAAACATGTTTTCCTATTGGGGTAAGAAGGC---GGCCA------ATAAAATTCCAGTTAAAAATAATGAACCGCAA---GATGGATTCAGT----------GTTGATCAATCAA----TTAATAATTTCGAG------TTGGA-GAGTCACATGTTGG--AGA |
| droMoj3 | scaffold_6498:1621288-1621508 + | AA----------TTTTCCGCATTATTATTTGAACGTTCAATCG----------ACAAGATCAAGGCCC-TTTTAGATG----------------------TTGAGTAAAATAAAGGAACAGAAACCCTA---------------------------------AAAT-------ACT--GTAGAAAATGTTTTCCTATTTTGCTAAAAAGACGA---TGA------ATAAAGTCTGTAATAAAAATAAT--------AA----TGG------CTCC-AGTAGT-TTTGTTGATGAAACACAAAACAATT------TTGAGATATG-TGATCAAATATTGG--AGA | |
| droGri2 | scaffold_14822:313552-313785 + | TT----------TTTTCCGCATTCATT--------TATCGAGTGATATTCTAGACAAGATCAAGGCCC-AATTA-GTG-----ATGTTTTGTACACATTGTTATCTAAAGTGAATCGTTGGATATTTAA---------------------------------ATATACA--------AATTGAGCATGTTTTCCTACTGGGCCAAAAAAACAGCGGCGA------ATAAAAACGCTGAAAAAAACAGT--------AA----TAA------T--------------AATAATGAAACGTCGAATAATTTAATG---GAGACACG-CAGTGAAATATTGA--AAA | |
| droWil2 | scf2_1100000004963:3138372-3138469 - | GGTAAAATTA-----CCCGCATTTCC---------------------------ATTAGGTTAAGGCTC-TAAAC-ATATTACAC---AAG-----------------AAACAACGCAACAAAA-TGAAAC-----------------------------CCAATA-----------------------------------------------------------------------------------------------------------------------------------------------CAAAACTTAACACAA-------------------AC | |
| dp5 | Unknown_group_139:7001-7233 + | AC---------------------------------------------------ACAAGATCAAGGTCT-TTGTA-ATA----------------------CTTAGTGAATACGAGGACCAGAATTTATATAGCTTGTGAATAGTTCTGCGTGTTCTTTTATGATATACA----AATAAAATGAACATGTTTTCTTACTGGAATAAGAAAAAAATTCAAAGCCGAAACAAACTCGAGG------------AAACAAAAACA----G------TGCCCATTTGT-TCGATAAACGAA------AATTATTTAAAGCCTGACACAGT-TGCATCA--GTTGAAAAAA | |
| droPer2 | scaffold_51:401562-401764 - | AC---------------------------------------------------ACAAGATCAAGGTCT-TTGTA-ATA----------------------CTTAGTGAATACGAGGACCAGAATTTATATAGCTTGTGAATAGTTCTGCGTGTTCTTTTATGATATACA----AATAAAATGAACATGTTTTCTTACTGGAATAAGAAAAC------------------------------------------AAAAACA----G------TGCCCATTTGT-TCGATAAACGAA------AATTATTTAAAGCCTGACACAGT-TGCATCA--GTTGAAAAAA | |
| droAna3 | scaffold_13250:2996612-2996621 + | ACAAAGTTTA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302413:227947-228071 + | TTCACATTCG-----C----------T--------CGCCATAT----------AAAACATCAAGGTCTTTTGTA-ATA-----T---TTGAAATA-----------------------------------------------GTGT---------TTTAGCGATATATTGCTTAAT--AGAAAACATGTTTTCATACTGGAGTAAAAAGAGTATGGCAA------AAAAA-----------------------------------------------------------------------------------------------------------------A | |
| droFic1 | scf7180000453435:25772-25891 + | AA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATATGTTTACCTACTGGAGCAAAAAGGGCATAGCGA------ACAATGTTGAGG------------ATAAAAATACAGAC-----------GAATTTGTGCCTCTGGGCGAA------AAAAGGTCATCT------TCTGAAAAGTCAAAAGTTGA--AGA | |
| droEle1 | scf7180000490710:795-955 + | TTCACATTTG-----C----------T--------AGTCAGAT----------CCAAGATCAAGGTCT-TTTTA-ATA-----C---TCTTAATA-----------------------------------------------GTTT---------TTTG----A-------CTGTT---AAAAAAATGTTTTCCTACTGGAGCAAAAAGAGTATTCCAA------GCAAAGTTGAAG------------AAAAAAGAAATGA-----------AACATTTGTGTCTATAAACGAA------AGTATTTC----------------------------------- | |
| droRho1 | scf7180000773995:14988-15135 - | C------------------------------------------------------AAGATCAAGGTCT-TTGTA-ATA-----G---TCTGAACA-----------------------------------------------ATTT---------CTTA----A-------TTGTT--AAAAAAAATGTTTTCCTACTGGAGCAGAAAGAGTATTCCAA------GCAAAATTGAGG------------AAAAAAAA---GAT---------GAAACATT--GTTGTCAAGCCAA------AATATTTTAGCA------ACAG-------------------AA | |
| droBia1 | scf7180000301737:9338-9345 - | TGAAAAAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| dm3 | chr3L:21925398-21925409 + | TGATA---------------------------------------------------AAACAA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
Generated: 05/21/2015 at 04:33 PM