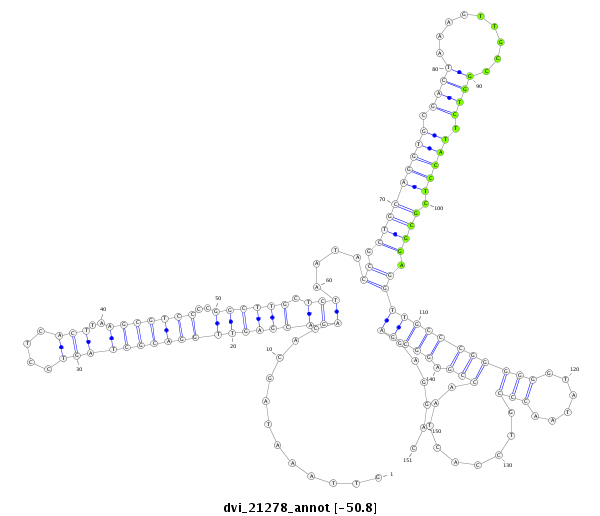
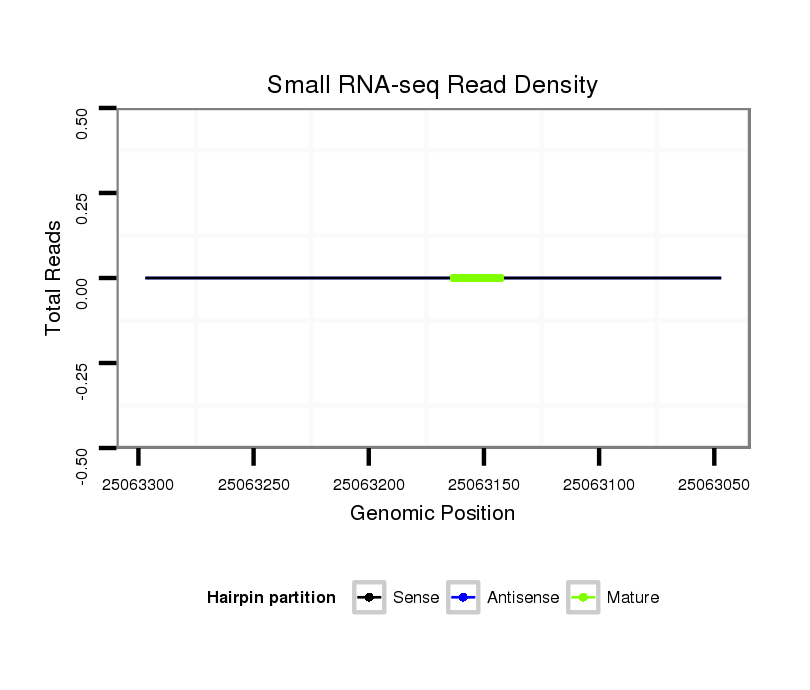
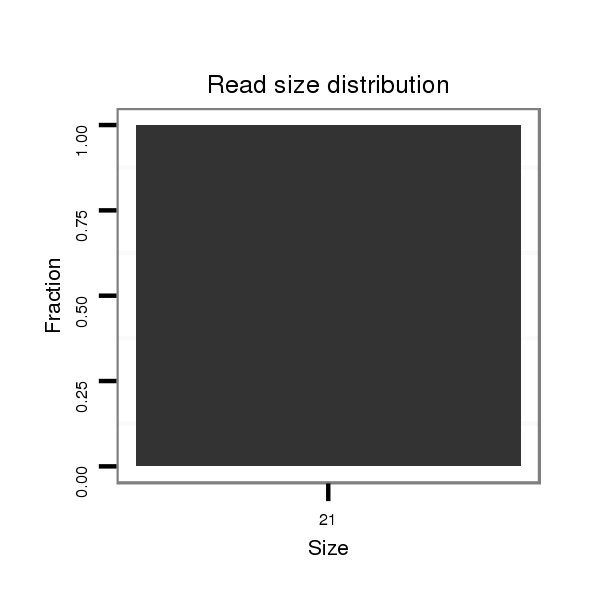
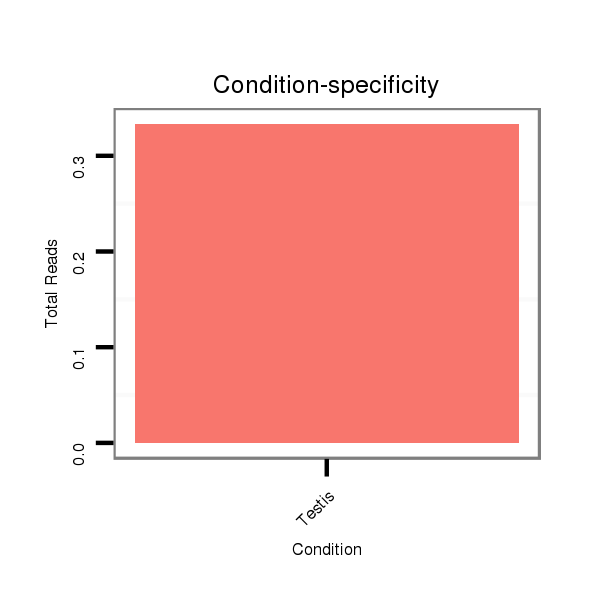
ID:dvi_21278 |
Coordinate:scaffold_13049:25063097-25063247 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
No conservation details. |
| -50.6 | -50.5 | -50.4 |
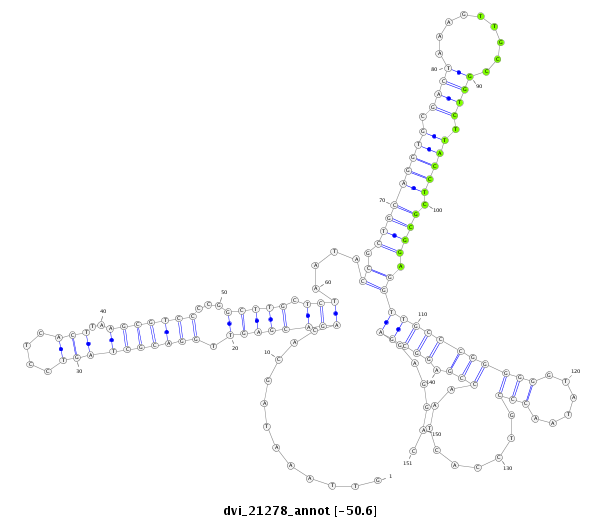 |
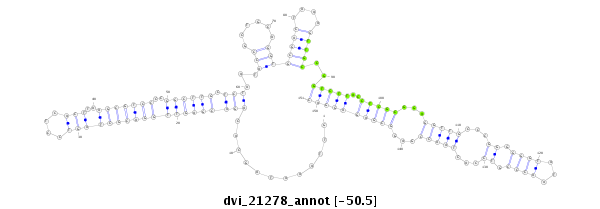 |
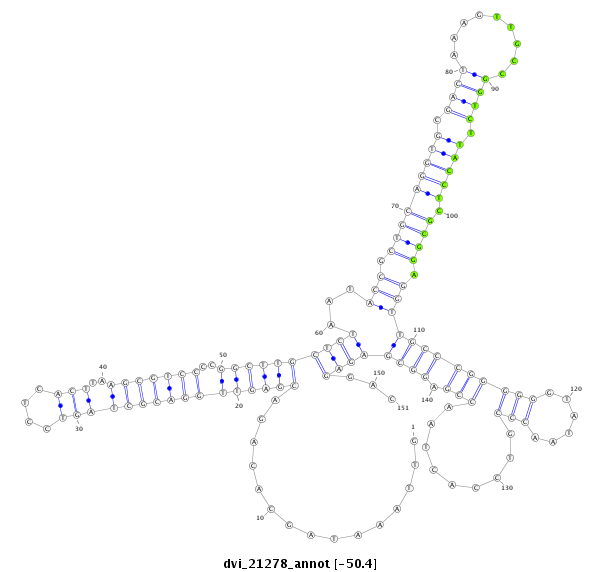 |
exon [dvir_GLEANR_11269:1]; CDS [Dvir\GJ11218-cds]; intron [Dvir\GJ11218-in]
No Repeatable elements found
| mature | star |
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- AGTGATGAATCCTGTTATTTCTTCACGGAGCTGGTGTTCAAAAAGGGCCGGTTAAATAGCACAGACGAGTTGGACGCTAGTCCTCACTTAAGCGTCCCCGGCTTGCTCTAATACCGCTGCAGGTGCGACTAAAGTTGCCGGTCTTACCTCGCGGAGGTTGCCCGGGGGGTATAACCCGTCCACTAACCGAGGCGAGAGGACACATCATCGCACCGGAAGAGGTCCAAATTTGACGACTACGATTCGACGTG **************************************************............(((((((((((((((((((....)))..)))))))..)))))).)))....((.(((((((((.((((.........)))).))))).)))).))(((((((((((......))).........))).)))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060678 9x140_testes_total |
V053 head |
SRR060670 9_testes_total |
V116 male body |
SRR060687 9_0-2h_embryos_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................................TTGCCGGTCTTACCTCGCGGA................................................................................................ | 21 | 0 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................CTAATACCGCTGCAGGTGTCC........................................................................................................................... | 21 | 3 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................GCCGTTCTTAGCTCGCGGA................................................................................................ | 19 | 2 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................................................................................................................................CTAACCTTGGCGAGAGGTC.................................................. | 19 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................................................................................................GGGAGTTTGCCCGGGTGG.................................................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...............................................................................................ACCCGGCTTGCTCTACCA.......................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TCACTACTTAGGACAATAAAGAAGTGCCTCGACCACAAGTTTTTCCCGGCCAATTTATCGTGTCTGCTCAACCTGCGATCAGGAGTGAATTCGCAGGGGCCGAACGAGATTATGGCGACGTCCACGCTGATTTCAACGGCCAGAATGGAGCGCCTCCAACGGGCCCCCCATATTGGGCAGGTGATTGGCTCCGCTCTCCTGTGTAGTAGCGTGGCCTTCTCCAGGTTTAAACTGCTGATGCTAAGCTGCAC
**************************************************............(((((((((((((((((((....)))..)))))))..)))))).)))....((.(((((((((.((((.........)))).))))).)))).))(((((((((((......))).........))).)))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
SRR060668 160x9_males_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
M028 head |
|---|---|---|---|---|---|---|---|---|---|
| .............................................................................................................................................................................................................GTAGCGGGGTCTTCTCAAG........................... | 19 | 3 | 13 | 0.23 | 3 | 3 | 0 | 0 | 0 |
| ....................................................................................................................................................AGCGGCTGAAACGGGCCCCC................................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 |
| ...................................................................TCAAGCTGCGATCGGGATT..................................................................................................................................................................... | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 1 | 0 |
| ...........................................................................................................................................CCAGAATGGTCGGCCTCC.............................................................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:25063047-25063297 - | dvi_21278 | AGTGATGAATCCTGTTATTTCTTCACGGAGCTGGTGTTCAAAAAGGGCCGGTTAAATAGCACAGACGAGTTGGACGCTAGTCCTCACTTAAGCGTCCCCGGCTTGCTCTAATACCGCTGCAGGTGCGACTAAAGTTGCCGGTCTTACCTCGCGGAGGTTGCCCGGGGGGTATAACCCGTCCACTAACCGAGGCGAGAGGACACATCATCGCACCGGAAGAGGTCCAAATTTGACGACTACGATTCGACGTG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
Generated: 05/16/2015 at 11:28 PM