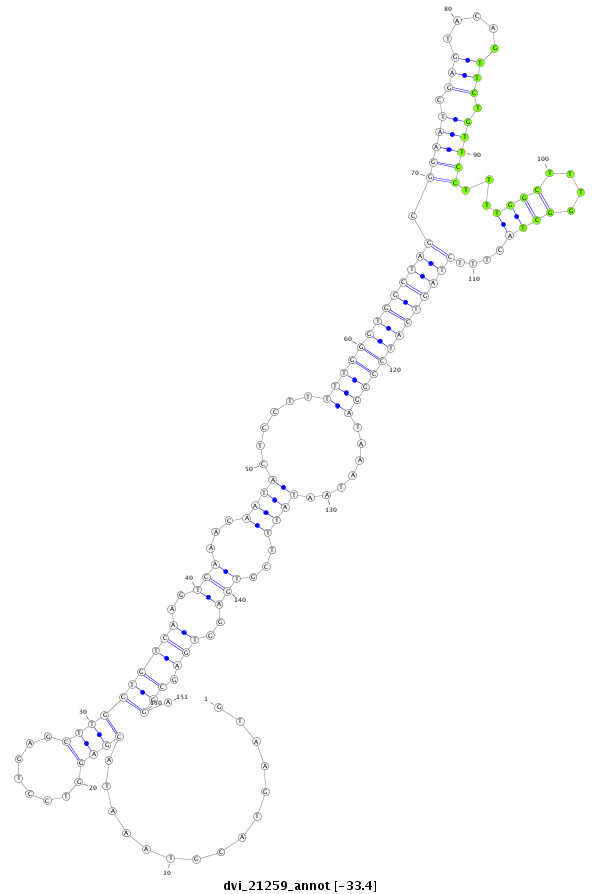
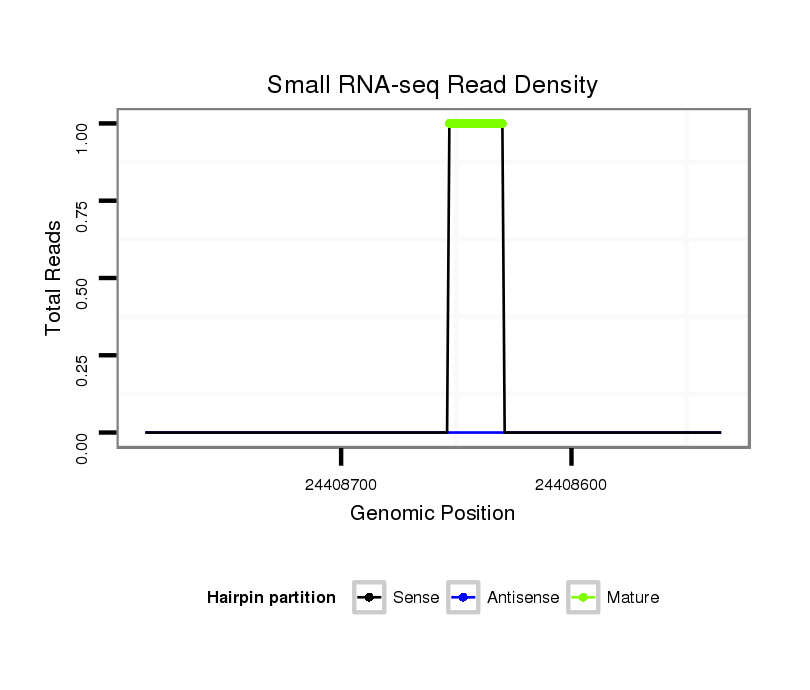
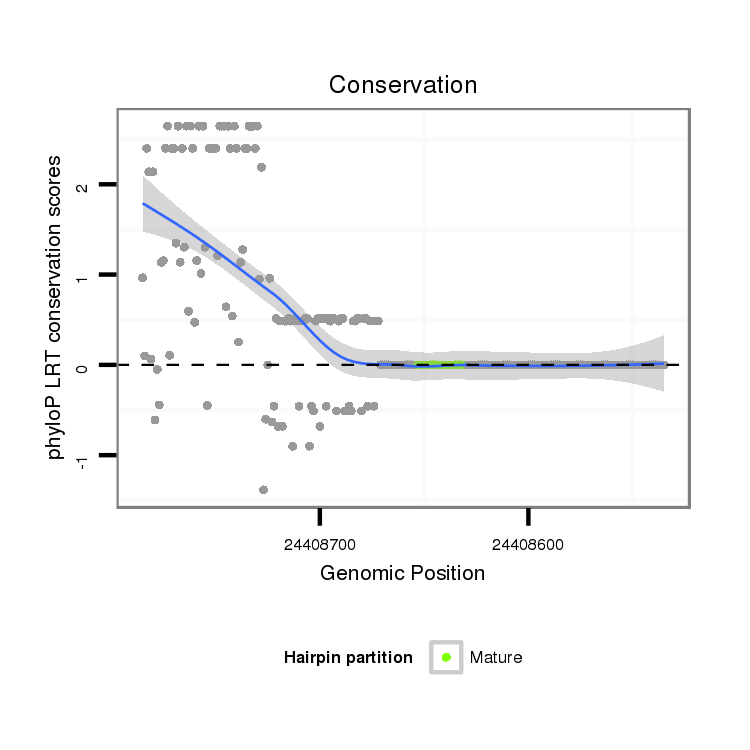
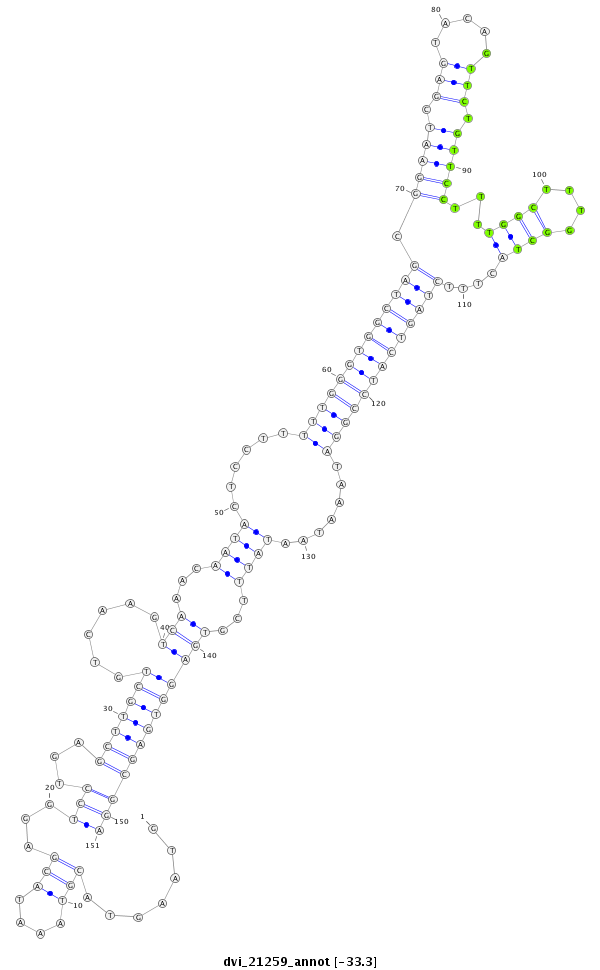
ID:dvi_21259 |
Coordinate:scaffold_13049:24408585-24408735 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -33.3 | -33.1 | -33.0 |
 |
 |
 |
exon [dvir_GLEANR_11273:1]; CDS [Dvir\GJ11223-cds]; intron [Dvir\GJ11223-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- GACGTCATCGCCATCGTTTCGTATCCCTATGTGGCGCTAATAGCACCGTGGTAAGTACGTAAATACGAGGTCCTGAGCTTGCTGTCAAGTCAAACAATACTCCTTTTTGGGTGGCTAGCGGAATCGAGTACAGTTCTGTTCCTTTTGGCTTTGGCTACTTTCTAGTCATCCGGATAAATAATATTTCGTGAGGTGAGCGGAAAAGAGCCGCGTCGACTTTTGTGCCGTGTCAATGTTTACAGAGTGACAGT **************************************************...............((((........))))((((((..(((...((((......(((((((((((((.(((((.(((.....))).)))))...((((....))))....))))))))))))).......))))...)))..))).))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060656 9x160_ovaries_total |
M047 female body |
V053 head |
SRR060657 140_testes_total |
SRR060674 9x140_ovaries_total |
SRR060670 9_testes_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................GTTCTGTTCCTTTTGGCTTTGGCT............................................................................................... | 24 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................CTCCTTGTTGGTTGGCGAGCGG.................................................................................................................................. | 22 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................TAGCACCGTGGTTAGTCAGT............................................................................................................................................................................................... | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................................................................TGGGTGGGTAGGGGAATC.............................................................................................................................. | 18 | 2 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................TATTTGGGTGGCTAGCTGGAT............................................................................................................................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................TGTGAGGTGAGACGAAAAGA............................................. | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................TGTTTGGGTGGCTAGCTGGAT............................................................................................................................... | 21 | 3 | 12 | 0.08 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................................................................GCAGAATCGAGTACACTT.................................................................................................................... | 18 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................................................................................................................................................................................................................................CGGATCAATGTTTAAAGAGT...... | 20 | 3 | 13 | 0.08 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .ACATCATCGGCATCGTTTCT...................................................................................................................................................................................................................................... | 20 | 3 | 17 | 0.06 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................ATCTAGTGCGGTGAGCGG................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................GAGCTAATACCACCATGGT....................................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
CTGCAGTAGCGGTAGCAAAGCATAGGGATACACCGCGATTATCGTGGCACCATTCATGCATTTATGCTCCAGGACTCGAACGACAGTTCAGTTTGTTATGAGGAAAAACCCACCGATCGCCTTAGCTCATGTCAAGACAAGGAAAACCGAAACCGATGAAAGATCAGTAGGCCTATTTATTATAAAGCACTCCACTCGCCTTTTCTCGGCGCAGCTGAAAACACGGCACAGTTACAAATGTCTCACTGTCA
**************************************************...............((((........))))((((((..(((...((((......(((((((((((((.(((((.(((.....))).)))))...((((....))))....))))))))))))).......))))...)))..))).))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
V116 male body |
V047 embryo |
M047 female body |
SRR060668 160x9_males_carcasses_total |
SRR060669 160x9_females_carcasses_total |
SRR060666 160_males_carcasses_total |
SRR060665 9_females_carcasses_total |
SRR060671 9x160_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .............................................................................................................CCAGAGATCGCCCTAGCT............................................................................................................................ | 18 | 3 | 20 | 1.15 | 23 | 0 | 7 | 0 | 4 | 4 | 4 | 0 | 0 | 2 | 2 | 0 |
| .......................................TCTCGTGCCACAATTCATGCAT.............................................................................................................................................................................................. | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................TAGGTATACACCACGATCAT................................................................................................................................................................................................................. | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................ATCGTCGTGGAACCATTCAT.................................................................................................................................................................................................. | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................TCGTCGTGGAACCATTCATG................................................................................................................................................................................................. | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................GACTCGAACAACATTTCA................................................................................................................................................................. | 18 | 2 | 11 | 0.09 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......TAGCGGTAACGAAGCATGG.................................................................................................................................................................................................................................. | 19 | 3 | 17 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...CAGGAGCGGTAGCCAAGCAT.................................................................................................................................................................................................................................... | 20 | 2 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .....................................................................................................................CGGCATAGTTCATGTCAAG................................................................................................................... | 19 | 3 | 17 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:24408535-24408785 - | dvi_21259 | GACGTCATCGCCATCGTTTCGTATCCCTATGTGGCGCTAATAGCACCGTGGTAAGTACGTAAATAC---------------GAGGTCCTGAGCTTGCTGTCAAGTCAAACAATACTCCTTTTTGGGTGGCTAGCGGAATCGAGTACAGTTCTGTTCCTTTTGGCTTTGGCTACTTTCTAGTCATCCGGATAAATAATATTTCGTGAGGTGAGCGGAAAAGAGCCGCGTCGACTTTTGTGCCGTGTCAATGTTTACAGAGTGACAGT |
| droGri2 | scaffold_15110:24317084-24317212 - | GCCGCCCTAACCACCGTTCCATATCTCTATATGGCGCTAACAGTACTAGGGTAAGTGCCCTACCAACCATTAGTAGTAGTAGCGGTCGTGGGCTTCTAGTAAAATCAATCAAATTACCTTATTAGGCGG----------------------------------------------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004540:1455740-1455795 + | AACGCCAACGTCACCGTTCCATTTCATTATATGGCGTTAACAGTACTGTGGTAAGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| dp5 | XR_group6:3595567-3595618 - | GAC------GCCATCGTTCCATTTCCCTCTATGGCGTTAATAGCACTGTGGTAAGTGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_9:1891598-1891649 - | GAC------GCCATCGTTCCATTTCCCTCTATGGCGTTAATAGCACTGTGGTAAGTGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_13337:11323005-11323058 + | -TCGGC---GCCATCGTTCCATTTCCCTCTATGGCGTTAATAGCACTGTGGTAAGTGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000396569:83597-83650 - | -TCGGC---GCCATCGTTCCATTTCCCTCTATGGCGTTAATAGCACTGTGGTAAGTGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302697:256724-256778 - | GTCGTC---GCCATCGCTCCATTTCCCTCTACGGCGTTAATAGCACTGTGGTAAGTGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000454065:1470426-1470482 + | GAC------GCCACCGTTCCATTTCCCTCTACGGCGTTAATAGCACTGTGGTAAGTGCACAAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491000:1280576-1280633 - | GTCGTC---GCCACCGTTCCATTTCCCTCTATGGCGTTAATAGCACAGTGGTAAGTGCCTA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000769817:1103-1157 - | GTCGTC---GCCACCGTTCCATTTCCCTCTACGGCGTTAATAGCACTGTGGTAAGTGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000302334:538273-538327 + | GTCGTC---GCCACCGTTCCATATCCCTCTATGGCGTTAATAGCACCGTGGTAAGTGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415815:85603-85657 - | GTCGTC---GCCACCGTTCCATTTCCCTCTACGGCGTTAATAGCACTGTGGTAAGTGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000408844:584479-584533 - | GTCGTC---GCCACCGTTCCATTTCCCTCTACGGCGTTAATAGCACTGTGGTAAGTGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dm3 | chr3L:15833824-15833878 - | GTCGTC---GCCACCGTTCCATTTCCCTCTATGGCGTTAATAGCACTGTGGTAAGTGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | 3l:15461743-15461797 - | GTCGTC---GCCACCGTTCCATTTCCCTCTATGGCGTTAATAGCACTGTGGTAAGTGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_0:7941942-7941996 - | GTCGTC---GCCACCGTTCCATTTCCCTCTACGGCGTTAATAGCACTGTGGTAAGTGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | 3L:18442751-18442805 - | GTCGTC---GCCACCGTTCCATTTCCCTCTATGGCGTTAATAGCACTGTGGTAAGTGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4784:18087622-18087676 - | GTCGTC---GCCACCGTTCCATTTCCCTCTATGGCGTTAATAGCACTGTGGTAAGTGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 11:00 PM