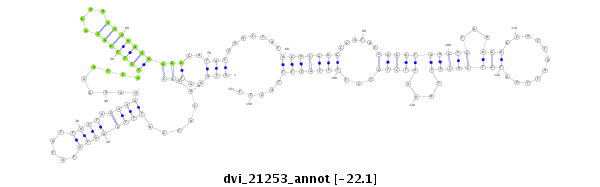
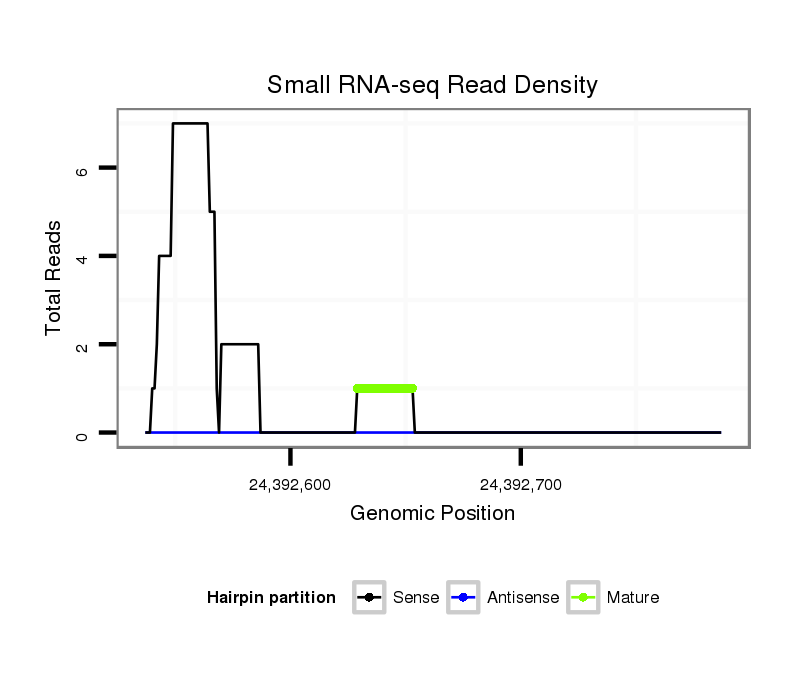
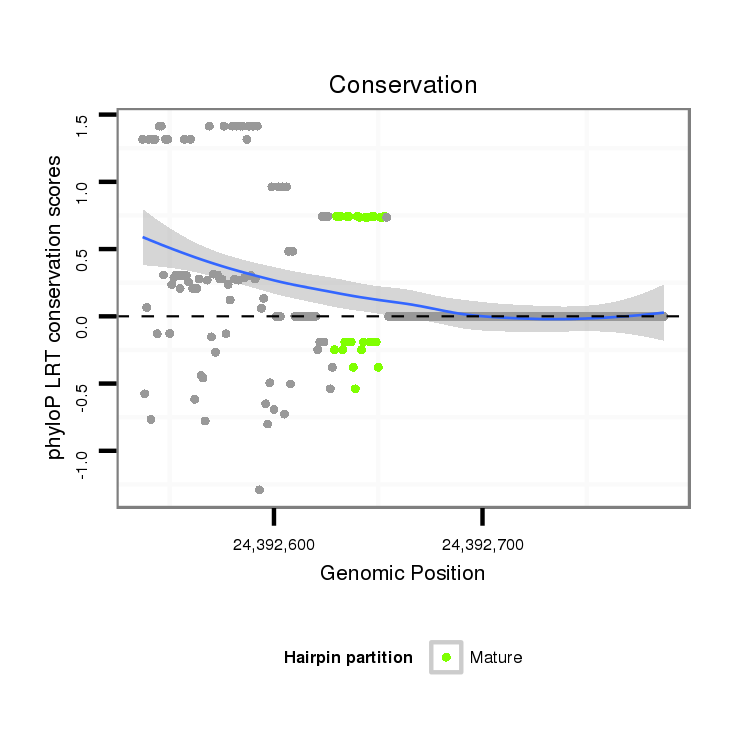
ID:dvi_21253 |
Coordinate:scaffold_13049:24392587-24392737 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -21.7 | -21.5 | -21.4 |
 |
 |
 |
CDS [Dvir\GJ14104-cds]; exon [dvir_GLEANR_13943:3]; intron [Dvir\GJ14104-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CGAGACCCAAACGCAGATCACAGGCGGCGGCAAATCTGGAGACACAATTCGTAAGTCCAACATCATTTCAATATATATTATTAGAAAAAAGAATTATCTTCGGAGTACCGAAGGGGAGATATTAACTATAGACGAGCCATATCAGATAAGTGTAAAGAGCATTAATTAGTCTCACTTTAAAATCTGTCTTTTGTTTTACTTAACTGATCTTTAATGTAGAAGCAATAATATGCTAAAGATTGTAATTTATA **************************************************(((..(((.......((((((((.....)))).)))).........(((((((....))))))))))..))).......(((((((......((((((((((...(((...........))))))))....)))))...))))))).....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
SRR060657 140_testes_total |
M061 embryo |
SRR060658 140_ovaries_total |
SRR060666 160_males_carcasses_total |
SRR060673 9_ovaries_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR1106714 embryo_2-4h |
SRR1106725 embryo_14-16h |
SRR060687 9_0-2h_embryos_total |
M027 male body |
SRR060682 9x140_0-2h_embryos_total |
SRR060671 9x160_males_carcasses_total |
V053 head |
V116 male body |
SRR060668 160x9_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......CCAAACGCAGATCACAGGCGGCGGC............................................................................................................................................................................................................................ | 25 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................ATCTGGAGACACAATTC......................................................................................................................................................................................................... | 17 | 0 | 1 | 2.00 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............GCAGATCACAGGCGGCGGC............................................................................................................................................................................................................................ | 19 | 0 | 1 | 2.00 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....CCCAAACGCAGATCACAGGCGGC............................................................................................................................................................................................................................... | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................ATATCTGATGAGCGTAAAGAGCA.......................................................................................... | 23 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................ATTATCTTCGGAGTACCGAAGGGGA...................................................................................................................................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...GACCCAAACGCAGATCACAGGCGGC............................................................................................................................................................................................................................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............GCAGATCACAGGCGGCGGCA........................................................................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................GATTATCGAAGGGGAGAT................................................................................................................................... | 18 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................TATCTGATGAGCGTAAAGAG............................................................................................ | 20 | 3 | 15 | 0.13 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 |
| ....................................................................................................................................................................................................................................TATGCTAAAGATTGGACT..... | 18 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................TATATTATTTGAGAAAAGAA.............................................................................................................................................................. | 20 | 2 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................GTCCAACATCGTATCACTA.................................................................................................................................................................................. | 19 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................GGGGGCGGCAAACCTGGTGA................................................................................................................................................................................................................. | 20 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...................................................................................................................................................................................AACTCTGTCTATGGTTTTAC.................................................... | 20 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GCTCTGGGTTTGCGTCTAGTGTCCGCCGCCGTTTAGACCTCTGTGTTAAGCATTCAGGTTGTAGTAAAGTTATATATAATAATCTTTTTTCTTAATAGAAGCCTCATGGCTTCCCCTCTATAATTGATATCTGCTCGGTATAGTCTATTCACATTTCTCGTAATTAATCAGAGTGAAATTTTAGACAGAAAACAAAATGAATTGACTAGAAATTACATCTTCGTTATTATACGATTTCTAACATTAAATAT
**************************************************(((..(((.......((((((((.....)))).)))).........(((((((....))))))))))..))).......(((((((......((((((((((...(((...........))))))))....)))))...))))))).....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060682 9x140_0-2h_embryos_total |
V116 male body |
V047 embryo |
SRR060661 160x9_0-2h_embryos_total |
SRR060679 140x9_testes_total |
SRR060663 160_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060655 9x160_testes_total |
SRR060681 Argx9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ........................................CTGTGCTAAGCATTTAGGTGG.............................................................................................................................................................................................. | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................TGTGCTAAGCATTTAGGTGG.............................................................................................................................................................................................. | 20 | 3 | 7 | 0.43 | 3 | 1 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ........................................CTGTGCTAAGCATTTAGGTG............................................................................................................................................................................................... | 20 | 3 | 9 | 0.33 | 3 | 0 | 2 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........................................CTGTGCTAAGCATTCGGG................................................................................................................................................................................................. | 18 | 2 | 6 | 0.33 | 2 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................CTGTGCTAAGCATTCGGGTG............................................................................................................................................................................................... | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................TGTGTTAAGAACTCAGGT................................................................................................................................................................................................ | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .CTCTGGACTTGAGTCTAGTG...................................................................................................................................................................................................................................... | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .......................................ACTGTGCTAAGCATTTAGGT................................................................................................................................................................................................ | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ............................................................................................................................................TGGTCTATGCACATTTCTA............................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:24392537-24392787 + | dvi_21253 | CGAGACCCAAACGCAGATCACAGGCGGCGGCAAATCTGGAGACACAATTCGTAAGTCCAACATCATTTCAATATATATTATTAGAAAAAAGAATTATCTTCGGAGTACCGAAGGGGAGATATTAACTATAGACGAGCCATATCAGATAAGTGTAAAGAGCATTAATTAGTCTCACTTTAAAATCTGTCTTTTGTTTTACTTAACTGATCTTTAATGTAGAAGCAATAATATGCTAAAGATTGTAATTTATA |
| droMoj3 | scaffold_6680:23813340-23813412 - | CGAGGCCCAAACGCAAATCACAGGCAGCAGAAAAAATGGAGACACAATTTGTAAGTGAACATTTATTTAAACA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_15110:24284566-24284625 + | CACGACCCAAACGCAGATCACAGGCGGCAACAAGTCTGGAGACACAATTTGTAAGTACAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004762:2793771-2793826 + | CACGGCCTAAACGAAGATCACAAGCTGCACAGAATCTGGAAACACAGTTTGTAAGT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000302428:5955040-5955143 - | CCCGCCCAAAGCGTCGGAATCTGGATTTCGTAAGTACAAATCGATAATTTGTTAATCCTTTTTATTATAA--------------TGAGAACCTTTAATTTTTCAGAGCCAAAATGGAG------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
Generated: 05/16/2015 at 10:50 PM