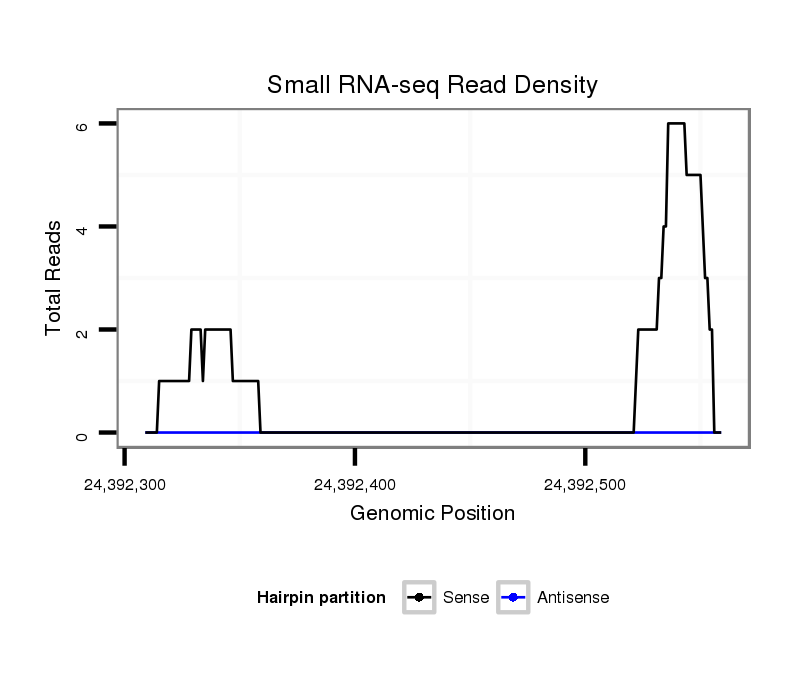
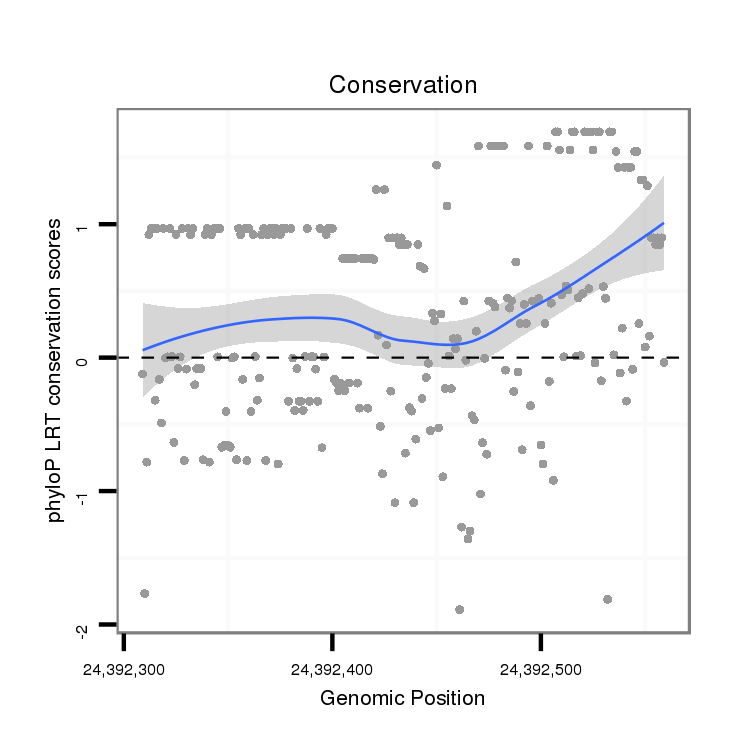
ID:dvi_21252 |
Coordinate:scaffold_13049:24392359-24392509 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ14104-cds]; exon [dvir_GLEANR_13943:3]; intron [Dvir\GJ14104-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CGGGAAGTCGATGATACGCTCTTGTGCTCCGAGGATGAAATGTTGCTGGTGATCAGTGACGATGTGCTTTAAATCTTTTATACAAATGCATAGTTGTTAAAAAAATTACTACACTCACATACACACCCAGCACAACCATTTTTTTTTCTTATGAATAAGTCATTTAAAACTAATATATATGTATATATATTATATGTCAAGCATACAAACTTAATTCGAAGACGTTTACGAGACCCAAACGCAGATCACAG **************************************************................(((.........((((...(((((((..(((((...((((.(((....(((..................................)))...))).)))).....)))))..))))))).)))).........)))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
V053 head |
M027 male body |
SRR060663 160_0-2h_embryos_total |
SRR060664 9_males_carcasses_total |
SRR060679 140x9_testes_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
V047 embryo |
SRR060669 160x9_females_carcasses_total |
SRR060674 9x140_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...................................................................................................................................................................................................................................ACGAGACCCAAACGCAGATC.... | 20 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................................TTACGAGACCCAAACGCAGA...... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................TTCGAAGACGTTTACGAGACCCAAACGC......... | 28 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......GTCGATGATACGCTCTTGT.................................................................................................................................................................................................................................. | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| CGGGAAATCGATGATGTGCTCT..................................................................................................................................................................................................................................... | 22 | 3 | 2 | 1.00 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................CTCCGAGGATGAAATGTTGCTGGT......................................................................................................................................................................................................... | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................GTTTACGAGACCCAAACGCA........ | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................CTTGTGCTCCGAGGATGA..................................................................................................................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................ATTCGAAGACGTTTACGAGACC................ | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| CGGGACTTCGATGAAACGCTCT..................................................................................................................................................................................................................................... | 22 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................ACCCAGCACTACCATTCT............................................................................................................. | 18 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ............................................................................................................................ACCCAGCACTACCATGTTTG........................................................................................................... | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| CGGGAAATCGATGATGTGCTC...................................................................................................................................................................................................................................... | 21 | 3 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................ACGATGTGCTTCCAATCCT.............................................................................................................................................................................. | 19 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ....................................................................TTAAATCATTTATACAACTGCT................................................................................................................................................................. | 22 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..........ATGGTACGCTTTGGTGCTC.............................................................................................................................................................................................................................. | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................................TGCATAGATCTTAAAAAAA.................................................................................................................................................. | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GCCCTTCAGCTACTATGCGAGAACACGAGGCTCCTACTTTACAACGACCACTAGTCACTGCTACACGAAATTTAGAAAATATGTTTACGTATCAACAATTTTTTTAATGATGTGAGTGTATGTGTGGGTCGTGTTGGTAAAAAAAAAGAATACTTATTCAGTAAATTTTGATTATATATACATATATATAATATACAGTTCGTATGTTTGAATTAAGCTTCTGCAAATGCTCTGGGTTTGCGTCTAGTGTC
**************************************************................(((.........((((...(((((((..(((((...((((.(((....(((..................................)))...))).)))).....)))))..))))))).)))).........)))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060668 160x9_males_carcasses_total |
V047 embryo |
SRR060666 160_males_carcasses_total |
SRR060679 140x9_testes_total |
SRR060684 140x9_0-2h_embryos_total |
V053 head |
SRR060655 9x160_testes_total |
SRR060678 9x140_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..............................................................................................................................................................CCGAAAATTTTGATTATATA......................................................................... | 20 | 2 | 7 | 0.43 | 3 | 0 | 0 | 1 | 1 | 1 | 0 | 0 | 0 |
| ........................................................................................................................................GTGATAAAAGAGAATACTTATT............................................................................................. | 22 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................ACATTTTGATTATATATACAA.................................................................... | 21 | 2 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................TTGTTTTAATGATGAGAGGGT.................................................................................................................................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .....................................................................................................................................................................................................................................CTCTGGACTTGAGTCTAGTG.. | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ............................................................TTCCACGAAATTTAGAATATA.......................................................................................................................................................................... | 21 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:24392309-24392559 + | dvi_21252 | CGGGAAGTCGATGATACGCTCTTGTGCTCCGAGGATGAAATGTTGCTGGTGATCAGTGACGATGTGCT------------------TTAAATCTTTTATAC----------AAATGCATAGTTGTTAAAAAAATTACTACACTCACATACACACCCAGCACAACCATTTTTT--------------------TTT------------------------CTTATGAATAA-GTCATTT--AAAACTAATATATAT----GTATA------TATATTATAT-GTCAAGCATACAAACTTAATTCGAAGACGTTTACGAGACCCAAACGCAGATCACAG |
| droMoj3 | scaffold_6680:23813390-23813531 - | CC------------------------------------------------------------------------------------------------------------------------------------------ACAGACACAGACACACTAGCC---TAATTTTT--------------------ATT------------------------TTTGTTGATGT-ATCAATT--AAAACTAATTTATGTATTTATATA------TGTATTTGAT-ATAAAGCATACAAGCATAATTCAAAAACATTCACGAGGCCCAAACGCAAATCACAG | |
| droGri2 | scaffold_15110:24296539-24296781 + | CACGAAATTTATGATCCGCTGTTGTGCTCAGACGATGATGAAATGATGATCAACAACGAGGATGTACTACTAGAAACCGACGAACTTTGAAACCCATACAC----------AGAAGCATAA----------------------------------------------TTTATTGACTGTACAATCAATTTACAATTACACTCATACAACTAACAAAATGTATATCAATTC-ACAAAAT--GAAACTAATAAATAT----GTGTA------TGTATCCTAT-ATTAAGTAAACAAGCTTAATTCAAAGGATTTT------------------------ | |
| droWil2 | scf2_1100000004762:2793651-2793793 + | AC------------------------------------------------------------------------------------------------------------------------------------------ACACACACATACACACAATACATCCTCTCTGT--------------------T-------------------------------GCTTGA-TAAACAAAATGAATTAATTTCC---TACAATTTTCAACTTTATATACTT-ATGAAGCGTACAAGCTTAACTCAAAGACATTTACACGGCCTAAACGAAGATCACAA | |
| droEle1 | scf7180000491255:2410052-2410094 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAGCCTTCAAACACAATTCGAAGACCTTCACCCGCCCCAAGCG---------- | |
| droBia1 | scf7180000302428:5955134-5955409 - | CAGGAAATCGAAAACACTTTGGTGTCAGATGAGGATAACTTTGAATTGGTCATCGACGAGGATGTCCTAATAGGAGGCCATGATTTTTAATAATTACATGTGAAAAAAAACCAACACATAGCAATAAGAAAGCTTAATACATTTAA---------------------------------ATAGTTAAGACATCATTATATTCGT-----TAGTCGATAGTACACCGAGTGCGGAACTCAAGAATCTAATTTAT---TTTAAACT------TTTATTTTTT-CAAAAGCCTTCAAACACAATTCGAAAACGTTTACCCGCCCAAA------------- | |
| droEug1 | scf7180000409456:146133-146237 + | TT--------------------------------------------------------------------------------------------------------------------------------------------------------------------CCTTAA--------------------T-------------------------------GTAAAA-GGAATCCAAAAATCTAATTTAT---TTTAATCT------TTTATTTCTTACAAAAGCCTTCAAAAACAATTCGAAAACCTTTACCCGCCCCAAGCGCAG------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
Generated: 05/16/2015 at 10:50 PM