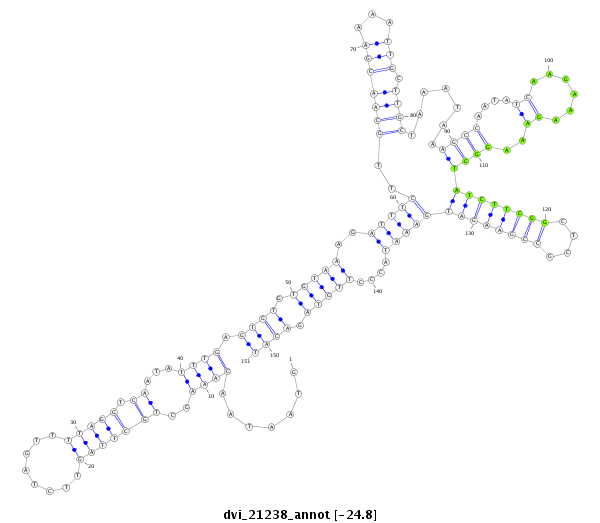
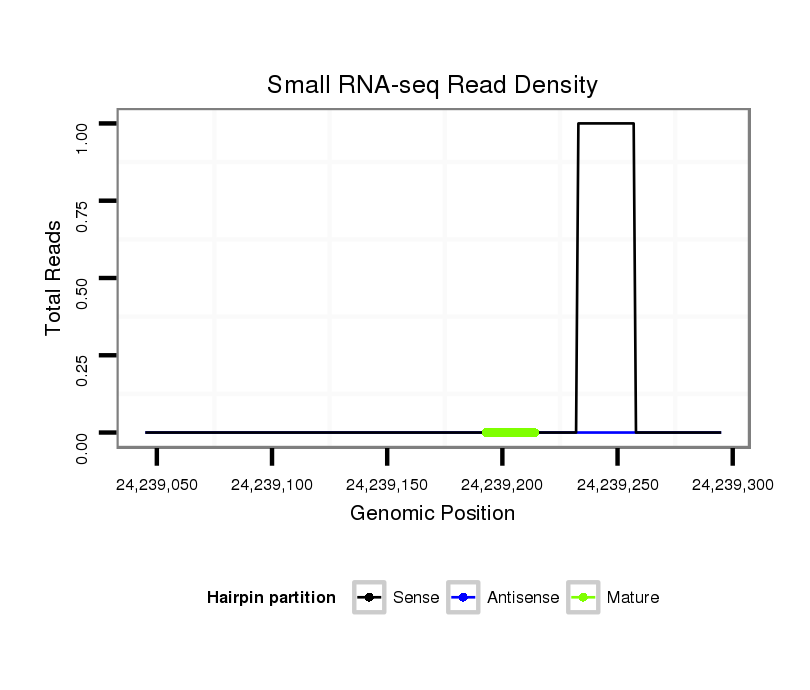
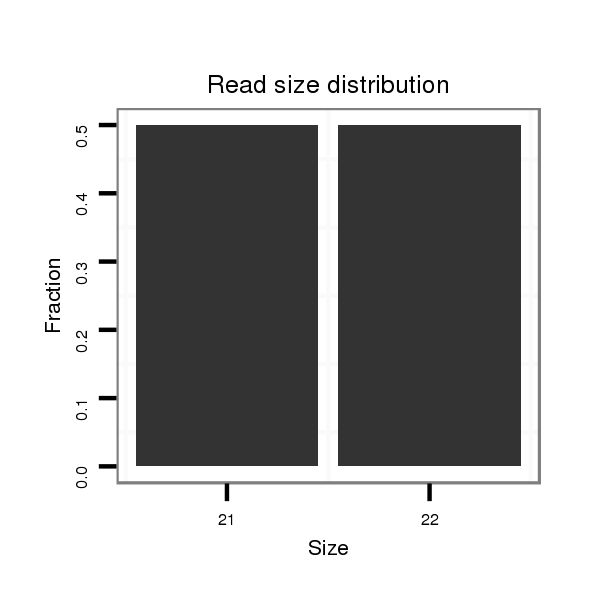
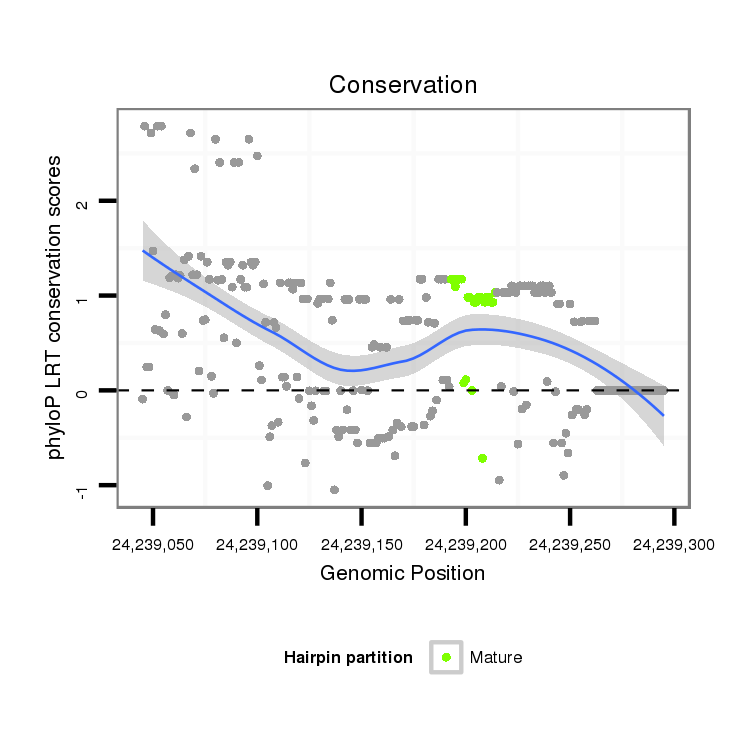
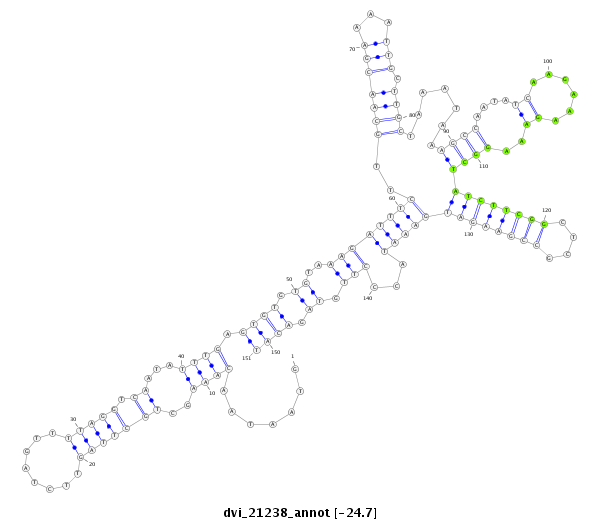
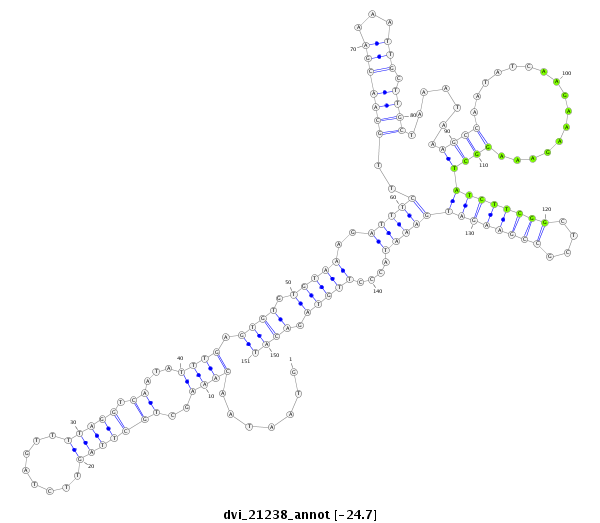
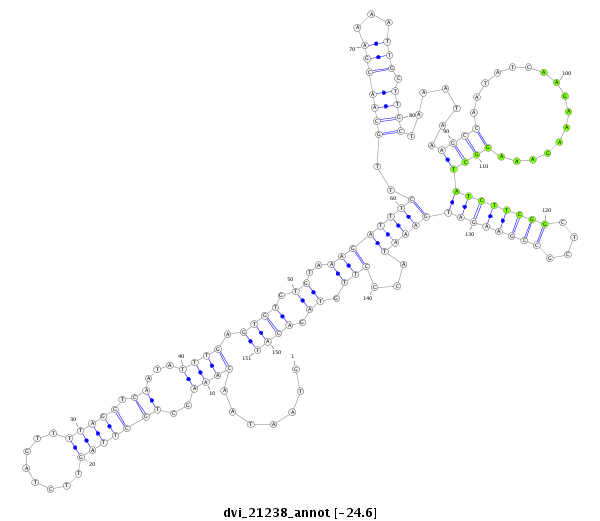
ID:dvi_21238 |
Coordinate:scaffold_13049:24239095-24239245 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -24.7 | -24.7 | -24.6 |
 |
 |
 |
CDS [Dvir\GJ14094-cds]; exon [dvir_GLEANR_13934:2]; intron [Dvir\GJ14094-in]
| Name | Class | Family | Strand |
| Helitron-2_DVir | RC | Helitron | - |
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CTCCATGACACAGGCGGCGCAGTTGGCCAAAACTCTGCGTTATGCGCCAGGTAATAACAAAGCTGCTTAGTTCTAGTTTTAGGTCAATATTTGAGTGTGTGTAAAGATTTCTTGCAACGAAAATTGCTTGCTAAATAAAGCCAATATCAAGAAAGAAAGGCTATCTTCGGCTCGCCGAAGATGAAATACCCTTGTAGACATACACAACAGCTTTATTTTATCACGGGGCGACTAAAAGACATTCTTTCATG **************************************************.......((((..(((((((........))))).))...)))).((((.(((((..(((((..(((((((...))).)))).......((((....((......))..))))((((((((....)))))))))))))....))))).))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060668 160x9_males_carcasses_total |
V116 male body |
SRR060663 160_0-2h_embryos_total |
SRR060669 160x9_females_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
GSM1528803 follicle cells |
SRR060666 160_males_carcasses_total |
SRR060658 140_ovaries_total |
M027 male body |
SRR060673 9_ovaries_total |
SRR060678 9x140_testes_total |
SRR060681 Argx9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................................................................GATTTCTTGGAAAGAAGATTGC............................................................................................................................ | 22 | 3 | 1 | 2.00 | 2 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................CCCTTGTAGACATACACAACAGCTT...................................... | 25 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................AGCCAAGCTCAAGAAAGAA.............................................................................................. | 19 | 2 | 4 | 0.50 | 2 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................AAGAGTTCCTGTAACGAAAA................................................................................................................................ | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........AGGTGGCGAAGTCGGCCAAAA........................................................................................................................................................................................................................... | 21 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................TAGGTCCAGCTTTAGGTCAA.................................................................................................................................................................... | 20 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................GCCAAGCTCAAGAAAGAA.............................................................................................. | 18 | 2 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................CGCCGAAGCTGACATACCCTT.......................................................... | 21 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ....................................................................................................................................................AAGAAAGAAAGGCTATCTTCGG................................................................................. | 22 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................................................................................................................................ATCAAGAACGAAAGGGGAT....................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................................................................................................................................................AGAAAGAAAGGCTATCTTCGG................................................................................. | 21 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
|
GAGGTACTGTGTCCGCCGCGTCAACCGGTTTTGAGACGCAATACGCGGTCCATTATTGTTTCGACGAATCAAGATCAAAATCCAGTTATAAACTCACACACATTTCTAAAGAACGTTGCTTTTAACGAACGATTTATTTCGGTTATAGTTCTTTCTTTCCGATAGAAGCCGAGCGGCTTCTACTTTATGGGAACATCTGTATGTGTTGTCGAAATAAAATAGTGCCCCGCTGATTTTCTGTAAGAAAGTAC
**************************************************.......((((..(((((((........))))).))...)))).((((.(((((..(((((..(((((((...))).)))).......((((....((......))..))))((((((((....)))))))))))))....))))).))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
V053 head |
SRR060677 Argx9_ovaries_total |
M028 head |
V116 male body |
SRR060686 Argx9_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................................................................................................................................TTATTTCGATTTTAGTTGTTTCT............................................................................................... | 23 | 3 | 7 | 1.14 | 8 | 8 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................TATCTCGGTTTTAGTGCTTTCT............................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................GGCTTCTACTTAATGGGAACATCT..................................................... | 24 | 1 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................GTATTTCGATTTTAGTTCTTTCT............................................................................................... | 23 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................CATTTCTTAAGATCGTTG..................................................................................................................................... | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| GAAGTAGTGTGTGCGCCGC........................................................................................................................................................................................................................................ | 19 | 3 | 18 | 0.17 | 3 | 0 | 0 | 0 | 0 | 2 | 1 | 0 |
| ................................................................................................................AAGTTGCATTTAACGAAC......................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................................................................................................CTTTCTTTCCGATAGAAGCC................................................................................. | 20 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:24239045-24239295 + | dvi_21238 | C--TC-CATGACACAGGCGGCGCAGTTGGCCAAAACTCTGCGTTATGCGCCAGGTAATAAC---AAAGCTGCTTAGTTCTA----GTTTTAGGTCAATATTTGAGTG------------------TGTGTA----AAGATTTCTTGCAAC---------GAAAATTGCTTGCTAAATAAAGCC-AATATCAAGAAAGAAAGGCTATCTTCGGCTCGCCGAAGATGAAATACCCTTGTAGACATAC-ACAACAGCTTTATTTTATCACGGGGCGACTAAAAGACATTCTTTCATG |
| droMoj3 | scaffold_6680:18864891-18865054 + | C--TC-CATGACACAGGCAGCGCAATTGGCCAAAACGCTGCGCTATGCCCCAGGTAATACCAACAACGTTGTTTAGTTCTACATG-T-------------T-CTTTAGCTACCAACTTTTGTTTATGTATTTTCGTAGATTACTAACGGCCAAGTTTTGTATTGT---------AA------C-AATATAAAGAAAAA------------------------------------------------------------------------------------------------ | |
| droGri2 | scaffold_15110:9505982-9506076 - | C--TC-CATGACACAGGCGGCGCAGTTGGCCAAAACGCTACGTTATGCGCCAGGTAATAAC---AATCTTGCTTAGTTCTACATAGTTGTAAATCAATATT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004511:1062108-1062163 + | G--TC-CATGACACAAGCGGCACAATTGGCTAAAACACTGCGCTATGCGCCAGGTAATA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | XR_group6:4516722-4516776 + | C--TC-CATGACACAAGCGGCGCAATTGGCCAAAACGTTGCGCTATGCGCCAGGTAAT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_9:2811216-2811270 + | C--TC-CATGACACAAGCGGCGCAATTGGCCAAAACGTTGCGCTATGCGCCAGGTAAT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_13337:4010023-4010079 - | C--TC-CATGACACAAGCGGCGCAATTGGCCAAAACGTTGCGCTATGCGCCAGGTAATAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droBip1 | scf7180000395450:144796-144851 - | C--TC-CATGACACAAGCGGCGCAATTGGCCAAAACGTTGCGCTATGCGCCAGGTAATA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000298561:2172-2291 - | G--TTGTACAACAATGTCCGCGTCCTTG-TAAGGGTTTTGCACCCACCACTCCCTGCCA-------------------------------------------------------------------------------------------------------------------AAAAATACAAAAAAACAAGAAAGGAA-GCTAACTTCGGCTAGCCGAAGTTGAAATACCCTTGCAG------------------------------------------------------- | |
| droFic1 | scf7180000453831:528297-528352 - | C--TC-CATGACACAAGCGGCGCAATTGGCCAAAACGTTGCGCTATGCGCCAGGTAATA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491199:1202397-1202455 + | C--TC-CATGACACAAGCGGCGCAATTGGCCAAAACGCTGCGCTATGCGCCAGGTAATAAT---A------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000774017:33541-33623 + | TACTA-AATTAAACAGGAAAGGAAGCTAGCTTCG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCAAGCCGACGTTTATATACCCTTGCAGTTATTACAAATCGACTACATTT--------------------------------- | |
| droBia1 | scf7180000302377:1618203-1618258 + | C--TC-CATGACACAAGCAGCGCAATTGGCCAAAACGCTGCGCTATGCGCCAGGTAATA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415363:161360-161504 + | A--TA-A-TGAAAGGG-------------------------------------------------------TTCGTTTCTG----TTTATTGAACAAATTTTAAGAA------------------TCTAAA-----AAATA-----------ATTTATAGAAGATGGCTTTATAAATA-----AAAAAACAAGAAAGGAA-GCTACCTTCGGCCAGCCGAAGCTTATATACCCTTGCAGATATAG-GCA--------------------------------------------- | |
| droEug1 | scf7180000409472:515861-515916 - | C--TC-CATGACACAAGCGGCGCAATTGGCCAAAACGTTGCGCTATGCGCCAGGTAATA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dm3 | chr3L:21687410-21687465 - | C--TC-CATGACACAAGCGGCGCAGTTGGCCAAAACGCTGCGCTATGCGCCAGGTAATA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | 3l:21205204-21205259 - | C--TC-CATGACACAAGCGGCGCAGTTGGCCAAAACGCTGCGCTATGCGCCAGGTAATA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_11:1608010-1608065 - | C--TC-CATGACACAAGCGGCGCAGTTGGCCAAAACGCTGCGCTATGCGCCAGGTAATA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | 3L:18824100-18824155 + | C--TC-CATGACACAAGCGGCGCAGTTGGCCAAAACGCTGCGCTATGCGCCAGGTAATA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4784:21344652-21344707 - | C--TC-CATGACACAAGCGGCGCAGTTGGCCAAAACGCTGCGCTATGCGCCAGGTAATA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 10:23 PM