
ID:dvi_21029 |
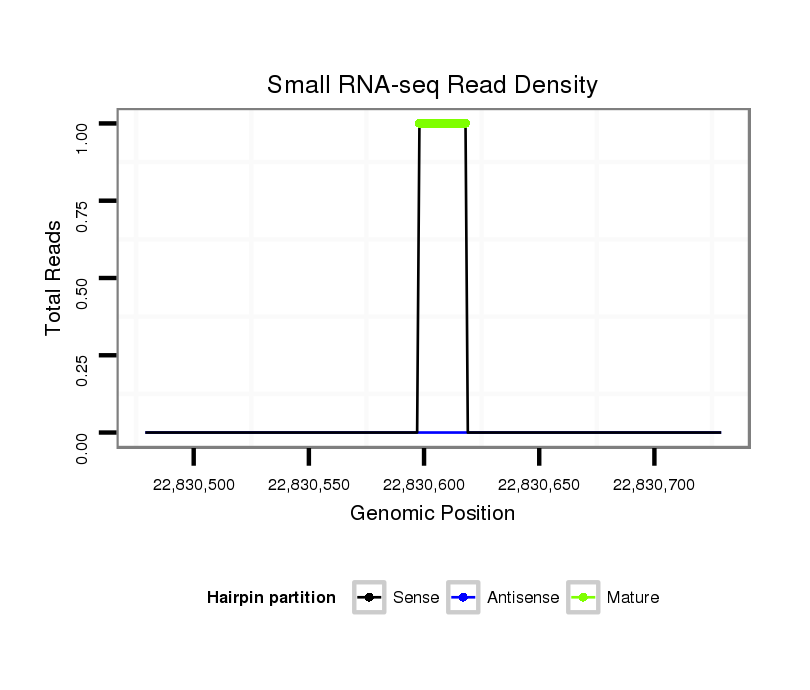
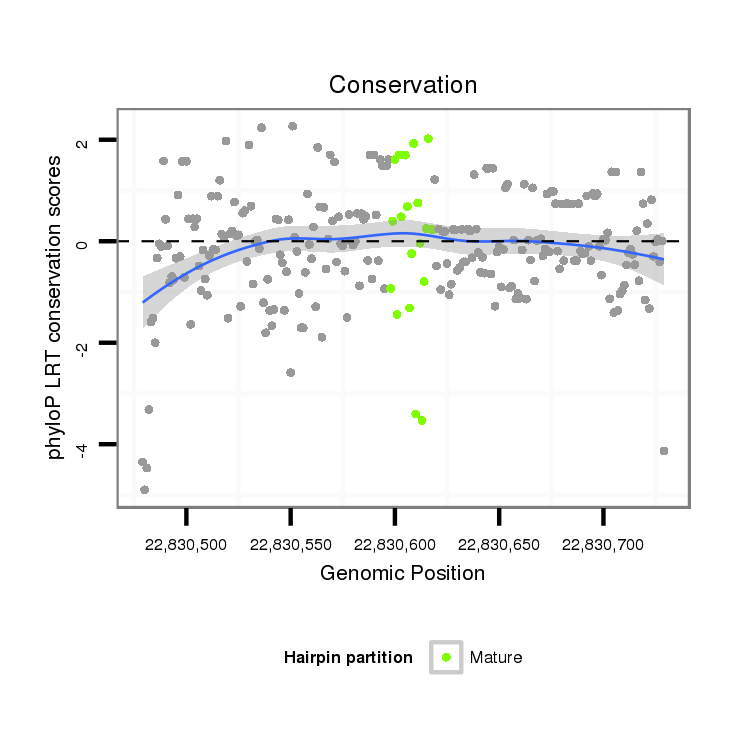
Coordinate:scaffold_13049:22830529-22830679 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -17.1 | -17.0 | -17.0 |
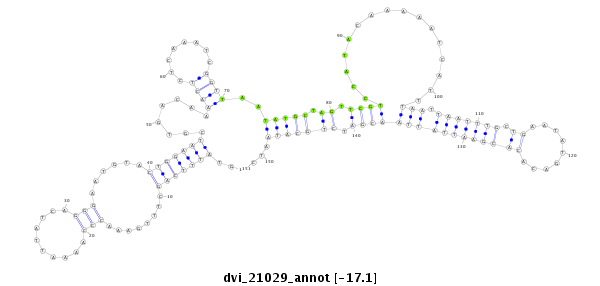 |
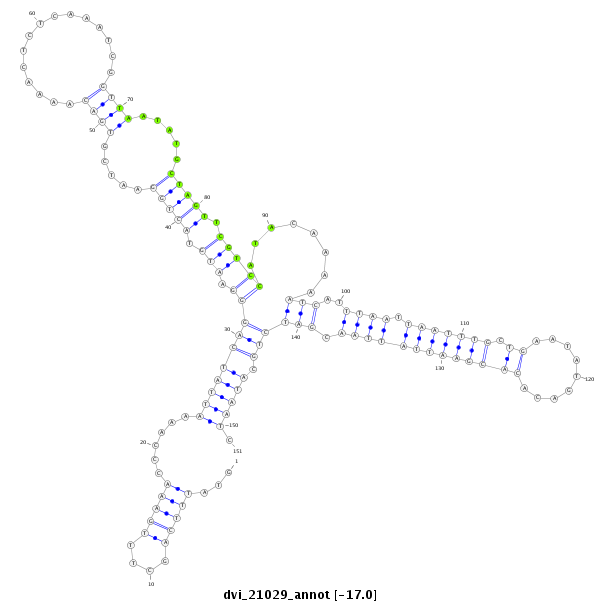 |
 |
exon [dvir_GLEANR_13843:4]; CDS [Dvir\GJ13995-cds]; intron [Dvir\GJ13995-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- GCCTGGTGGTATACCAAGGATTTGTCTGGCAGCAACAATTTGAATGGAAAGTATTTCAGCTTTGAAACCCAAAATTATCAGGGAATGTACTGGAATCGTGACAAAACTCTCAAATCGGTTAATATGCTAGTTCGTCCATACAAAAATCATTTAATTAATTTGCTGAATATGACACACGAATTATTAACGATCTGCATAATCCCATCCGATTACGAAAGATCATGCATTATCAAGATCATAAAATAATAAAG **************************************************..(((((((........(((..........)))......)))))))........((((........))))..(((((.((.((((................((((.((((((.((.........)))))))))))))))).)))))))...************************************************** |
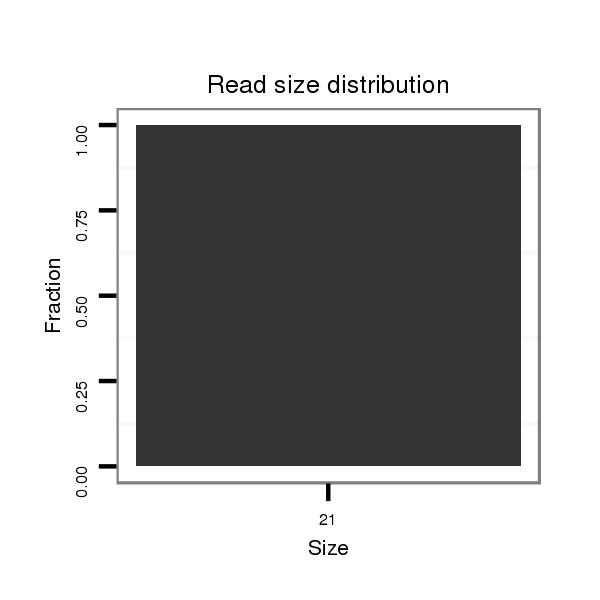
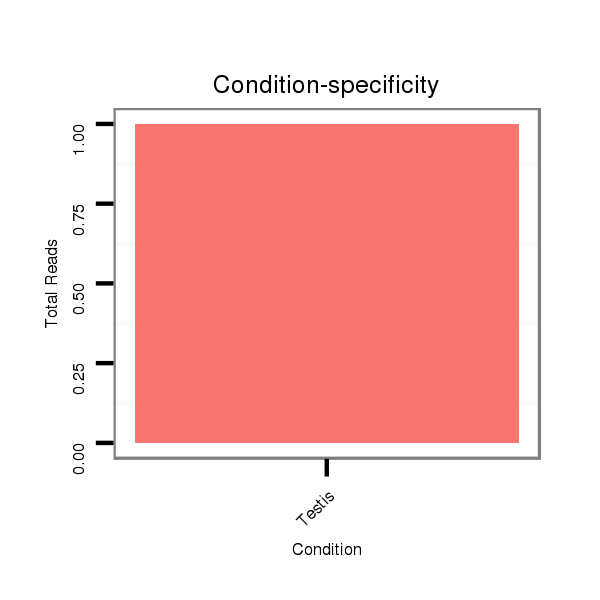
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060655 9x160_testes_total |
SRR060689 160x9_testes_total |
SRR060671 9x160_males_carcasses_total |
M047 female body |
SRR060664 9_males_carcasses_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........TGCCGGGGATTTGTCTGGCAG........................................................................................................................................................................................................................... | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................TAATATGCTAGTTCGTCCATA............................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................ACCCAAAATTATCCCGGAA...................................................................................................................................................................... | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| GCCTGGGGGTGTACCGAG......................................................................................................................................................................................................................................... | 18 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........................................................................................................................................................................................CACCGATGTGCATAATCC................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| ......................................................TTCACCTTTGGAAACCAAAA................................................................................................................................................................................. | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
|
CGGACCACCATATGGTTCCTAAACAGACCGTCGTTGTTAAACTTACCTTTCATAAAGTCGAAACTTTGGGTTTTAATAGTCCCTTACATGACCTTAGCACTGTTTTGAGAGTTTAGCCAATTATACGATCAAGCAGGTATGTTTTTAGTAAATTAATTAAACGACTTATACTGTGTGCTTAATAATTGCTAGACGTATTAGGGTAGGCTAATGCTTTCTAGTACGTAATAGTTCTAGTATTTTATTATTTC
**************************************************..(((((((........(((..........)))......)))))))........((((........))))..(((((.((.((((................((((.((((((.((.........)))))))))))))))).)))))))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
SRR060682 9x140_0-2h_embryos_total |
SRR060665 9_females_carcasses_total |
SRR060687 9_0-2h_embryos_total |
SRR060669 160x9_females_carcasses_total |
SRR060677 Argx9_ovaries_total |
SRR1106722 embryo_10-12h |
SRR060684 140x9_0-2h_embryos_total |
M061 embryo |
SRR1106720 embryo_8-10h |
SRR1106729 mixed whole adult body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..................................................................................................................................................................................................................CTGCTTTCTAGTACCTAA....................... | 18 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................AAACTGTGGGTTTTAATA............................................................................................................................................................................. | 18 | 1 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................AATTGCAGGACCTATTAGGG................................................ | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................CTTGGGGTTGTAATGGTCCC........................................................................................................................................................................ | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................AAATTAATTAAACGGCTTGTA................................................................................. | 21 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................TAATGAATTATACGACTTATA................................................................................. | 21 | 3 | 15 | 0.13 | 2 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................................................................................................AATTAATTAAGCGACTTGT.................................................................................. | 19 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................CGAAATAGTTCTAGT............. | 15 | 1 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 |
| ................................................................................CCCTTACACCACCTTAG.......................................................................................................................................................... | 17 | 2 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...............................................................................................AGCACTATTTTGAGGGTTG......................................................................................................................................... | 19 | 3 | 19 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........CATATGTTTACTAAACAG................................................................................................................................................................................................................................. | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:22830479-22830729 + | dvi_21029 | GCCTGGTG----GTATACCAAGGATT----TGTCTG----------------------------------------------------------------------------------------GCAGCAACAATTTGAATGGAAAGTATTTCAGCTTTGAAACCCAAAATTATCAGGGAATGTACTGGAATCGTGACAA---------AACTCTCAAATCGGTTAATATGCTAGTTCGTCCATACAAAAATCATTTAATTAATTTGCTGAATATGACACACGAATTATTAACGATCTGCATAATCCCATC--CGATTACGAAAGATCAT----------------------GCATTATCAAGATCATAAAATAA----TAA-AG |
| droMoj3 | scaffold_6496:15595973-15596091 + | AGCAAT---------------------------------------------------------------------------------------------------------------------------------CTCAATGGAAAATACTTTAAAACCCAAACGGACAGCGAACAGGGAATCTTTTGGGATCAATGGTACAGCTTCAAGTCTTTAAAGTCTGTCCAAATGCAGATACGGCCA------------------------------------------------------------------------------------------AA----------------------------------------------G-AA | |
| droGri2 | scaffold_15245:13399276-13399523 + | GCTTGGTG----GTATG------ATT----TTTGTTCCCG-------CAGGTAGGTCAACGGTTTATCTAACTGGTAATATCATA-------------------TATTTAATATTCATTCCACTTTTCTAGTAATCTCAATGGCAAATACTTTAAAAGACAAACGGATAATGCCGAGGGCATTTTTTGGGATCGCTGGTACAGCTTCAAATCTTTGAAGTCTGTCCAGATGCTTATACGGCCA------------------------------------------------------------------------------------------AAGAGC--GCCAAGTTTAATTAATCCATGAAAAGGA--ACTA--T-CAATTA | |
| droWil2 | scf2_1100000004521:10489384-10489504 - | AATC------------------------------------------------------------------------------------------------------------------------------------TAAACGGACGATATTATACAAGTGAAATCGAAAAAAGGGATAGTATGAGCTGGAATGGCTGGTT---------TTCTCTGAAATCAGTACAAATGCTCATTCGACCA------------------------------------------------------------------------------------------AAAAACTCTTCA----------------------------AA----TCA-AT | |
| dp5 | 3:5372258-5372468 + | GCCTGGTG----GTACG------ACT----TCTGTTCGCG-------AAGGTAATTCATCCGTCCACTGA-------GTATCACACAAACACATATGAATTATTCAGTTAATA----TCCTCTTCCCATAGCAATTTGAATGGCAAGTACTTCAAGGCCGAGGTGGACAATGTCCAGGGCATTTTCTGGGAGCAATGGTATAGCTTCAGATCTCTGAGATCCGTACAGATGCTAATCCGTCCA---------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_9058:597-655 + | TTAATGCG----CAAC-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTGCGCACATACGACGTCCATTTGATTAATTTGATAAAAAGGA--------------------------------------------------------------------------------------------------------T-AC | |
| droAna3 | scaffold_13266:1506438-1506528 + | GCGTGGTG----GTATG------ATT----TTTGTTCAAG-------AAGGTAGGG------------------GGACTATAGTT-------------------TA-----------------------------TTACTTTAT------AGTACTTTTTAAGCCAAGAAATATCAGAA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAT-AT | |
| droBip1 | scf7180000395801:49655-49692 + | CAAAAAAT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAG----------------------GGACTGTAACGGTTATA-ATTAA----AAA-AG | |
| droKik1 | scf7180000302199:22225-22322 - | TCAGGTAC----------------------------------------------------------------------------------------------------------------------------------------------GTTAGTAT------------TA----------------------------------------------------------GCTAATTCAATTATGCAATAATGTTCTCAAAACCTTAAGGAATAAAATACATTTAACATTTGCAATTTACATA----------------------------------------------------------------------AT----CGC-AT | |
| droFic1 | scf7180000453180:25493-25584 - | AATTC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AATAAAAATAACGCAAGACTTTTGAACCCTC----------------------TGCAAGAGTATGAAAAAAAC--TCCAAGTCA-AGGAGTAAACAAGAAATA--AAAT----TAA-AG | |
| droEle1 | scf7180000490289:2783-2795 + | TAAAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAA----TAA-AC | |
| droRho1 | scf7180000778271:6295-6424 + | TTAAAACCTTCAAAATAATCAGAAATGCATTATGTTGTCGGATGAAACAAGCATGTCA-----------------------------------------------------------------------------------------------------CAAATTAACAATTAAATTGG---------------------------------------------------------ACAATCATACAAAAACCAATAATTTAATTTTTTAAATACAT---------------------------------------------------------------------------------------------GACAA----TAATTA | |
| droBia1 | scf7180000302291:2405148-2405261 - | CAGCA--------------------------------------------------------------------------------------------------------------------------------ACTTGAATGGGAAATACTTCAAGAGCGAGGTTGACAGTCCGCAGGGCATATACTGGGAGCCCTGGTACAGCTTTCGATCCCTGAAGTCCGTGCAGATGCTCATCCGCCC----------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000412980:16823-17018 - | TTTAGCTG------------------------------------------------------------------------------------------------------------------------------------------------------AGTAACGGGTATCTGACAGTGAAAGTACTCGACTTGTTTCAACATCTTTTACTTTTTAAA---------TAGGCTC---AATACAAACTTTAATGCTTCAAAATACTCATCTAAAACTGAGCACAAGGGTTCAGCAAGCAGCGTAACGCAATCAACTCTCTCGCGAGAGACT----------------------GTATTTCCAAAAAAACAACACAA----TAA-AG | |
| droEug1 | scf7180000408816:24013-24074 + | AACAAA---------------------------------------------------------------------------------------------------------------------------------TTATTTTTAAAGTCGATCAGCTTCAAACTTCAAAATTAATAAAAAATAT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AC----TGA-AA | |
| dm3 | chrX:9857729-9857789 - | GCCTGGTG----GTATGGAAGGGAGTGCTTCGAGAGGTGAGCAAA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTACGCATA-----------------------------------------------------------------------------------------------------------------------------------TTAAGTAT-CT | |
| droYak3 | 3L:15067011-15067043 - | TGTAGG--------------------------------------------------------------------------------------------------------------------------------------------GTATTCCAGCTTTGGAACTCGAAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T-TA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/16/2015 at 08:01 PM