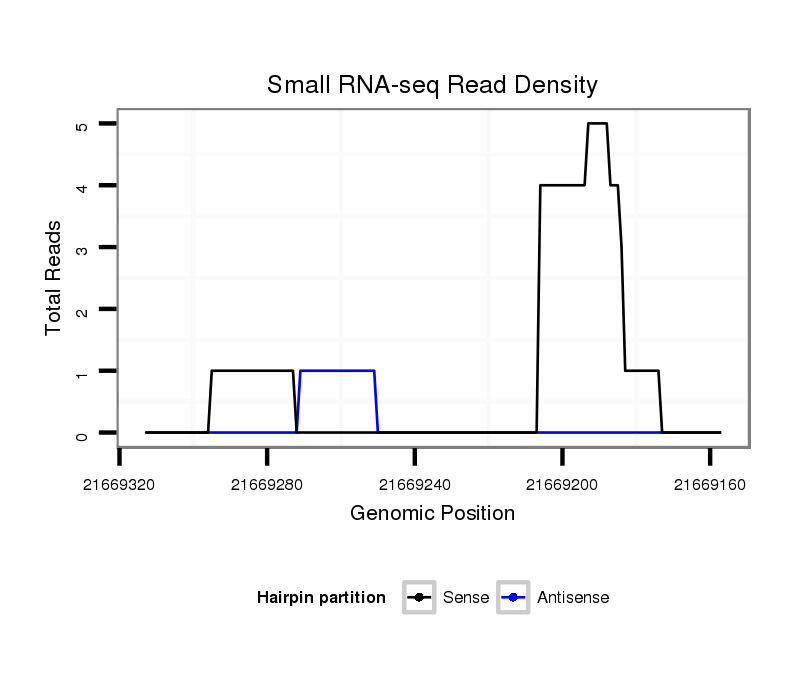
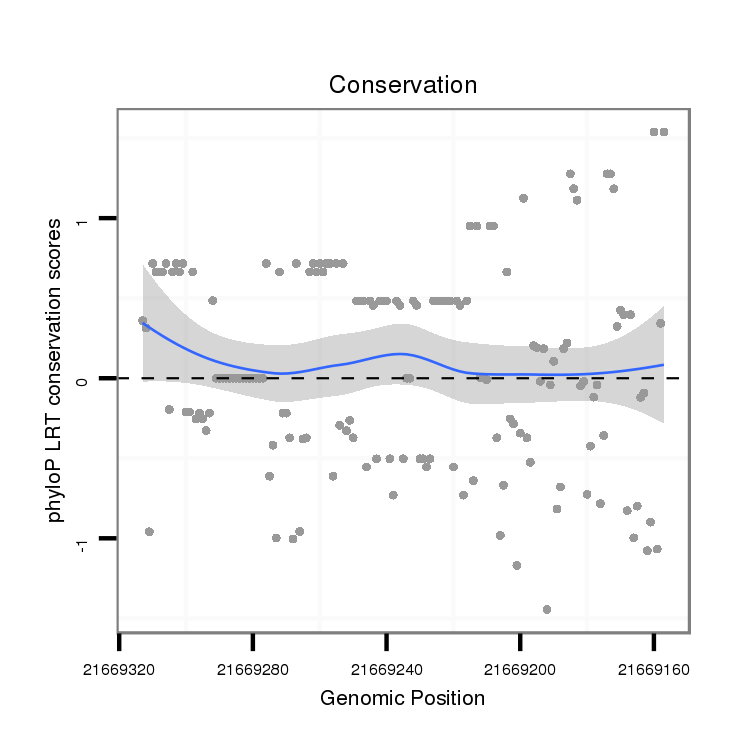
ID:dvi_20894 |
Coordinate:scaffold_13049:21669207-21669263 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_11431:2]; CDS [Dvir\GJ11389-cds]; exon [dvir_GLEANR_11431:1]; CDS [Dvir\GJ11389-cds]; intron [Dvir\GJ11389-in]
No Repeatable elements found
| ##################################################---------------------------------------------------------################################################## TTATGGCAGCAGTTACAGTGTGTGCCACATCGAACAGTGTCGAGGATAACGTGAGTAGAGTCAGTTTAACGATACATGTGTAGCCAATTATATTTGGTTTTTTATAGCCGATTGATTCGAAAGATCAATGTAACGCGTTTTGCTTCAATACGCTGAA **************************************************..............((...............((((((......))))))......))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060667 160_females_carcasses_total |
SRR060660 Argentina_ovaries_total |
SRR060664 9_males_carcasses_total |
SRR060665 9_females_carcasses_total |
SRR060668 160x9_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060683 160_testes_total |
SRR060658 140_ovaries_total |
V053 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................................................................................................CCGATTGATTCGAAAGATCAATG........................... | 23 | 0 | 1 | 2.00 | 2 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................CCGATTGATTCGAAAGATCAAT............................ | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................AGAGTGTGCCACATCGAACAGTGT..................................................................................................................... | 24 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...........................................................................................................CCGATTGATTCGAAAGATC............................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................AAGATCAATGTAACGCGTTT................. | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................TGTGTGCCACATCGAACAGTGTC.................................................................................................................... | 23 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................ACAGTGTCGAGGATAACCCGATT..................................................................................................... | 23 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................CAGTGTCGAAGCTAACGCG........................................................................................................ | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........GCAGTGACCGTGTGGGCCA.................................................................................................................................. | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...............................................................................................................GGGATTCGAAAGAACAATG........................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
AATACCGTCGTCAATGTCACACACGGTGTAGCTTGTCACAGCTCCTATTGCACTCATCTCAGTCAAATTGCTATGTACACATCGGTTAATATAAACCAAAAAATATCGGCTAACTAAGCTTTCTAGTTACATTGCGCAAAACGAAGTTATGCGACTT
**************************************************..............((...............((((((......))))))......))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060681 Argx9_testes_total |
V047 embryo |
M028 head |
V053 head |
|---|---|---|---|---|---|---|---|---|---|
| ..........................................TCCTATTGCACTCATCTCAGT.............................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ....................................................................................................................AGCGTTCTAGCTACATTG....................... | 18 | 2 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 |
| ..................................GTCACAGACGCTATTGCACT....................................................................................................... | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 |
| .......CCGTCAATGGCGCACACGGT.................................................................................................................................. | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 |
| ....................................................................................................................AGCTTTCTAGTTCCATTAT...................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:21669157-21669313 - | dvi_20894 | TTA------TGGCAGC-AGTTACAGTGTGTGCCACATCGAACAGTGTCGAGGATAACGTGAGTAGAGTCAGTTTAACGATACATGTGTAGCC-----------AATTATATTTGGTTTTTTATA--------------------------------------------------------------------------GCCGATTGATTCGAAAGATCAATGTAACGCGTTTTGCTTCAATACGCTGAA |
| droMoj3 | scaffold_6680:16157315-16157477 - | TTACATTATTGGCAGCTAGTTACGACTT----------------TCAAGAAACTGATGTGAGTACAGTATATTTTACAATATCTGC--AGTTGTTATATTTTTTGTTATATATGTTTGTTTATA--------------------------------------------------------------------TAATAAATAGGCCAACTCAACTGAAACATGTAGTTCGGCTTGCCTACATGAGATGAA | |
| droGri2 | scaffold_15110:21911864-21912017 + | TTT------TGGCAAC-AGTCGCAGTGAG---------------TGATGTGATTTCTGTGAGTAGATTCAA---------------------------------------------------------------------ATAAAAATATGGATATAAATCTGTTTTTTATAAATTGATTTGTTTCTTCATTTT-GAAAAAGACGGACGCGGACAATCAATG---CAAATCTTGCTTCAGGGCTCTCAA | |
| droWil2 | scf2_1100000004511:4245123-4245140 + | TTG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTACATGGCAATTCA | |
| droEle1 | scf7180000490982:177975-178051 + | TGGC----------------------------------------------------------------------------------------------------------------TTTCCTTAGCCGCCGCAGATCCAGTCAAA------------------------------------------------------------AATATGGACAATCAGTGTGGCGAACATTGCTTGAATATATTGAA | |
| droTak1 | scf7180000415564:106035-106077 - | ATT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGGAAACGAAGTGTAGGGAATATTGTTTTACGGTCCTAAA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
Generated: 05/16/2015 at 11:07 PM