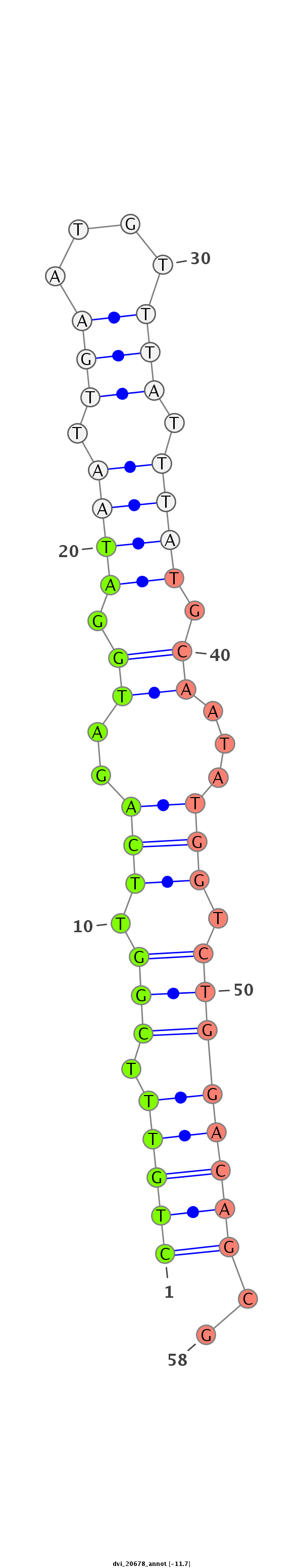
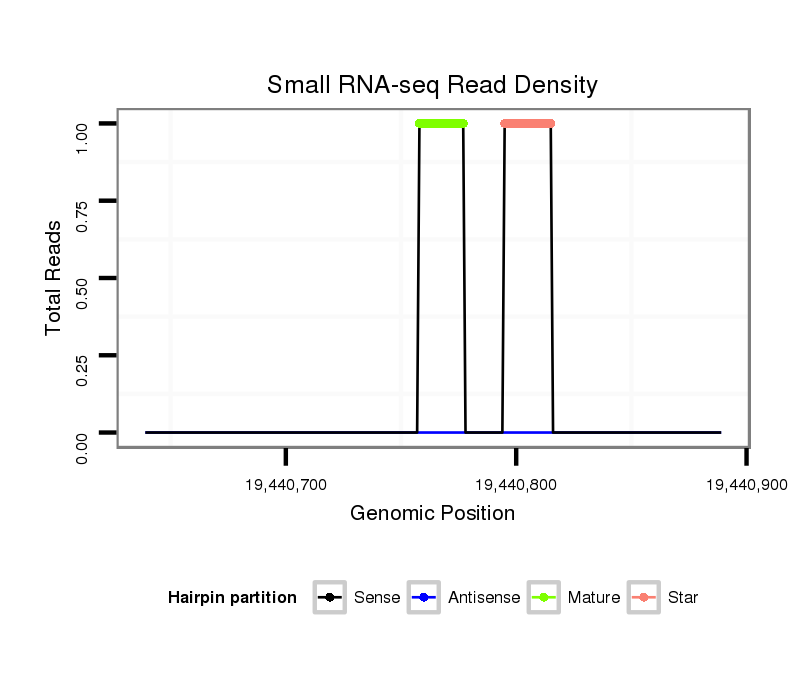

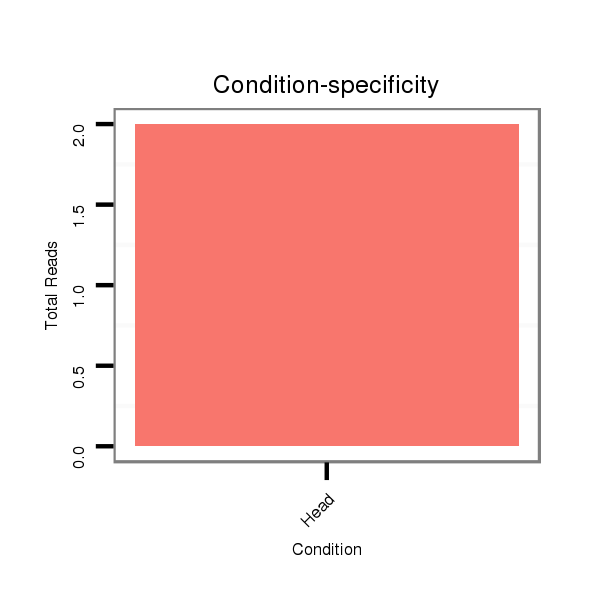
ID:dvi_20678 |
Coordinate:scaffold_13049:19440689-19440839 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
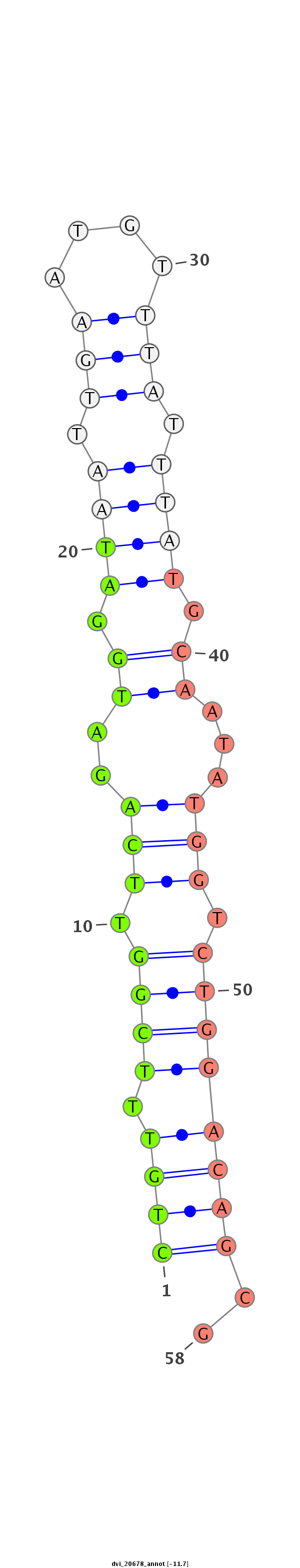
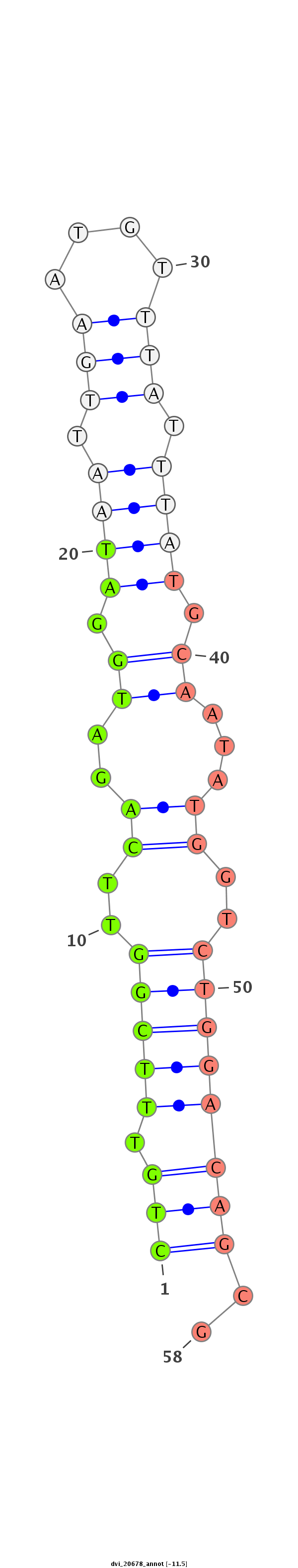
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
No conservation details. |
| -11.7 | -11.7 | -11.5 |
 |
 |
 |
CDS [Dvir\GJ13830-cds]; exon [dvir_GLEANR_13690:1]; intron [Dvir\GJ13830-in]
No Repeatable elements found
| -----------------------------------------------###--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CGCAAGTACACTTGATAACCATTTAGTACTGACTACGTGCACTTAACATGGTGAGCAATCGATTCGATGAGAAAAAAGATAATTTTACAACCAATTCCAGTGCGTGGTTTGAAACCCCGCTGTTTCGGTTCAGATGGATAATTGAATGTTTATTTATGCAATATGGTCTGGACAGCGATATTTTTTGAAATGAATTTTCATGGAGCAATTCCAAAGGTTGAGTTCATACGTGTATAAGTTTGAAACTGATT ***********************************************************************************************************************(((((.(((.(((..((.((((.(((....))).)))).))...))).))))))))..************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M028 head |
SRR060676 9xArg_ovaries_total |
V053 head |
SRR060672 9x160_females_carcasses_total |
M047 female body |
SRR060684 140x9_0-2h_embryos_total |
GSM1528803 follicle cells |
SRR060667 160_females_carcasses_total |
SRR060668 160x9_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................................................CTGTTTCGGTTCAGATGGAT................................................................................................................ | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................TCATGGAACAATTCCAAAGGTTGAGT............................ | 26 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................TGCAATATGGTCTGGACAGCG.......................................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................CAATATGGTCTGCACCGTGAT........................................................................ | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................................CATAGGTGGATAAGTTTGCAA...... | 21 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................................................................................ACCAATTCCAGTTGGTGGGT.............................................................................................................................................. | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ................................................................................................................................................................ATATGGTCTGCACCGTGAT........................................................................ | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 |
| ........................................................................................................................................................................................................................GTGGAGTTCTTACGTGT.................. | 17 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
|
GCGTTCATGTGAACTATTGGTAAATCATGACTGATGCACGTGAATTGTACCACTCGTTAGCTAAGCTACTCTTTTTTCTATTAAAATGTTGGTTAAGGTCACGCACCAAACTTTGGGGCGACAAAGCCAAGTCTACCTATTAACTTACAAATAAATACGTTATACCAGACCTGTCGCTATAAAAAACTTTACTTAAAAGTACCTCGTTAAGGTTTCCAACTCAAGTATGCACATATTCAAACTTTGACTAA
**************************************************************************(((((.(((.(((..((.((((.(((....))).)))).))...))).))))))))..*********************************************************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M028 head |
SRR060662 9x160_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|
| ...........................................................................................................................................................................................................TCGTCAAGCTTTCCAACT.............................. | 18 | 2 | 5 | 0.20 | 1 | 1 | 0 |
| ..........................................................................................................CAAACTTGGTGGCGACAA............................................................................................................................... | 18 | 2 | 7 | 0.14 | 1 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:19440639-19440889 + | dvi_20678 | CGCAAGTACACTTGATAACCATTTAGTACTGACTACGTGCACTTAACATGGTGAGCAATCGATTCGATGAGAAAAAAGATAATTTTACAACCAATTCCAGTGCGTGGTTTGAAACCCCGCTGTTTCGGTTCAGATGGATAATTGAATGTTTATTTATGCAATATGGTCTGGACAGCGATATTTTTTGAAATGAATTTTCATGGAGCAATTCCAAAGGTTGAGTTCATACGTGTATAAGTTTGAAACTGATT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
Generated: 05/16/2015 at 08:29 PM