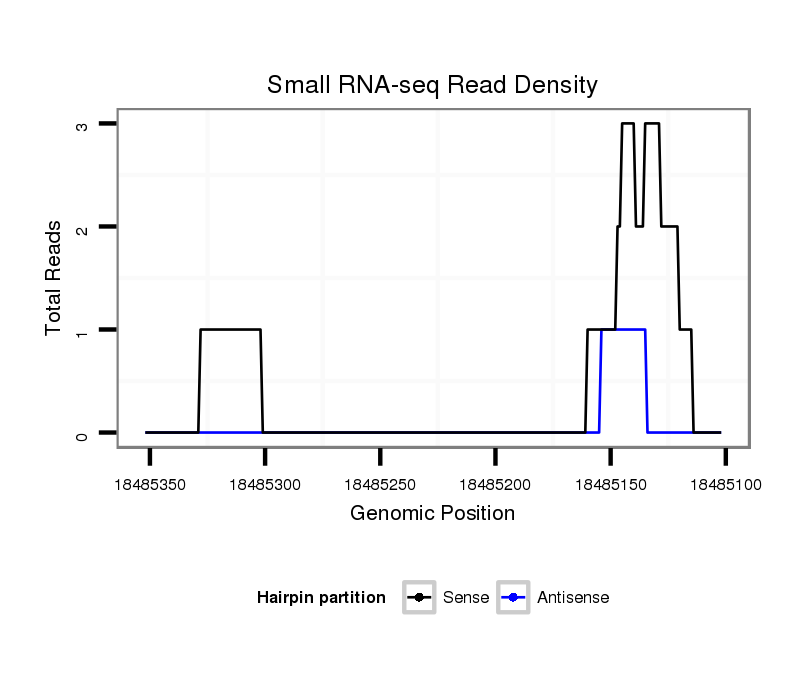
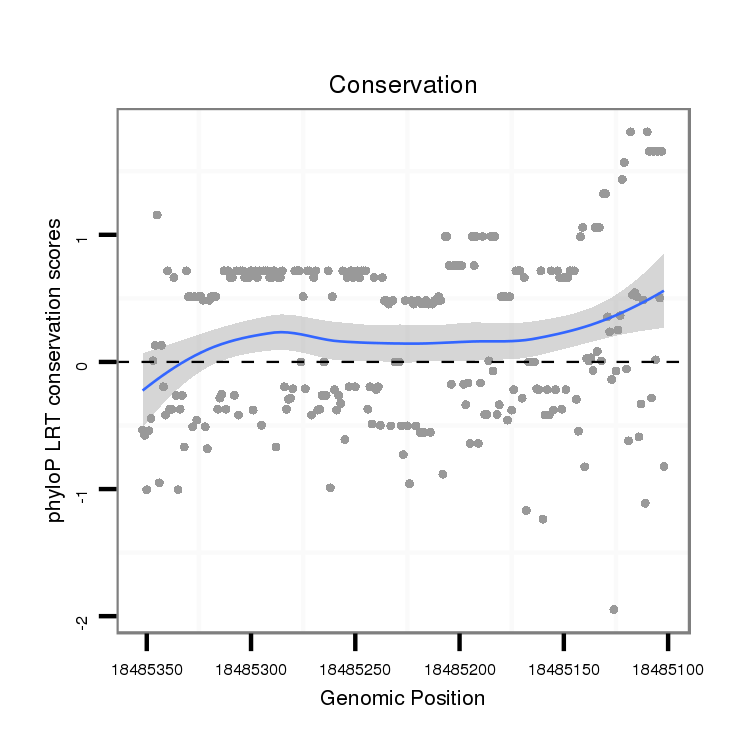
ID:dvi_20478 |
Coordinate:scaffold_13049:18485152-18485302 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ11548-cds]; exon [dvir_GLEANR_11579:2]; intron [Dvir\GJ11548-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CATTCGCACCGTAGCCTTGTCTTTAATAAGTTGTTAGTAAGAGGAAATTCCCAATCAAAATGCACGGTGGGTAAAAATAAAAGAAGACATTTTAAGTCGCAGGTGTTTCGCCTGAGTACTAGTACATAGAGTTTGAACTGTTTGAATTGTTTTATACTATATGATATACATAAAATAAGTTTTCTACTAATTCATATTCAGCGTCATTGCGAGTCGGTATCGGAGTAATCCTGCTGGCAATATGCCTGCCC **************************************************.................((((((..(((...(((((((((((((.((..((((((...))))))..........((((.(((..((((..........))))...))).))))....)).))))))..)))))))....)))..)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M061 embryo |
SRR060655 9x160_testes_total |
SRR060657 140_testes_total |
SRR060675 140x9_ovaries_total |
SRR060688 160_ovaries_total |
V047 embryo |
SRR060678 9x140_testes_total |
SRR060660 Argentina_ovaries_total |
SRR060676 9xArg_ovaries_total |
M047 female body |
SRR060659 Argentina_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060674 9x140_ovaries_total |
SRR060680 9xArg_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................................................................................................................................................................TGCGAGTCGGTATCGGAGTAATCCT................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................ATTGCGAGTCGGTATCGGA........................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................AATAAGTTGTTAGTAAGAGGAAATTCC........................................................................................................................................................................................................ | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................................TATCGGAGTAATCCTGCTGGC............. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................GGTATCGGAGTAATCCTGTTGGC............. | 23 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................CATATTCAGCGTCATTGCGAG...................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................TACATAGAGTTTGAACCGTTTGAA......................................................................................................... | 24 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................TAGTACATAGAGGTTGAAA................................................................................................................. | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................TACTAGTACATAGAGGTTGTA.................................................................................................................. | 21 | 2 | 6 | 0.33 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................TAGTACATAGAGTTTGGCCTT............................................................................................................... | 21 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................TAGTACATAGAGGTTGTA.................................................................................................................. | 18 | 2 | 7 | 0.29 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 |
| .......................................................................................................................TAGTACATAGAGGTTTAA.................................................................................................................. | 18 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................TCGGGATCGGAGTAATGAT................... | 19 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................................................TACTAGTACATAGAGTTTGG................................................................................................................... | 20 | 1 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................TTCGGGATCGGAGTAATG..................... | 18 | 3 | 20 | 0.15 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 1 | 0 | 0 |
| ....................................................................................................................TACTAGTACATAGAGGTTTA................................................................................................................... | 20 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................................................................TAGTACATAGAGGTTGAG.................................................................................................................. | 18 | 2 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................CGGGATCGGAGTAATGAT................... | 18 | 3 | 17 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
GTAAGCGTGGCATCGGAACAGAAATTATTCAACAATCATTCTCCTTTAAGGGTTAGTTTTACGTGCCACCCATTTTTATTTTCTTCTGTAAAATTCAGCGTCCACAAAGCGGACTCATGATCATGTATCTCAAACTTGACAAACTTAACAAAATATGATATACTATATGTATTTTATTCAAAAGATGATTAAGTATAAGTCGCAGTAACGCTCAGCCATAGCCTCATTAGGACGACCGTTATACGGACGGG
**************************************************.................((((((..(((...(((((((((((((.((..((((((...))))))..........((((.(((..((((..........))))...))).))))....)).))))))..)))))))....)))..)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V116 male body |
V053 head |
SRR060682 9x140_0-2h_embryos_total |
SRR060658 140_ovaries_total |
SRR060677 Argx9_ovaries_total |
SRR1106724 embryo_12-14h |
GSM1528803 follicle cells |
SRR1106730 embryo_16-30h |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................................................................................................GTCGCAGTAACGCTCAGCCA................................. | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...............................................................................................AAGCGTCCACAAAGCGGACTCAT..................................................................................................................................... | 23 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................GTTTTAGCTGGCACCCATTTTTA............................................................................................................................................................................. | 23 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................GAGGACTCGTGATCATGAATC.......................................................................................................................... | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................TCGCAGTAACGTTCCGTCATAGC............................. | 23 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................................CCTCATTAGGTCGACAGTCA.......... | 20 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................................................................CCACCCATTTTTCTTTTCC....................................................................................................................................................................... | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ....................................................................ACCATTTTTATTTTC........................................................................................................................................................................ | 15 | 1 | 20 | 0.45 | 9 | 0 | 0 | 0 | 0 | 0 | 0 | 9 | 0 |
| ............................................................................................................GAGGACGCATGATCATGAATC.......................................................................................................................... | 21 | 3 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................ATAAGTCCCAGTAAC.......................................... | 15 | 1 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ............ACGAAACAGGAATTATTCAAC.......................................................................................................................................................................................................................... | 21 | 3 | 8 | 0.13 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:18485102-18485352 - | dvi_20478 | CATTCGCACCGTAGCCTTGTCTTTAA------TAAGTTGTTAGTAAGAGGAAATTCCCAATC-----AAAATGCACGGTGGGTAAAAATAAAAGAAGACATTTTAAGTCGCAGGTGTTTCGC-----CTGAGTACTAGTACATAGAGTTTGAACTGTT--------------------------------------TGAATTGTTTTATACTATA-TGATATACATAAAATAAGTTTTCTACTAATTCATATTCAGCGTCATTGCGAGTCGGT-ATCGGAGTAATCCTGCTGGCAATATGCCTGCCC------- |
| droMoj3 | scaffold_6680:19285735-19285964 - | CAGCAACATCGAAATCAGAAAT--------------------ACGAAAGGTATTTCCCAATCAATCGACAATGCAAGGTGAGCAAAA--AAATGAAAT-TTA-TATTTGGCAGGTGTTTTGCGCGACCTGCGTACCA---TCTGCAGCTAGTACAGTTCCCGCGAATATTACCATATACTATTGTTT---------------------------------------------AATTTCCC----ATTGTTTTTTAGTGTCATTGAGATTTGGT-ATTGGGGTGATCCTGCTTGCCCTGTGCCTGCCT------- | |
| droGri2 | scaffold_15110:9164023-9164281 + | GAACCACAACAAAATCTAATATTTTACACTCGCAAGAGGTTAACAAAAGGAATTTCCCACTCTCTCGAAAATGCAAGGTAATCGAAA-TGAATGACAA-ATGTACAGTCGTAGATGTTTTAG-CGAACAAAG-------------------------------AAATATTCATATGTGCTTATATATTAAGATGCTTGCAT--------GTGA-ACGAAATTACAAGAAACACGTTTCCG----GATTTATTGCAGTGACATTTCGATTTAGT-ATTGGAGTTATCCTTCTGGCATTATGGCAGCCC------- | |
| droWil2 | scf2_1100000004511:3141833-3141852 - | TG-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTGGTCTTGTGCCTGACA------- | |
| dp5 | XR_group6:2082152-2082197 + | TTG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGAGGTGCTGTTTGGAGCCTGCCTGCTGATCTTCTGCCTGCCG------- | |
| droPer2 | scaffold_9:375397-375428 + | TT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGAGCCTGCCTGCTGATCTTCTGCCTGCCG------- | |
| droBip1 | scf7180000396709:82219-82220 + | CT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droFic1 | scf7180000454113:2087454-2087477 - | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCTGATGGTGATATGTCTGCCGT------ | |
| droEle1 | scf7180000491193:4539580-4539618 + | TC-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGATGTCTTCTGCTGGTGACTTGTCAGCCGGCTTTCT | |
| droRho1 | scf7180000777860:4225-4255 + | TC-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGATGGCTTCTTGTGGTGACTTGCCAGCC-------- | |
| droEre2 | scaffold_4820:172366-172404 + | GAAATATATT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GATTATTTTACACTATA-TGATAAAAATA-------------------------------------------------------------------------------A------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 04:35 AM