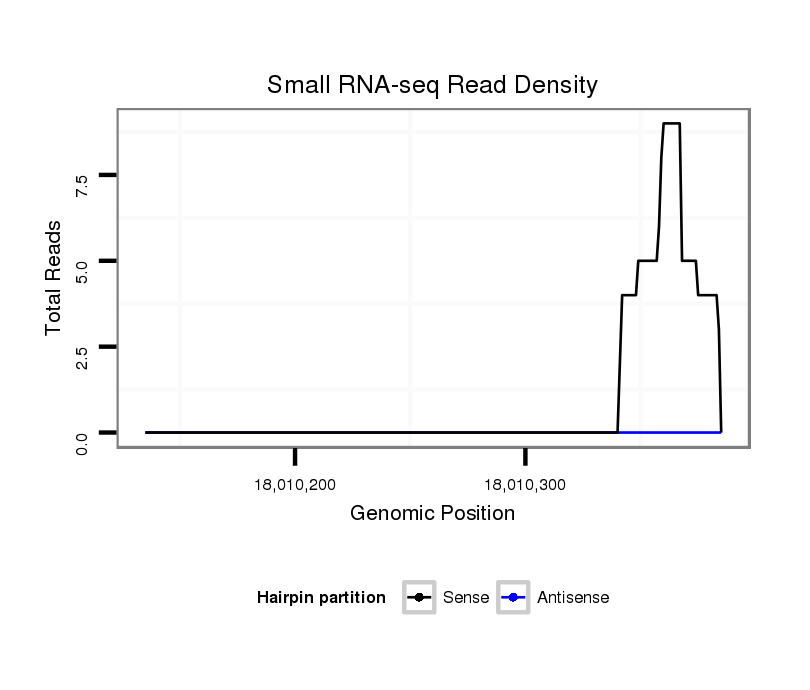
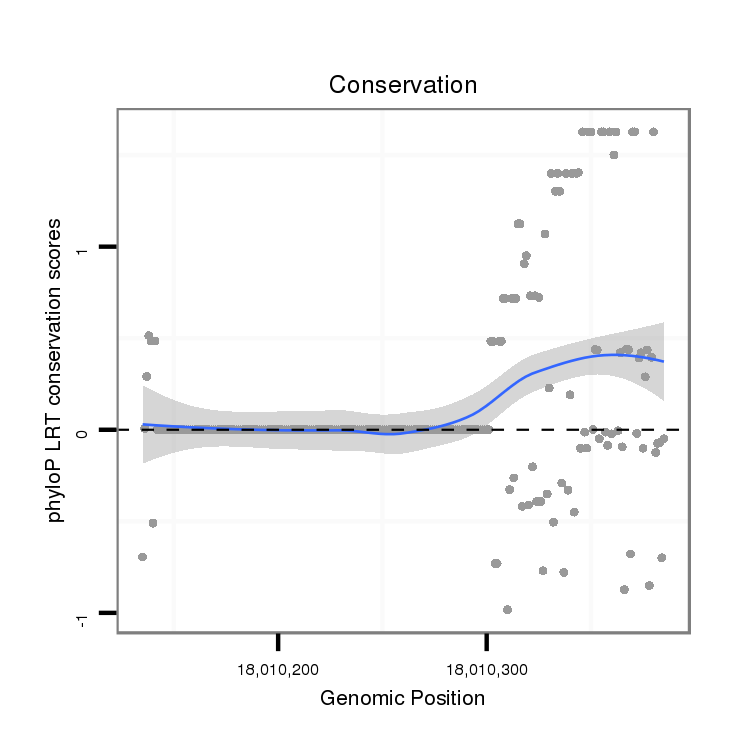
ID:dvi_20404 |
Coordinate:scaffold_13049:18010185-18010335 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ13738-cds]; exon [dvir_GLEANR_13606:9]; intron [Dvir\GJ13738-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## GATAGACTGTACTCTGATTCAACCTAAAGAAATATAATAATTTTTTTTGTGGATTTGAATCGGTAACTTCTCGGTTTCAGCTGTGCATCGGGTCGAATTATGTTTCTTCTAGAGTTTCTTCAAAGTAGCTCTCATGCTTGTTAAATCTGATCTATAATTGTAAAAAATAATTAATGGTTATTACAATAATCTTATTTACAGGCTACTTAATACGTATAAAAAGATACACAAGTTCATGAATGTAATTGCTT **************************************************......(((((((.......)))))))(((.((((.(((((((..(((((((........((((((.((....)).))))))))))...)))..))))))).)))).))).......(((.(((.((((((....))))))))).)))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060676 9xArg_ovaries_total |
M047 female body |
SRR060654 160x9_ovaries_total |
SRR060674 9x140_ovaries_total |
SRR060677 Argx9_ovaries_total |
SRR060679 140x9_testes_total |
SRR060681 Argx9_testes_total |
SRR060688 160_ovaries_total |
V047 embryo |
SRR060682 9x140_0-2h_embryos_total |
SRR060689 160x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................................................................................................................................................................TAATACGTATAAAAAGATACACAAGT.................. | 26 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................TTAATACGTATAAAAAGATACACAAGT.................. | 27 | 0 | 1 | 2.00 | 2 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................................TACACAAGTTCATGAATGTAATTGCT. | 26 | 0 | 1 | 2.00 | 2 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................ATACACAAGTTCATGAATGTAATTGC.. | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................GTGCATCGGGTCGAATTATAA.................................................................................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................TATAAAAAGATACACAAGTTCATGAA........... | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................................ACACAAGTTCATGAATGTAATTGCT. | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................TCACATCCTTCTTAAATCTGAT.................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................................................................................................................................TCACATCCTTTTTAAATCTGAT.................................................................................................... | 22 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .......................CTAAGGAAATATAAGAATT................................................................................................................................................................................................................. | 19 | 2 | 7 | 0.14 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................TAAAGAAATTTAATAATTT................................................................................................................................................................................................................ | 19 | 1 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...................................................................TTCTCCGTTTCAGCTGTCC..................................................................................................................................................................... | 19 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
CTATCTGACATGAGACTAAGTTGGATTTCTTTATATTATTAAAAAAAACACCTAAACTTAGCCATTGAAGAGCCAAAGTCGACACGTAGCCCAGCTTAATACAAAGAAGATCTCAAAGAAGTTTCATCGAGAGTACGAACAATTTAGACTAGATATTAACATTTTTTATTAATTACCAATAATGTTATTAGAATAAATGTCCGATGAATTATGCATATTTTTCTATGTGTTCAAGTACTTACATTAACGAA
**************************************************......(((((((.......)))))))(((.((((.(((((((..(((((((........((((((.((....)).))))))))))...)))..))))))).)))).))).......(((.(((.((((((....))))))))).)))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060667 160_females_carcasses_total |
SRR060669 160x9_females_carcasses_total |
GSM1528803 follicle cells |
SRR060670 9_testes_total |
M027 male body |
SRR060666 160_males_carcasses_total |
SRR060668 160x9_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................AAGTTGCATCGAGAGTAC................................................................................................................... | 18 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................CAACACGTAGCCCAGCTTAA........................................................................................................................................................ | 20 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................TAGCCATAGAAGATCCAA............................................................................................................................................................................... | 18 | 2 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................................................TTAGCCATTGAAGAG................................................................................................................................................................................... | 15 | 0 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................................ATGTGATCCAGGACTTACA........ | 19 | 3 | 16 | 0.19 | 3 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 1 |
| ....................................................................AGATTCAAAGTCGACACGT.................................................................................................................................................................... | 19 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................................AGGTGTGCAAGTACTTAC......... | 18 | 2 | 9 | 0.11 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................ATCCAAAGAAGACCCCAAAG..................................................................................................................................... | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................AAAGAAGTTTCATTG.......................................................................................................................... | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:18010135-18010385 + | dvi_20404 | GATAGACTGTACTCTGATTCAACCTAAAGAAATATAATAATTTTTTTTGTGGATTTGAATCGGTAACTTCTCGGTTTCAGCTGTGCATCGGGTCGAATTATGTTTCTTCTAGAGTTTCTTCAAAGTAGCTCTCATGCTTGTTAAATCTGATCTATAATTGTAAAAAATAATTAATGGTTAT--------------------TACAATAATCTTATTTACAGGCTACTTAATACGTATAAAAAGATACACAAGTTCATGAATGTAATTGCTT |
| droMoj3 | scaffold_6680:23555025-23555105 + | AAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------TACGTAATATTAAT--------------------TACAT------AAATTGCAGATTACTTAATACTTATAAGAAGATACATAAATTCATAAATGTGATTTCCC | |
| droGri2 | scaffold_15110:24050398-24050493 + | ATAAGTC----------------------------------------------------------------------------------------------------------------------------------------------------------------------ATTGTTATCATTATTATCATTACTTTTTTG---------CAATTACAGACTTCTAAATACTTATAAAAAGGTTCATAAATTTATGAATGTTATCGCAT | |
| droWil2 | scf2_1100000004943:1211195-1211256 + | TTT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CATTTTCAGATTGATGAAAACTTACAAAAAGATTCACCGCTTTATGACAGTAATTTCAT | |
| droAna3 | scaffold_13340:7429745-7429787 - | A------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGATTTACGAGAAAATACACACGACCATAAAGGTGCTGGGTC | |
| droRho1 | scf7180000776882:5340-5410 + | TTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T--------------------TTCAATGAGCG--TGTTCAGTGTGCTTA-TAAATATATAAATGTACACAAATTGATGCATCAAATTGTTT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
Generated: 05/16/2015 at 11:47 PM