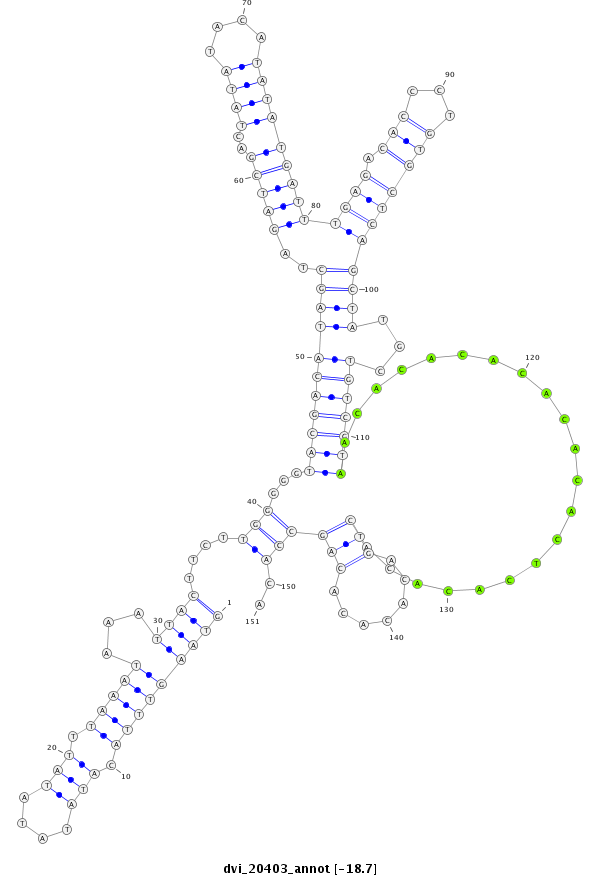
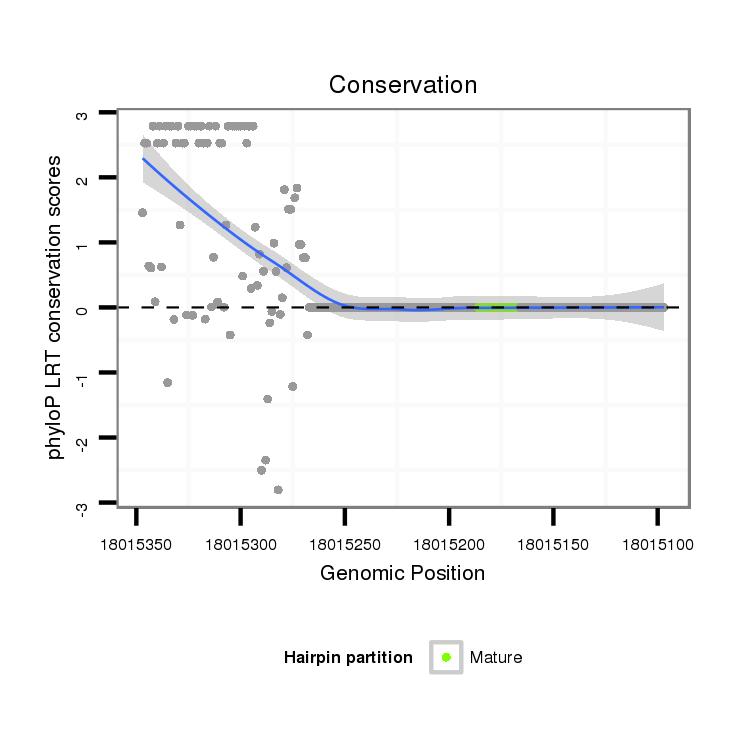
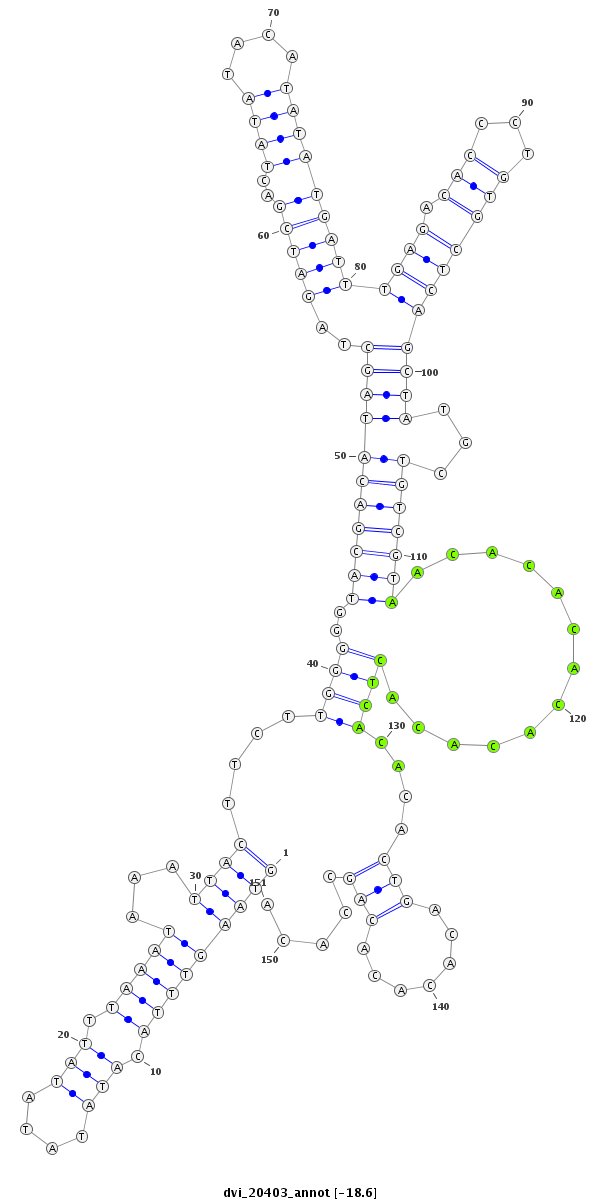
ID:dvi_20403 |
Coordinate:scaffold_13049:18015147-18015297 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -18.6 | -18.4 | -18.4 |
 |
 |
 |
exon [dvir_GLEANR_11610:4]; CDS [Dvir\GJ11582-cds]; intron [Dvir\GJ11582-in]; Antisense to intron [Dvir\GJ13738-in]
| Name | Class | Family | Strand |
| (TG)n | Simple_repeat | Simple_repeat | + |
| AT_rich | Low_complexity | Low_complexity | + |
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- TGCCCTGGATGACAATGTGGCTTTCATGTCAGACTTACCTCTGATAAAAAGTAAGTTTACATATATATATTTAAATAAATTACTTCTTGGGGGTACGACATAGCTAGATCGACTATATACATATATGATTTGAGACACCCTGTGCTCAGCTATGCTGTCGTAACACACACACACACTCACACACTGACACACACAGCCACACATACGTATGGTTCATGCTGCGCCCCTTAGGCAGAGGCTGGGCTTCTCCC **************************************************(((((((((.(((....))).)))))...))))....(((...(((((((((((..(((((..((((....)))))))))((((.(((...)))))))))))...))))))).....................(((.......))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060686 Argx9_0-2h_embryos_total |
V053 head |
SRR060672 9x160_females_carcasses_total |
SRR060666 160_males_carcasses_total |
SRR060669 160x9_females_carcasses_total |
SRR060654 160x9_ovaries_total |
M061 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................................................................................CTTGAGGGTACGACATATGTAG................................................................................................................................................ | 22 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................AACTTGCTTCTTGGGGGT............................................................................................................................................................. | 18 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................GCTCCTTGTGGGTACGACA....................................................................................................................................................... | 19 | 3 | 10 | 0.40 | 4 | 0 | 0 | 2 | 1 | 1 | 0 | 0 |
| ....................................................................................................................TTACAGATATGATTTGATACA.................................................................................................................. | 21 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................................................................................................................................................AACACACACACACACTCACA...................................................................... | 20 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
ACGGGACCTACTGTTACACCGAAAGTACAGTCTGAATGGAGACTATTTTTCATTCAAATGTATATATATAAATTTATTTAATGAAGAACCCCCATGCTGTATCGATCTAGCTGATATATGTATATACTAAACTCTGTGGGACACGAGTCGATACGACAGCATTGTGTGTGTGTGTGAGTGTGTGACTGTGTGTGTCGGTGTGTATGCATACCAAGTACGACGCGGGGAATCCGTCTCCGACCCGAAGAGGG
**************************************************(((((((((.(((....))).)))))...))))....(((...(((((((((((..(((((..((((....)))))))))((((.(((...)))))))))))...))))))).....................(((.......))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060682 9x140_0-2h_embryos_total |
V116 male body |
SRR1106729 mixed whole adult body |
SRR060686 Argx9_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
M028 head |
M027 male body |
SRR060685 9xArg_0-2h_embryos_total |
M047 female body |
SRR060687 9_0-2h_embryos_total |
SRR060659 Argentina_testes_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060664 9_males_carcasses_total |
SRR060681 Argx9_testes_total |
SRR060663 160_0-2h_embryos_total |
SRR060668 160x9_males_carcasses_total |
SRR060679 140x9_testes_total |
M061 embryo |
SRR060680 9xArg_testes_total |
SRR060672 9x160_females_carcasses_total |
SRR060661 160x9_0-2h_embryos_total |
SRR1106728 larvae |
SRR060689 160x9_testes_total |
V053 head |
SRR060657 140_testes_total |
SRR1106727 larvae |
V047 embryo |
SRR060658 140_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..................CCGAAAGTGTAGTCTGAAT...................................................................................................................................................................................................................... | 19 | 2 | 3 | 11.00 | 33 | 6 | 9 | 2 | 1 | 1 | 3 | 4 | 1 | 1 | 1 | 1 | 0 | 0 | 1 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................CCGAAAGTGTAGTCTGAATT..................................................................................................................................................................................................................... | 20 | 3 | 19 | 6.58 | 125 | 16 | 23 | 4 | 15 | 23 | 11 | 6 | 5 | 6 | 3 | 0 | 4 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 1 | 1 | 3 | 1 | 0 | 0 | 0 | 1 |
| .................TCCGAAAGTGTAGTCTGAAT...................................................................................................................................................................................................................... | 20 | 3 | 14 | 6.00 | 84 | 5 | 13 | 42 | 10 | 3 | 1 | 2 | 3 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 1 | 1 | 0 |
| ..................CCGAAAGTGTAGTCTGAA....................................................................................................................................................................................................................... | 18 | 2 | 6 | 4.17 | 25 | 11 | 1 | 0 | 3 | 3 | 0 | 0 | 0 | 0 | 1 | 1 | 2 | 0 | 1 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................TCCGAAAGTGTAGTCTGAA....................................................................................................................................................................................................................... | 19 | 3 | 20 | 4.10 | 82 | 15 | 9 | 14 | 12 | 8 | 2 | 1 | 1 | 2 | 2 | 4 | 2 | 0 | 0 | 3 | 0 | 0 | 0 | 1 | 0 | 1 | 1 | 0 | 2 | 2 | 0 | 0 | 0 |
| ..................CCGAAAGTGTAGTCTGAATC..................................................................................................................................................................................................................... | 20 | 3 | 9 | 0.67 | 6 | 2 | 1 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................ACAGTCAGAATGGAGACTT.............................................................................................................................................................................................................. | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................CCGAAAGTTTAGTCTGAAT...................................................................................................................................................................................................................... | 19 | 2 | 5 | 0.40 | 2 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................ACAGTCAGAATGGAGACTTG............................................................................................................................................................................................................. | 20 | 3 | 5 | 0.40 | 2 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................TGTGTGAGTGTGTGACTGTGTG........................................................... | 22 | 0 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................GAAAGTATAGTCTGAATC..................................................................................................................................................................................................................... | 18 | 2 | 10 | 0.10 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................AGTCTGAGTGGCAACTATT............................................................................................................................................................................................................ | 19 | 3 | 11 | 0.09 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................TTCCGAAAGTATAGTCTGAA....................................................................................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:18015097-18015347 - | dvi_20403 | TGCCCTGGATGACAATGTGGCTTTCATGTCAGACTTACCTCTGATAAAAAGTAAGTT-----TACATAT---ATA----TA------TT-TAAA-TAAATTACTTCTTGGGGGTACGACATAGCTAGATCGACTATATACATATATGATTTGAGACACCCTGTGCTCAGCTATGCTGTCGTAACACACACACACACTCACACACTGACACACACAGCCACACATACGTATGGTTCATGCTGCGCCCCTTAGGCAGAGGCTGGGCTTCTCCC |
| droMoj3 | scaffold_6680:23568465-23568538 - | TGCCCTGGATGAGAATGTGGCGTTCATGTCAGACTTACCTCTAATAAAAAGTAATTT-----CATTTA--AG-GA----CA------C---TAA-----A--------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_15110:24059348-24059413 - | TGCTTTGGATGATAATGTGGCTTTTATGTCAGATTTACCTCTGATAAAAAGTAAGTT-----TAAATTT------------------------A-----T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004540:1019148-1019208 - | CGCTCTTGATGATAACGTGGCGTTTATGTCAGATCTTCCCTTAATAAAGAGTAAGTT-----AATA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | XR_group8:456621-456679 + | TGCCATAGATGACAACGTAGCCTTCATGTCAGACCTTCCACTAATAAAAAGTAAGAT-----TA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_96:11288-11346 - | TGCCATAGATGACAACGTAGCCTTCATGTCAGACCTTCCACTAATAAAAAGTAAGAT-----TA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_13337:17032096-17032168 - | TGCCCTAGACGATAATGTGGCTTTCATGTCGGATTTGCCGCTAATAAAAAGTAAGCT-----ATAT-A----AAA----TA--CTCCT-----------T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000396641:519825-519901 - | TGCCCTAGACGACAATGTGGCTTTCATGTCGGATTTGCCGCTAATAAAAAGTAAGCT-----AT-ATA----ATA----TA--TTCATT-T-AA-----A--------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302808:75665-75721 + | TGCCCTCGACGATAATGTGGCTTTTATGTCCGACTTGCCGCTTATAAAGAGTAAGTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000453831:437721-437797 + | TGCCCTCGACGACAACGTGGCGTTTATGTCGGACTTGCCGCTTATAAAGAGTGAGTT-----GAAACA--AGACA----CA------TT-TCAA-----T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491255:2682696-2682775 - | TGCCCTCGACGATAACGTGGCGTTTATGTCAGACTTGCCGCTTATAAAGAGTGAGTT-----CTAACA--AAAAC----TA--TCTCTT-T-AA-----A--------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000780059:149833-149889 - | TGCCCTCGACGATAACGTGGCGTTTATGTCAGACTTGCCGCTTATAAAGAGTGAGTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000302377:1716801-1716857 - | TGCCCTCGACGACAATGTGGCGTTTATGTCGGACTTGCCGCTCATAAAGAGTAAGTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415388:304166-304237 + | TGCCCTCGACGATAATGTGGCGTTTATGTCAGACTTGCCGCTTATAAAGAGTGAGTT-----AATCAA--ATAAA----TA------T-----------T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409472:425886-425972 + | TGCCCTCGATGACAATGTGGCGTTCATGTCAGACTTACCGCTTATAAAGAGTAAGTT-----GATCCA--AAAAA----TATCCTC-TTTT-AAATAAAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dm3 | chr3L:21604401-21604457 + | TGCCCTCGACGACAATGTGGCGTTCATGTCAGACTTGCCGCTCATAAAGAGTAAGTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | 3l:21122178-21122234 + | TGCCCTCGACGACAATGTGGCGTTCATGTCAGATTTGCCGCTCATAAAGAGTAAGTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_11:1524860-1524942 + | TGCCCTCGACGACAATGTGGCGTTCATGTCAGACTTGCCGCTCATAAAGAGTAAGAGTTCTCTAGATCTATAAAC----TA--TCA---------TA--A--------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | v2_chr3L_random_021:29983-30063 + | TGCCCTCGACGACAACGTGGCGTTCATGTCGGACTTGCCACTCATAAAGAGTAAGTG-----CACCTA--ATGGGCATAT-----C-TT-TTAA-----T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4784:21259761-21259838 + | TGCCCTCGACGACAATGTGGCGTTCATGTCAGACTTGCCGCTCATAAAGAGTAAGTT-----CACATT--GG-CA--TAT-----C-TT-TCAA-----T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 11:55 PM