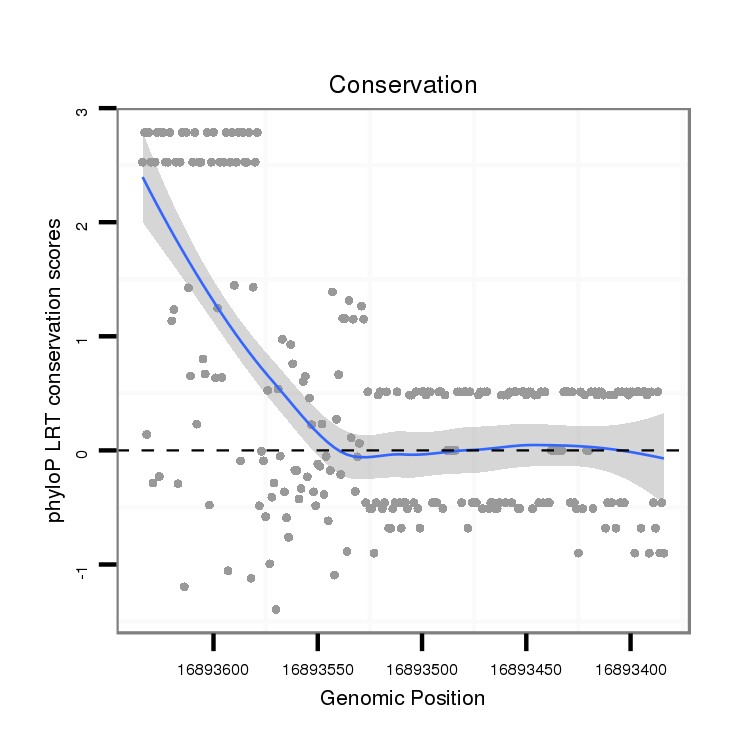
ID:dvi_20252 |
Coordinate:scaffold_13049:16893434-16893584 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_11690:1]; CDS [Dvir\GJ11668-cds]; intron [Dvir\GJ11668-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CAAACAGATATGGAACGACTTAAGCAAGGACTTGTCGGTGTGCAGGTCTGGTAAGTAGTAAACTATCTAAACAACTTTAATTTAAATAAATAGTCTTTAAGAGCAATCAAAGCCTATACATTATGCGAGGCTAGTTGAAGACTTAGCTGTCTAGAAAATAACTATATCCAATGCTGATAAAATTGAATCCTACTCAGCTAGCAAAATAACAACAGATTCATCGACCCAACTGACAACATGTTGGTTCTCTG **************************************************....((((....)))).........................(((((((((.(((..((...((.((.....)).)))).)))..)))))))))((((((..(((..........((((.......))))..........)))..)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
SRR060679 140x9_testes_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
M047 female body |
V116 male body |
M027 male body |
SRR060675 140x9_ovaries_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060672 9x160_females_carcasses_total |
M028 head |
SRR060673 9_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................TTGTCGGTGTACAGCTATGGT....................................................................................................................................................................................................... | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................GAATCCTACTCAGGGAGCCA............................................... | 20 | 3 | 7 | 0.43 | 3 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................................................................................................................................TTGAATCCCACTCAGGGAGC................................................. | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................ATTCACCGCCCCACCTGACA................ | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................................ACTGGCAAAATGTTGGTTC.... | 19 | 2 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................AGTCTGTAAGAGCAACCAA............................................................................................................................................. | 19 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................CAATGCTGATAAAAATGAAA............................................................... | 20 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................ATTCATCGACGCTACTGGC................. | 19 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ................GACGAAACCAAGGACTTGT........................................................................................................................................................................................................................ | 19 | 3 | 18 | 0.11 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................TTAAGAGAAATCAAAGTCT........................................................................................................................................ | 19 | 2 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ....................................................................................................................................................................................................................................ACTGGCAAAATGTTGGTTCC... | 20 | 3 | 12 | 0.08 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................GTAGTAAACTATGAATACAA................................................................................................................................................................................. | 20 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..................................................................................................................................................................................................................................GAACTGGCAAAATGTTGGTT..... | 20 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................TGACTAGATCCCATGCTG........................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GTTTGTCTATACCTTGCTGAATTCGTTCCTGAACAGCCACACGTCCAGACCATTCATCATTTGATAGATTTGTTGAAATTAAATTTATTTATCAGAAATTCTCGTTAGTTTCGGATATGTAATACGCTCCGATCAACTTCTGAATCGACAGATCTTTTATTGATATAGGTTACGACTATTTTAACTTAGGATGAGTCGATCGTTTTATTGTTGTCTAAGTAGCTGGGTTGACTGTTGTACAACCAAGAGAC
**************************************************....((((....)))).........................(((((((((.(((..((...((.((.....)).)))).)))..)))))))))((((((..(((..........((((.......))))..........)))..)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060660 Argentina_ovaries_total |
V047 embryo |
V053 head |
V116 male body |
SRR060685 9xArg_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
GSM1528803 follicle cells |
SRR060663 160_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...GGTCTATACCTTGTTGAATTC................................................................................................................................................................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................TTCTGAATCGAGAGATCTT............................................................................................... | 19 | 1 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................GTAAGTAGCTGTGGTGACTG................. | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................AGTCGAACGTTTTATTGGAGT..................................... | 21 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................ATAAGTAGTAGGCTCCGAT...................................................................................................................... | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................................TGACTGTTGTATAACCTA..... | 18 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .........................................................................TGAAATTAAATTTTTTCATCAA............................................................................................................................................................ | 22 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .......................................CACGCCCGGACCATTC.................................................................................................................................................................................................... | 16 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ................................................................TGGATTTGTTGAAATTATA........................................................................................................................................................................ | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................TGGGGTGACTGTGGTATA.......... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:16893384-16893634 - | dvi_20252 | CAAACAGATATGGAACGACTTAAGCAAGGACTTGTCGGTGTGCAGGTCTGGTAAGTAGT--A-----A---ACTATCTAAACAACT--------------TT--------AATTTA----------------AATAAA-------TAGTCTTTAAGAGCAATCA----AAGCCTATACATTATGCGAGGCTAGTTGAAGACTTAGCTGTCTAGAAAATAACTA--------TAT---------------CCAATGCTGA-----TAAAATTGAATCCTACTCAGCTAGCAAAATAACAACAGATTCATCGACCCAACTGACAACATGTTGGTTCTCTG |
| droMoj3 | scaffold_6680:19790907-19791013 + | CAAACGGACATGGAACGGCTCAAGCAGGGACTAGTCGGCGTGCAAGTTTGGTGAGTGCT--T-----A---T-------------C--------------TT--------ATTTCTAAAATATATTAATATAATTTAG----AGAGATTCTTTTAG-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_15110:15258237-15258503 + | CAGACAGACATGGAACGACTTAAGCAAGGATTAGTTGGCGTCCAAGTTTGGTAAGTGAT--T-----G---C-------------TTCAATTTTGTATCCTC--------AACTCT----------------G----A-------CATCCCTTGAGAGACATTATTCATTCTCTTCAACACACTTATGGTTTTTTGAAAGTTTTG-----TAAAACACGGCTTAATGGTTGTAACAAAAGGTCTGATAGTTTTCGCCGATTACGCAATTTTGAAACCCATTTG------AAAGTGTGATC--AATCATCTGCTCCATTAACAAGATTTTGCTCATGCC | |
| droWil2 | scf2_1100000004762:5892801-5892857 + | CAAACCGATATGGAACGCCTAAAGCAGGGACTTGTTGGCGTTCAAGTGTGGTAAGTA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | XR_group6:1838436-1838492 - | CAGACCGACATGGAGCGGCTCAAGCAGGGTCTCGTCAGCGTTCAAGTTTGGTGAGTA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_9:134931-134987 - | CAGACCGACATGGAGCGGCTCAAGCAGGGTCTCGTCAGCGTTCAAGTTTGGTGAGTA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_13337:6104002-6104067 - | CAGACGGATATGGAGAGGCTGAGACAGGGACTAGTCAGTGTGCAAGTGTGGTATGTACC--A-----AATA---AT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000396419:43781-43854 - | CAGACGGATATGGAGCGGCTGAAACAAGGACTAGTCAGTGTGCAAGTGTGGTAAGTACC--A-----AAAA---GTT-----------------------TT--------AGTTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302391:972518-972574 + | CAGACGGACATGGAGCGGCTAAAGCAGGGACTGGTCAGCGTGCAAGTGTGGTGAGTA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000454113:2821997-2822076 + | CAGACCGATATGGAGCGACTCAAGCAGGGACTGGTCAGCGTCCAAGTGTGGTAAGTCCA-TT--GGA---AAATCTCTAAGC---------------------------------------------------------------TACT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491193:3741181-3741271 - | CAAACGGACATGGAGCGACTGAAGCAGGGACTGGTCAGCGTTCAAGTGTGGTTAGTCCT--T--GGA---AGA--TC-----------------------CTACAATTTTAATCT--------T--------TTTAAA-------T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droRho1 | scf7180000779608:11412-11507 + | CAAACGGACATGGAGCGACTCAAACAGGGACTGGTCAGCGTTCAAGTGTGGTGAGTCCT--T--GGA---AGA--TC-----------------------ATAAAACACTAATCT--------A--------TTTA------------------AAATAAAT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000302428:4082883-4082976 - | CAAACGGACATGGAGCGGCTCAAGCAGGGATTGGTCAGCGTCCAAGTGTGGTGAGTCCT--A-----AAAA---GTC-----------------------CTAAGATTAAAA----------------------AGTCCTAGA--TAGT----------AA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415392:263898-263984 - | CAAACGGACATGGAGCGACTCAAGCAGGGATTGGTCAGCGTCCAAGTGTGGTGAGTACT--G-----AAAA---GTC-----------------------CTAGAATTTAGA----------------------TATC-------TAGT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409711:3728413-3728468 - | CAAACGGATATGGAGCGACTTAAGCAGGGATTGGTCAGCGTTCAAGTTTGGTAAGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| dm3 | chr3L:11847342-11847397 - | CAAACGGACATGGAGCGACTCAAGCAGGGATTAGTCAGCGTCCAAGTGTGGTGAGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droSim2 | 3l:11552057-11552167 - | CAAACGGACATGGAGCGACTCAAGCAGGGATTAGTCAGCGTCCAAGTGTGGTAAGTTCCATTATGAAA---GCCAAATTCTCAGTA--------------TT--------ATTTCAAATATATA--------T------------------TTAAGAAAAAT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_0:4037498-4037553 - | CAAACGGACATGGAGCGACTCAAGCAGGGTTTAGTCAGCGTCCAAGTGTGGTAAGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droYak3 | 3L:11889348-11889403 - | CAAACGGACATGGAGCGACTCAAGCAGGGATTAGTCAGCGTCCAAGTGTGGTGAGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droEre2 | scaffold_4784:11835948-11836003 - | CAAACGGACATGGAGCGACTCAAGCAGGGATTAGTCAGCGTCCAAGTGTGGTGAGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 09:40 PM