
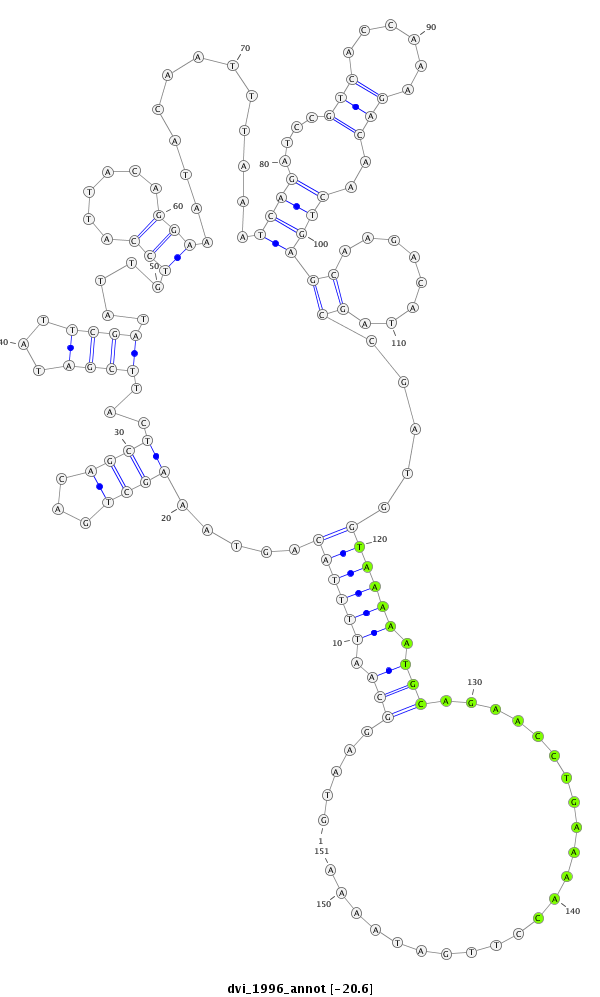
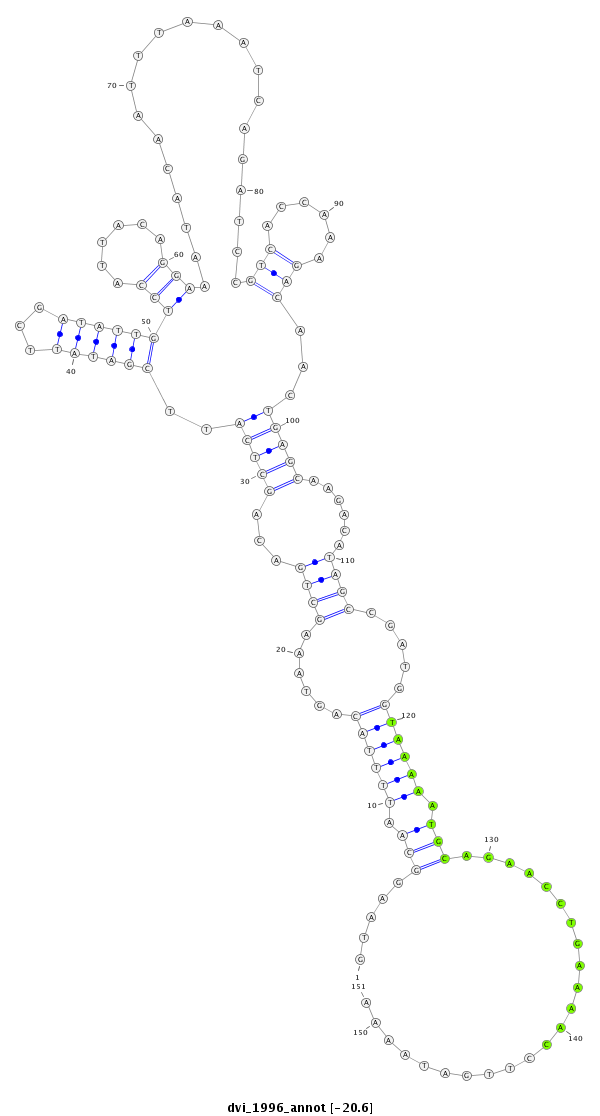
ID:dvi_1996 |
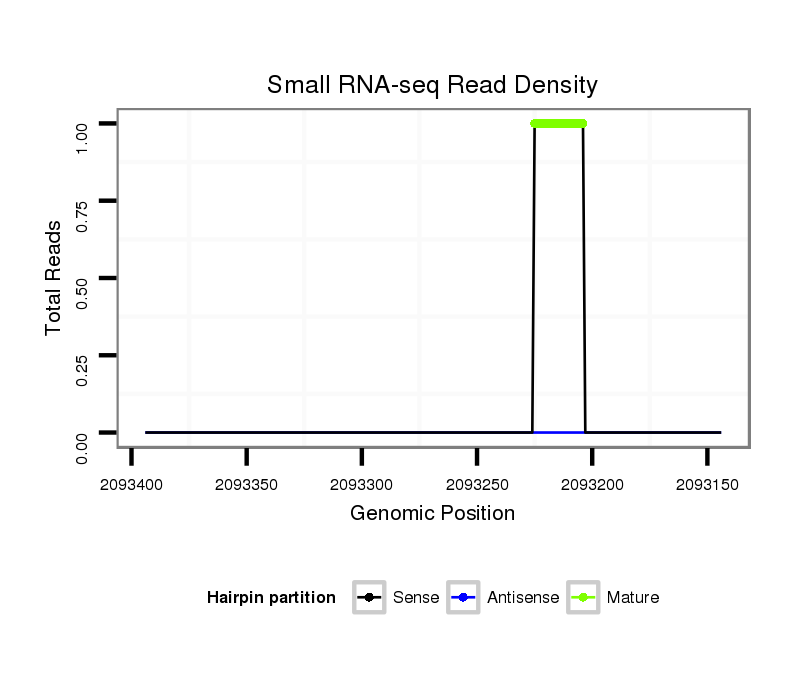
Coordinate:scaffold_12726:2093194-2093344 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -20.7 | -20.6 | -20.6 |
 |
 |
 |
CDS [Dvir\GJ15200-cds]; exon [dvir_GLEANR_15525:3]; intron [Dvir\GJ15200-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- AGATTTCTTTACTACACTCAGACAAAGTTGTCACTGCTGGCTCTCAAGAGGTAAGGCAATTTTACAGTAAAGCTGACAGCTCATTCGATATTCGATATTGTCCATTACAGGAAATACAATTTAAATCAGATCCGTCACCAAAGACAACTGAGCAAGACATAGCCGATGGTAAAAATGCAGAACCTGAAAACCTTGATAAAAGACAAAAAGTTACGCCAAAGCTAAATGAAAATAAGACAGTTGATGAAAAA **************************************************.....(((.((((((.....((((...))))....((((((...))))))(((......))).............((((....(((......)))..))))((........)).....)))))).))).......................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060679 140x9_testes_total |
V053 head |
V116 male body |
SRR060682 9x140_0-2h_embryos_total |
V047 embryo |
SRR060661 160x9_0-2h_embryos_total |
SRR060671 9x160_males_carcasses_total |
SRR1106728 larvae |
M047 female body |
SRR060669 160x9_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................................TCAAGATGTAAGGCAATT.............................................................................................................................................................................................. | 18 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................CTCAAGATGTAAGGCAATT.............................................................................................................................................................................................. | 19 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........TATTACTCTCAGACAAAGGTGT............................................................................................................................................................................................................................ | 22 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................TAAAAATGCAGAACCTGAAAAC............................................................ | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................................ATTTAAATGAAAATAAGACAGTC......... | 23 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........TACTACTGTCAGAGAAAGTTGTC........................................................................................................................................................................................................................... | 23 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................TCAAGATGTAAGGCAATTC............................................................................................................................................................................................. | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........TACTACAGTCAGGCAAAGATG............................................................................................................................................................................................................................. | 21 | 3 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................................ATAGGACAGATGATGAAA.. | 18 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........TAATACTCTCAGACAAAGATGTC........................................................................................................................................................................................................................... | 23 | 3 | 7 | 0.14 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................ATAGCCTGTGGTAATAATGC......................................................................... | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................ACGCCAAAGAGAAATGAAA.................... | 19 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................................................................................................................................CCTGAATTCCCTGATAAAAG................................................. | 20 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................................................................................................................................................................................GGAAACCCTGATAAAAGA................................................ | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................CATCCAGAACCTGAAAACG........................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .................................................................................................TTGAGCATTCCAGGAAATAC...................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
TCTAAAGAAATGATGTGAGTCTGTTTCAACAGTGACGACCGAGAGTTCTCCATTCCGTTAAAATGTCATTTCGACTGTCGAGTAAGCTATAAGCTATAACAGGTAATGTCCTTTATGTTAAATTTAGTCTAGGCAGTGGTTTCTGTTGACTCGTTCTGTATCGGCTACCATTTTTACGTCTTGGACTTTTGGAACTATTTTCTGTTTTTCAATGCGGTTTCGATTTACTTTTATTCTGTCAACTACTTTTT
**************************************************.....(((.((((((.....((((...))))....((((((...))))))(((......))).............((((....(((......)))..))))((........)).....)))))).))).......................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M027 male body |
SRR060666 160_males_carcasses_total |
V116 male body |
SRR060667 160_females_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................CCGTTAAGATGTCAATTCGACG............................................................................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................GTCTTGGGTTTCTGGAACTA...................................................... | 20 | 3 | 14 | 0.07 | 1 | 0 | 1 | 0 | 0 | 0 |
| .................................................................................................................................................................................GTCTTGGGCTTCTGAAACTA...................................................... | 20 | 3 | 15 | 0.07 | 1 | 0 | 0 | 1 | 0 | 0 |
| ............................................................AAATGTCATTTCGCTTGCC............................................................................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................TCTTGGCAGTGGTTACTT.......................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
| .......AATTGCTGCGAGTCTGTTT................................................................................................................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 |
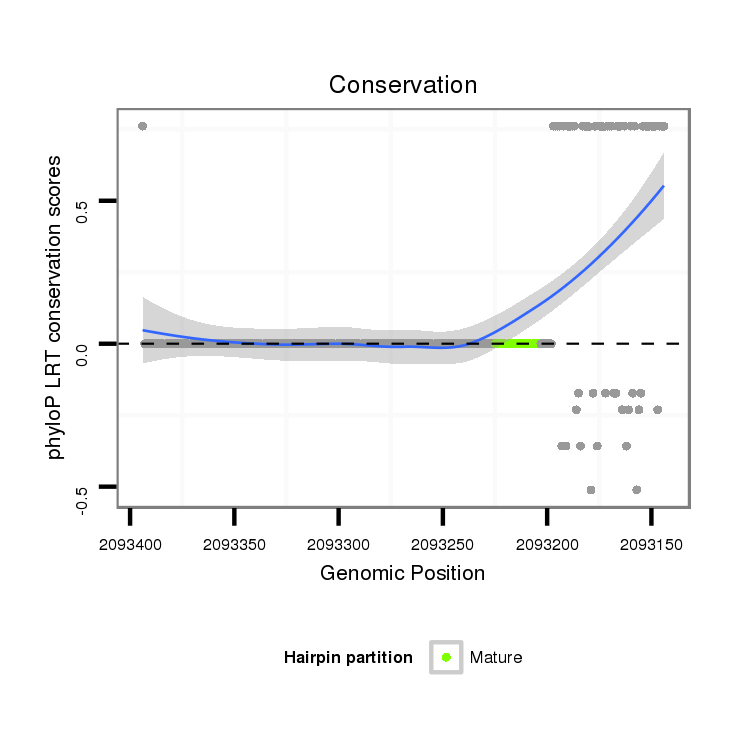
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12726:2093144-2093394 - | dvi_1996 | AGATTTCTTTACTACACTCAGACAAAGTTGTCACTGCTGGCTCTCAAGAGGTAAGGCAATTTTACAGTAAAGCTGACAGCTCATTCGATATTCGATATTGTCCATTACAGGAAATACAATTTAAATCAGATCCGTCACCAAAGACAACTGAGCAAGACATAGCCGATGGTAAAAATGCAGAACCTGAAAACCTTGATAAAAGACAAAAAGTTACGCCAAAGCTAAATGAAAATAAGACAGTTGATGAAAAA |
| dm3 | chr3R:313698-313752 + | A----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAAATAAAAAATAGTACGGTACAGCCAAACAAATAGTAAAGTATTGATGATAAA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
Generated: 05/17/2015 at 04:43 AM