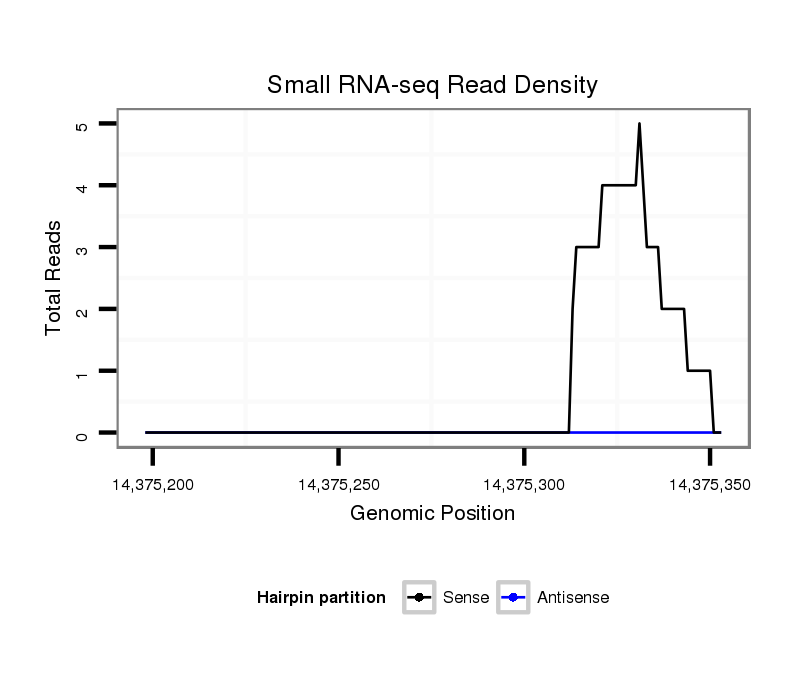
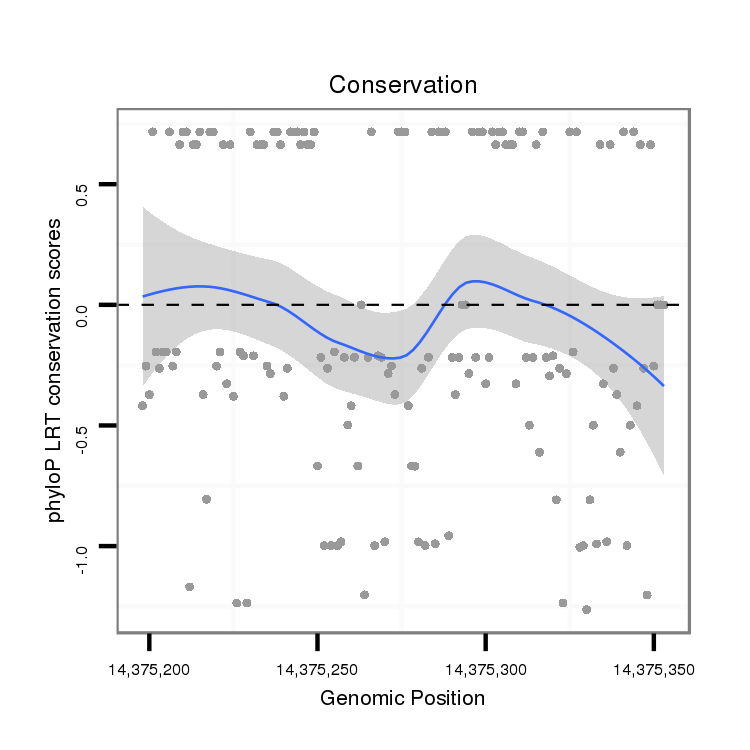
ID:dvi_19938 |
Coordinate:scaffold_13049:14375248-14375303 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_13444:2]; CDS [Dvir\GJ13561-cds]; CDS [Dvir\GJ13561-cds]; exon [dvir_GLEANR_13444:3]; intron [Dvir\GJ13561-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------################################################## TACTGAGCTTGGATTCCAGCTATGCCCAGGTGAAGGGTATACTAATAGAGGTCTGTGCGGGTTTCGGTAGTTCTAGTTAACGCTGAAAATAATCTAATATTTGCAGAAGGGCATAATGCAAGAGTCTAGTTCGGATGCCGAGCAGAATGTCGAGTA **************************************************.....(((((((((((((.(((......)))))))))...........))))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060678 9x140_testes_total |
SRR060670 9_testes_total |
SRR060657 140_testes_total |
SRR060659 Argentina_testes_total |
SRR060679 140x9_testes_total |
GSM1528803 follicle cells |
SRR060658 140_ovaries_total |
SRR060669 160x9_females_carcasses_total |
SRR060681 Argx9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................................................................................................................GTCTAGTTCGGATGCCGAGCAGA.......... | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................GATGCCGAGCAGAATGTCGA... | 20 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................ATGCAAGAGTCTAGTTCGG...................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................TGCAAGAGTCTAGTTCGGATGCC................. | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................ATGCAAGAGTCTAGTTCGGA..................... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................GTCTGTGCGGGCTTCAGTAG...................................................................................... | 20 | 2 | 3 | 0.67 | 2 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................AGAGTGTAGTTCGGCTGCAGA............... | 21 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....GAGCTTGGATTCCAACAA...................................................................................................................................... | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................CGGGTTTCCGTAGTT.................................................................................... | 15 | 1 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................................TAAGAGGTGTCTGTGCGGGT.............................................................................................. | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......................TGCCCAGGTGAAGAGTCTC................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .....................................................TGTGCGGGCTTCGGTAGAA.................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
ATGACTCGAACCTAAGGTCGATACGGGTCCACTTCCCATATGATTATCTCCAGACACGCCCAAAGCCATCAAGATCAATTGCGACTTTTATTAGATTATAAACGTCTTCCCGTATTACGTTCTCAGATCAAGCCTACGGCTCGTCTTACAGCTCAT
**************************************************.....(((((((((((((.(((......)))))))))...........))))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M028 head |
M027 male body |
SRR060679 140x9_testes_total |
SRR060681 Argx9_testes_total |
|---|---|---|---|---|---|---|---|---|---|
| ..........................GTCCACTTCCCATTTGCTTACC............................................................................................................ | 22 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 |
| ...................................................................................................................TACGGTCTCAGAGCAAGC....................... | 18 | 2 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 |
| ..................................................................................................................GTACGGTCTCAGAGCAAGC....................... | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 1 | 1 |
| .........ACCTATGGTCGACACGAGT................................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 |
| .........ACCTAAGGTCGACACAAGT................................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:14375198-14375353 + | dvi_19938 | TACTGAGCTTGGATTCCAGCTATGCCCAGGTGAAGGGTATACTAATAGAGGTCTGTGCGGGTTTCGGTAGTTCTAGTTAACGCTGAAAATAATCTAATATTT-GCAGAAGGGCATAATGCAAGAGTCTAGTTCGGATGCCGAGCAGAATGTCGAGTA |
| droMoj3 | scaffold_6680:12917216-12917366 - | AGTTGTGCTCGGATCCCAAGTACGCACACGTCAAGGGCGTACTTATAGAGGTATTATCTAAGATA-ATATTTTCGATTATATTATAATATAGTTT--CATTTATTAGAAGGGAATACTGGAAGACAGCAGTGACCCAGATGTAGATCAAGATGG--- | |
| droGri2 | scaffold_15110:15843677-15843826 - | AATTAAATTTAGATGCCAAGTATACCCCAACAAGGGGTGTACGAATAGAGGTAAATATATGTAAA-CAAACAACAATTATATATATACATATATA--CAATT-GCAGAAGGGCATTAAGCATTGCTTCAATATTCAGGCAGAACAAAAAGTGGA--- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
Generated: 05/17/2015 at 04:59 AM