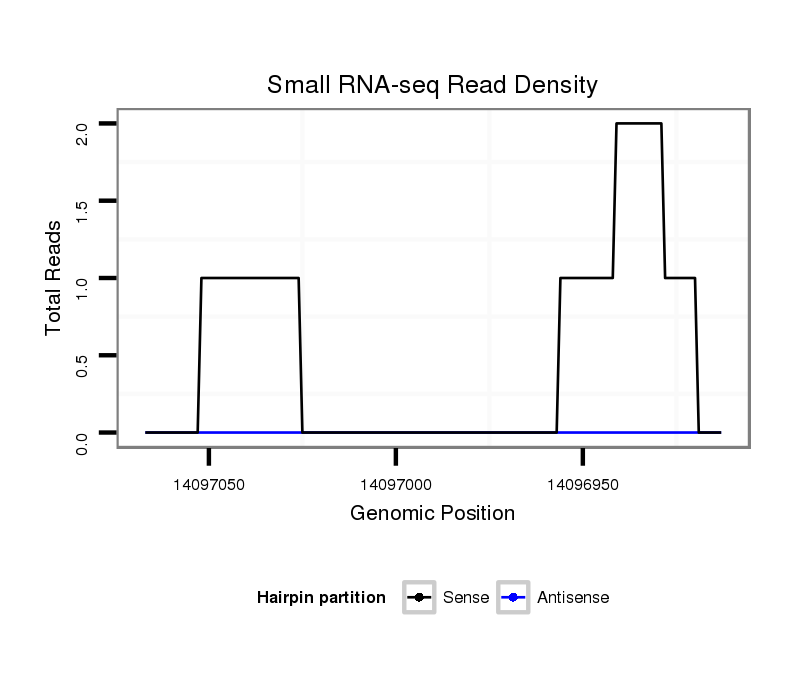
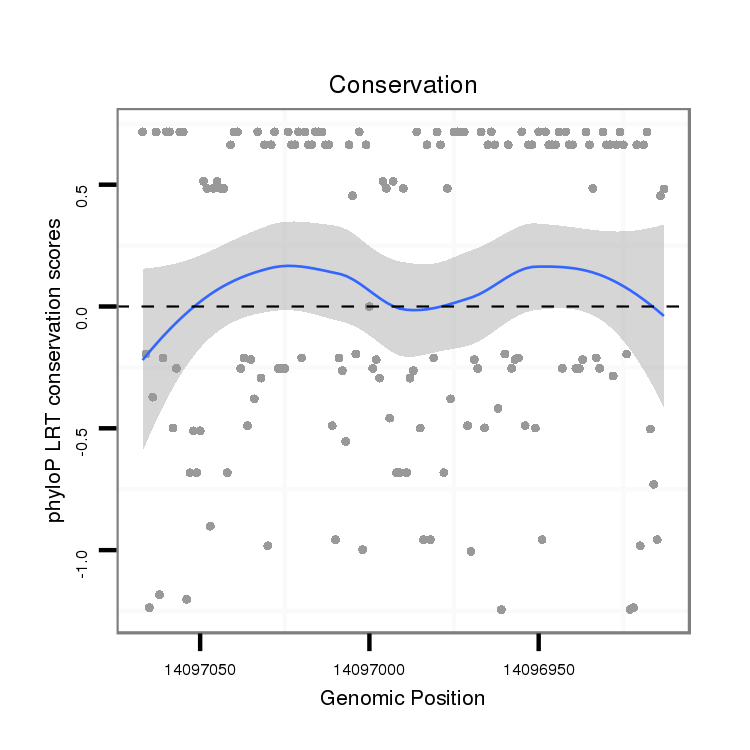
ID:dvi_19922 |
Coordinate:scaffold_13049:14096963-14097017 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_11821:2]; exon [dvir_GLEANR_11821:1]; CDS [Dvir\GJ11808-cds]; CDS [Dvir\GJ11808-cds]; intron [Dvir\GJ11808-in]
No Repeatable elements found
| ##################################################-------------------------------------------------------################################################## TGCCTATAAATATGCTGTTGCCAGCGCTTTTAATTGGGCTAATTGCATTGGTAAGCGTAAAGCGAGCGATCACTTGGCCCATATCTAAGGGTTATTATATTTCAGTGCCAATTGCCATATGGCAATGCTAGAGCTTTGGTCAGCCCCCCAGGGGT **************************************************......................((((((((........)))))).........))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060689 160x9_testes_total |
SRR060680 9xArg_testes_total |
V116 male body |
|---|---|---|---|---|---|---|---|---|
| ...............................................................................................................TTGCCATATGGCAATGCTAGAGCTTTGG................ | 28 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 |
| ..............................................................................................................................GCTAGAGCTTTGGTCAGCCCCC....... | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 |
| ...............TGTTGCCAGCGCTTTTAATTGGGCTAA................................................................................................................. | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 |
| .....................................................................................TAGGGGTTATTATCTATCA................................................... | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 |
|
ACGGATATTTATACGACAACGGTCGCGAAAATTAACCCGATTAACGTAACCATTCGCATTTCGCTCGCTAGTGAACCGGGTATAGATTCCCAATAATATAAAGTCACGGTTAACGGTATACCGTTACGATCTCGAAACCAGTCGGGGGGTCCCCA
**************************************************......................((((((((........)))))).........))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060669 160x9_females_carcasses_total |
M027 male body |
|---|---|---|---|---|---|---|---|
| .....................................................................AGTGAACCGAGTACAGATTC.................................................................. | 20 | 2 | 3 | 0.33 | 1 | 1 | 0 |
| .........................................................................................................................................CCAGGCGGGGCGTCCCCC | 18 | 3 | 15 | 0.07 | 1 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:14096913-14097067 - | dvi_19922 | TGCCTATAAATATGCTGTTGCCAGCGCTTTTAATTGGGCTAATTGCATTGGTAAGCGTAAAGCGAG------CGATCACTTGGCCCATATCTAAG-GGTTA------T-------TATATTTCAGTGCCAATTGCCATATGGC-AATGCTAGAGCTTTGGTCAGCCCCCCAGGGGT |
| droMoj3 | scaffold_6680:13208216-13208366 + | TGGTTCTAACCATA------------CTTCTCATTGGACTGGCTGCATTGGTAAGCGCATTGCGATAGTCATC-GTC--------CTTCCCCAAG---TTAGTCTTATTCGTGTATCGACTGCAGATCCGATTGCCCTGTGGCCAGTGCCGAAG-TCTGGCCAGCAGCTCAATCGT | |
| droGri2 | scaffold_15110:16101877-16102030 - | TATTTGCAAATATCAATATGGCAGCTCTTTCCTGTTGTCTAATTGCACTGGTAAGCCAGA-G-AAAAGTCTTC-AAAACCTTTCAAATAACAGAGATGGTA------T-------TCATTTTCAGACTCATCTCCCATTTGGC-AATGCTAGAGCCTTGGCCAGTGTCACA----- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
Generated: 05/17/2015 at 04:49 AM