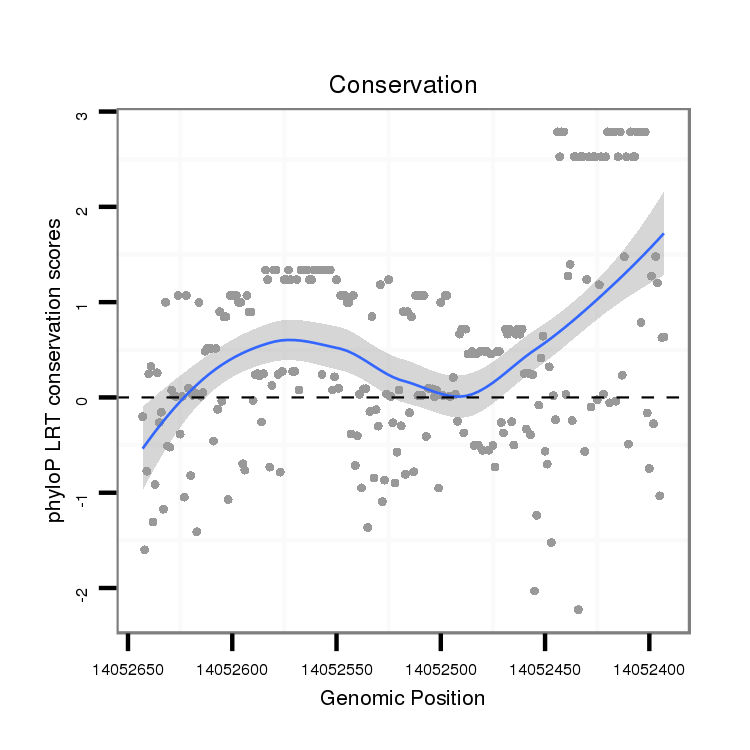
ID:dvi_19901 |
Coordinate:scaffold_13049:14052443-14052593 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_11823:7]; CDS [Dvir\GJ11810-cds]; intron [Dvir\GJ11810-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TGCCTTGCCGCCTTGTCAGGTATGTTGCCTGAAACTGTTGGCTAATGGATATATGTATATGAATATCTGCACTTGCTAATGGAAATTTAATGGCAATTGCTTAGAGTTTCCTCTATCTGCTCCGTACAAGCAATAATATAGCTCATACGACCCACTGGATGTATATTGGATATCTGATTGTGTTCCCTCTATTTTTGACAGATACACGGCGCTAGGCCCGCAGTCAACCTGACCTGGTATAACACCACTTC **************************************************..(((((((((((..((((((.((((((.(((....))).)))))).)))..)))..)))............((((..(((.........)))..)))).((....)))))))))).((((((......))))))................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060682 9x140_0-2h_embryos_total |
SRR060663 160_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
V116 male body |
SRR060686 Argx9_0-2h_embryos_total |
SRR060689 160x9_testes_total |
SRR060661 160x9_0-2h_embryos_total |
M027 male body |
SRR060679 140x9_testes_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060658 140_ovaries_total |
SRR060659 Argentina_testes_total |
SRR060657 140_testes_total |
SRR060680 9xArg_testes_total |
SRR060681 Argx9_testes_total |
SRR060666 160_males_carcasses_total |
SRR060664 9_males_carcasses_total |
SRR060665 9_females_carcasses_total |
SRR060676 9xArg_ovaries_total |
SRR060668 160x9_males_carcasses_total |
SRR060669 160x9_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ........................TTGCCTGAAACGGTTGGGTG............................................................................................................................................................................................................... | 20 | 3 | 6 | 6.00 | 36 | 5 | 8 | 3 | 1 | 2 | 3 | 3 | 1 | 0 | 3 | 3 | 2 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................TTGCCTGAAACGGTTGGGT................................................................................................................................................................................................................ | 19 | 2 | 3 | 6.00 | 18 | 1 | 1 | 4 | 5 | 3 | 2 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................TTGCCTGAAACGGTTGGG................................................................................................................................................................................................................. | 18 | 2 | 5 | 2.20 | 11 | 3 | 2 | 1 | 1 | 2 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................TGCCTGAAACGGTTGGGTG............................................................................................................................................................................................................... | 19 | 3 | 19 | 1.95 | 37 | 8 | 7 | 4 | 0 | 5 | 3 | 2 | 4 | 0 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................TGCCTGAAACGGTTGGGT................................................................................................................................................................................................................ | 18 | 2 | 6 | 1.67 | 10 | 2 | 0 | 1 | 1 | 1 | 1 | 1 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....TAGCCGCCTTGTCTGGTA..................................................................................................................................................................................................................................... | 18 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................GCAAATGATTAGAGGTTCC............................................................................................................................................ | 19 | 3 | 17 | 0.41 | 7 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 |
| ............................................................................................GCGAATGATTAGAGTTTCC............................................................................................................................................ | 19 | 3 | 20 | 0.35 | 7 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 2 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 |
| .........................................................................................................................................................................................................................CCGCAGCCAACCGGACCTG............... | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................ATCTGGTAGTGTTCCCTC.............................................................. | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................TGCCGGAAACTGTAGGCAAA.............................................................................................................................................................................................................. | 20 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........TCCTTGTCAGGTAAGTTCC............................................................................................................................................................................................................................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................ATTCCTCTTCCTGCTCCGT.............................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
ACGGAACGGCGGAACAGTCCATACAACGGACTTTGACAACCGATTACCTATATACATATACTTATAGACGTGAACGATTACCTTTAAATTACCGTTAACGAATCTCAAAGGAGATAGACGAGGCATGTTCGTTATTATATCGAGTATGCTGGGTGACCTACATATAACCTATAGACTAACACAAGGGAGATAAAAACTGTCTATGTGCCGCGATCCGGGCGTCAGTTGGACTGGACCATATTGTGGTGAAG
**************************************************..(((((((((((..((((((.((((((.(((....))).)))))).)))..)))..)))............((((..(((.........)))..)))).((....)))))))))).((((((......))))))................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060674 9x140_ovaries_total |
V053 head |
SRR060676 9xArg_ovaries_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060675 140x9_ovaries_total |
M047 female body |
SRR1106727 larvae |
SRR060677 Argx9_ovaries_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060679 140x9_testes_total |
SRR060669 160x9_females_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060678 9x140_testes_total |
V116 male body |
SRR060658 140_ovaries_total |
SRR060681 Argx9_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060688 160_ovaries_total |
SRR1106728 larvae |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................................................................................................CGTGATTATATAGAGTATGAT..................................................................................................... | 21 | 3 | 7 | 6.14 | 43 | 11 | 0 | 6 | 6 | 3 | 3 | 0 | 2 | 2 | 2 | 1 | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | 0 |
| .........................................................................................................................................................................................................................GGCGTCAGTGTGACTGGGC............... | 19 | 3 | 9 | 0.56 | 5 | 0 | 5 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................GTGATTATATAGAGTATGAT..................................................................................................... | 20 | 3 | 20 | 0.40 | 8 | 1 | 0 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 2 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................TTACCGAATCTCAAAAGAGATT....................................................................................................................................... | 22 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................CGTTAAGGAATCTCAAAGTGG.......................................................................................................................................... | 21 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .CGGAAGGGTGGAACAGTC........................................................................................................................................................................................................................................ | 18 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................GAACAGGCATGGTCGTTATT................................................................................................................... | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................................GGCGTCAGTGTGACTGGTC............... | 19 | 3 | 18 | 0.11 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................GTAAGAATCTCAAAGGAGAT........................................................................................................................................ | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................GAGGGAATCTCAAAGGAGA......................................................................................................................................... | 19 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..........................................ATTACCAATATACATA................................................................................................................................................................................................. | 16 | 1 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................................................................................................................................................................CCGGGGGTCAGTGGGTCTG.................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:14052393-14052643 - | dvi_19901 | TGCCTTGCCGCC----------------------TTGTCAGGTATGT--TGCCTGAAACTGTTG-GCTAATGGATATAT----------------------------GT----------------ATATGAAT--ATCTGCACTTG----CTAATGGAAATTTAATGGCAATTGCTTAGAGTTTCCTCTA--TCT-----------------GCTCCG---------------------------TACAAGC---AATAATATAGCTCATACGACCCACTGG------------ATGTATATTGGATA-TCTGATTGTGTTCCCTCTAT---TTTTGACAGATACACGGCGCTAGGCCCGCAGTCAACCTGACCTGGTATAACACCACTTC-- |
| droMoj3 | scaffold_6680:13259959-13260253 + | TGCCTAGCTAGCTGGTAGCTACCTGGCTGACAGTCAGGTAAAAATTGTTCCCCC------------GTAATGGCAATAT----------------------------AT----------------GCATGTAT--ATGCGCACCAG----CTAATGGAAATTTAATGGCAATTGCCTCGACTTGTCGCCA--AGTGGCGCCCACCCGCCGCTGCTGAGCAGC-----------------------------G---AATCACATAACGCATACGCCCCATTGGGCAGCGCTGTACACGCAAATAGACTAAATTGACGGTGTTTTCTTTACCACTACACACAGATACACGGCGCACGGCCCGCAGTCAATCTAACCTGGTATAATTCAACTTC-- | |
| droGri2 | scaffold_15110:16148725-16148988 + | TGCCTGCTTGGCTGCTTGCCAAC-----------TAGGTAGACATTTTTTACCTGAAATTGTTGGGCTAATGGAAA------------------------------TGT----------------GTATGAAT--ATGTGCACTTGCCAGCTAATGGAAATTTAATGGCAATTGCTTATAGTTC----------------------ATGACAGCCCCGCCGC--A--------AAGT-ACCACCCCAAAGGC---AGTCACATAACACGTACGCCCCATT-----------------------------TTTGATTGTGTTTTATTATT---TTGTGACAGATACATGGCGCCCGTCCGGCAGTTAACCTAACCTGGTATAATGCAACATC-- | |
| droWil2 | scf2_1100000004762:4599023-4599331 + | CTACTGTCTGCC--------AA---GG---TACTATAGTAAAAACGT--TTCAT---------GGGGTAATGGGCATACATGTACATATATATAAATGAATATGTATGCCCTTATCTATATGTTTGTGTGTGTGAATATGCACTTGACGTTTAATGGAAATTTAATAGCGATTGCCTTTCTAAACCAATATCGACAGTATTTGCCCATGGCAGGAAAATAGA--A--------AAGTTGTTAATTTATAGGTTTAAATCATGATATGCATAATCAT--------------------------------------------CATTTCGAT---TTTCAACAGATTCATGGTGCACGCCCAGCGGTTAATCTAACTTGGTATAATGCCACATC-- | |
| dp5 | XR_group6:2801649-2801865 - | TGCCTCT-----------------------------------------------------GTC-----------------GGGATAGAGATAGAAAGGCACAGAAATGT--GTAGTTAAACGGAGGCATGGGT--AAATGCACTTGTTGTTTAATGGAAATATAATAAC--------------------ACACACAGGAGTGG---AGGGCGGGTAAATGGGCAAATGTCGACAAGTTGTGAA------------------------------CAG--------------------------------------------TTTTCAATT---CTCCAATAGATTCACGGTGCTCGCCCGGCTGTTAACCTCACGTGGTACAACACAACCTC-- | |
| droPer2 | scaffold_9:1097394-1097610 - | TGCCTCT-----------------------------------------------------ATC-----------------GGTATAGAGATAGAAAGGTACAGAAATGT--GTAGTTAAACGGAGGCATGGGT--AAATGCACTTGTTGTTTAATGGAAATATAATAAC--------------------ACACACAGGAGTAG---AGGGCGGGTAAATGGGCAAATGTCGACAAGTTGTGAA------------------------------CAG--------------------------------------------TTTTCCATT---CTCCAATAGATTCACGGTGCTCGCCCGGCTGTTAACCTCACGTGGTACAACACAACCTC-- | |
| droAna3 | scaffold_13337:22761727-22761788 + | TAT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAATAACAGATTCACGGAGCGCGGCCACCAGTTAACCTTACTTGGTATAACATTACATC-- | |
| droBip1 | scf7180000396426:88009-88069 - | AT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAATTACAGATTCATGGAGCTCGGCCACCAGTAAACCTTACTTGGTATAACGTTACAGC-- | |
| droKik1 | scf7180000301734:36498-36569 - | TGCTTTACCA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTT---TCTTTTAAGATTCACGGGGCTCGCCCAGCTGTTAACCTTACTTGGTATAACTCAACATC-- | |
| droFic1 | scf7180000453155:43219-43285 + | TTTCC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTT---ACTTTATAGATCCACGGGGCTCGCCCAGCTGTTAATCTTACTTGGTATAATTCGACTTC-- | |
| droEle1 | scf7180000487448:35724-35784 - | TCC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATTTTCAGATTTACGGCGCTCGCCCAGCTGTTAATCTTTCATGGTATAATTCAACTAA-- | |
| droRho1 | scf7180000776885:7855-7913 + | TAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTTAGATTCACGGCGCTCGCCCAGCTGTAAATCTTACATGGTATAATTCAACTAA-- | |
| droBia1 | scf7180000302377:85466-85533 - | CTCTGT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATT---TCCTAACAGATTCACGGCGCACGACCAGCTGTTAACCTTACGTGGTATAATTCAACTAC-- | |
| droTak1 | scf7180000412753:5424-5493 + | CTTTTT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATT---TCATACTAGATCCAAGGAGCTCGACCAGCTGTTAACCTTACGTGGTATAATTCATCAATCT | |
| droEug1 | scf7180000408944:17777-17839 + | CTA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T---TTTTTTTAGATTCACGGGGCTCGACCAGCTGTTAATCTTACATGGTACAATTCCAGTAC-- | |
| dm3 | chr3L:23005812-23005872 + | AC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTTTATAGATTCACGGCGCTCGACCGGCAGTAAATCTTACTTGGTATAATACAACCAC-- | |
| droSim2 | 3l:22490970-22491030 + | AC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTTCATAGATTCACGGAGCTCGACCGGCAGTAAATCTTACTTGGTATAATACAACCAC-- | |
| droSec2 | scaffold_41:27619-27679 + | AC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTTTATAGATTCACGGAGCTCGACCGGCAGTAAATCTTACTTGGTATAATACAACCAC-- | |
| droYak3 | v2_chrUn_3514:11526-11585 + | TTT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTATAGATTCGTGGGGCGCGACCAGCAGTAAATCTTACTTGGTATAACACAACCAC-- | |
| droEre2 | scaffold_4784:22660063-22660130 + | TCTTTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAC---TTTTAATAGATCCGCGGGGCTCGACCAGCAGTAAATCTTACTTGGTATAACACAACTAC-- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 04:44 AM