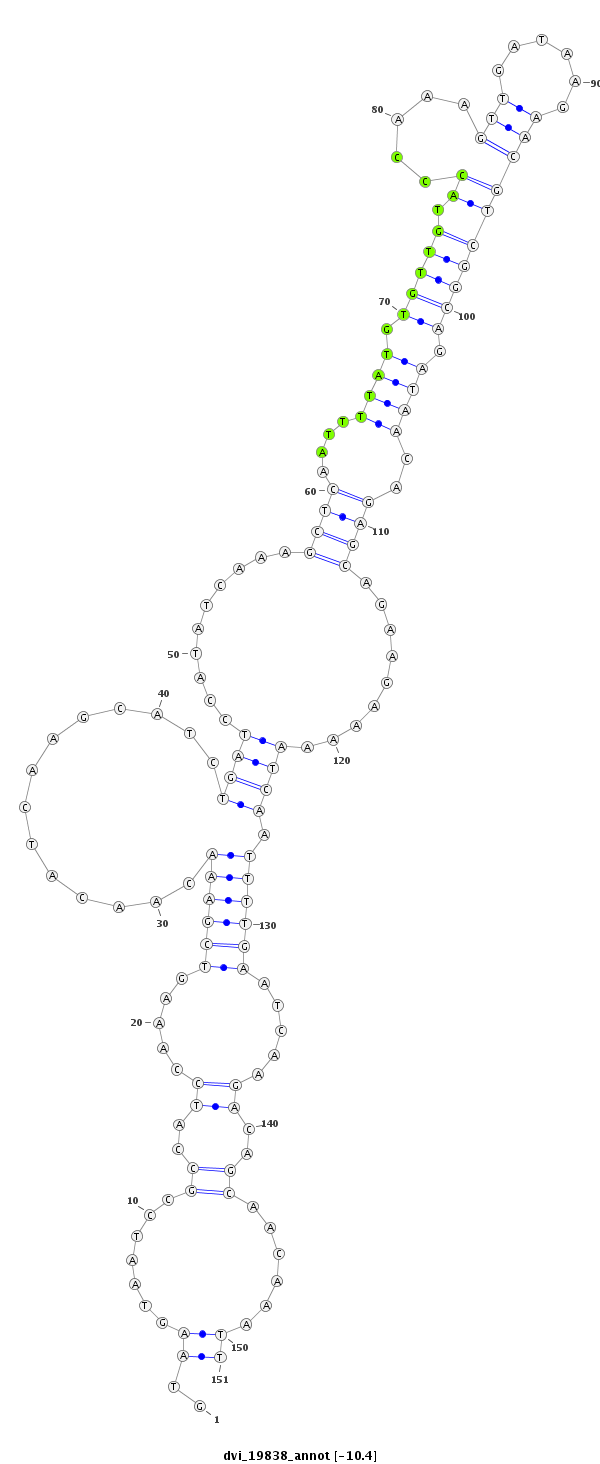
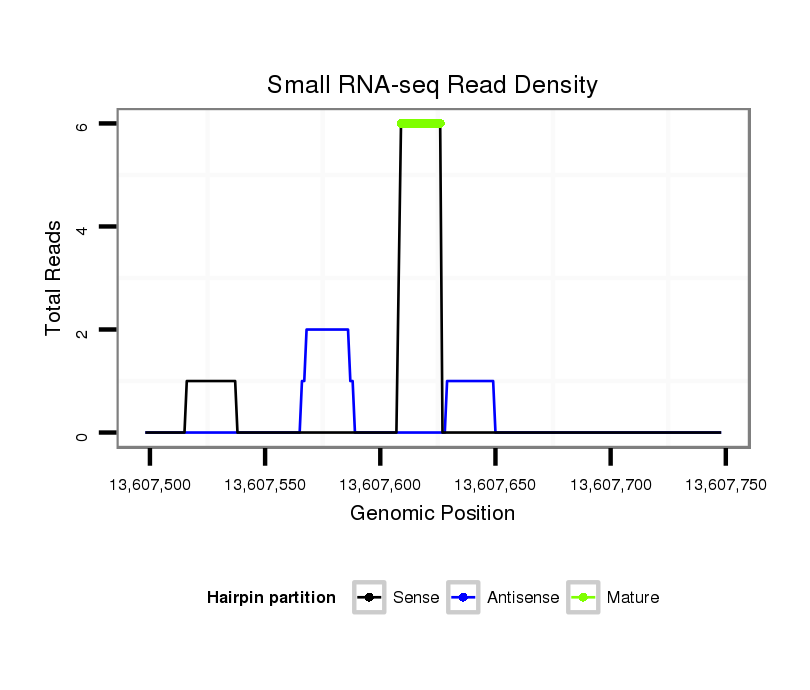
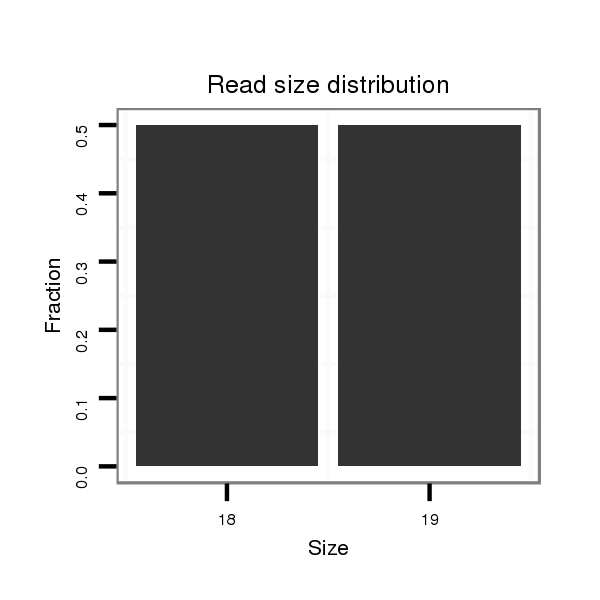
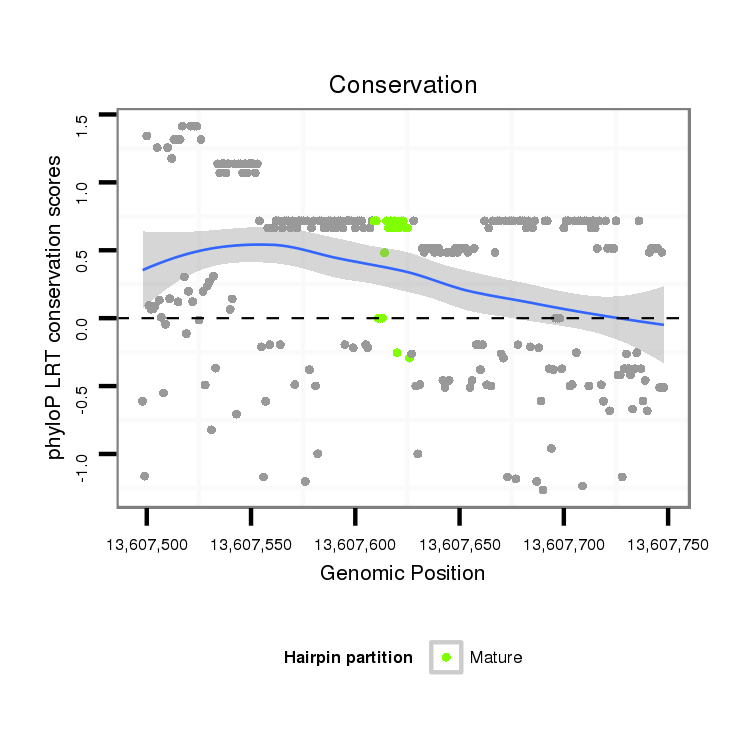
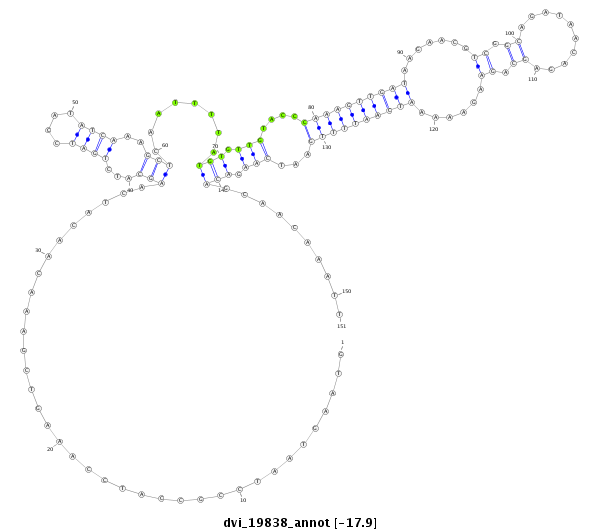
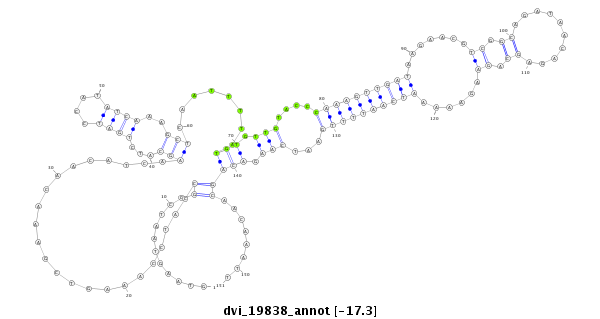
ID:dvi_19838 |
Coordinate:scaffold_13049:13607548-13607698 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -17.9 | -17.3 | -17.1 | -17.1 |
 |
 |
 |
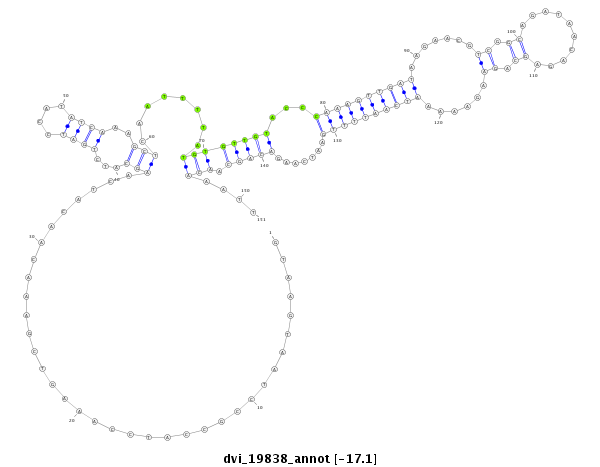 |
exon [dvir_GLEANR_13421:1]; CDS [Dvir\GJ13538-cds]; intron [Dvir\GJ13538-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- GCCCAAAATGAATCCCGCCAAACACATCCCTAACATTGATGATTTAATGTGTAAGTAATCCGCCATCCAAAGTCGAAACAACATCAAGCATCTGATCCATATCAAAGCTCAATTTTATGTGTTGTACCCAAAGTTGATAAGAACGTCGGCAGATAACAGAGCAGAAGAAAAATCAATTTTGAATCAAGACAGCAACAAATTGGAATTTTTTGTTTGTCTAACAACTAATAAGTGTGCAAGGCACATATACT **************************************************..((.......((..((.....((((((..............((((..........((((....((((.(((((.((.....(((......)))))))))).))))..)))).........)))).)))))).....))..))......))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M061 embryo |
SRR060671 9x160_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060667 160_females_carcasses_total |
V047 embryo |
SRR060686 Argx9_0-2h_embryos_total |
SRR060666 160_males_carcasses_total |
SRR060687 9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................................................................ATTTTATGTGTTGTACCC.......................................................................................................................... | 18 | 0 | 1 | 3.00 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................AATTTTATGTGTTGTACCC.......................................................................................................................... | 19 | 0 | 1 | 3.00 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................AGTTTATGTGTTGTACCC.......................................................................................................................... | 18 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................CAAACACATCCCTAACATTGAT................................................................................................................................................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................CCCTAACATTGATGATTTAATGTCGAT..................................................................................................................................................................................................... | 27 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................AAAATCGATTTAGAATCAAGA.............................................................. | 21 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................AACAAGTTGGAATTATTTGT...................................... | 20 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .........................................................................................................................................................TAACATAGCAGATGAAAAA............................................................................... | 19 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .............................................................................................................................................................AGCGCAGGAGAAAAATCA............................................................................ | 18 | 2 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................................................................................................................AAAGGTGAAAAGAACCTCG....................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .............................................................................................................AAATTTTATGTGTTGTACCC.......................................................................................................................... | 20 | 1 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
CGGGTTTTACTTAGGGCGGTTTGTGTAGGGATTGTAACTACTAAATTACACATTCATTAGGCGGTAGGTTTCAGCTTTGTTGTAGTTCGTAGACTAGGTATAGTTTCGAGTTAAAATACACAACATGGGTTTCAACTATTCTTGCAGCCGTCTATTGTCTCGTCTTCTTTTTAGTTAAAACTTAGTTCTGTCGTTGTTTAACCTTAAAAAACAAACAGATTGTTGATTATTCACACGTTCCGTGTATATGA
**************************************************..((.......((..((.....((((((..............((((..........((((....((((.(((((.((.....(((......)))))))))).))))..)))).........)))).)))))).....))..))......))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060656 9x160_ovaries_total |
SRR060676 9xArg_ovaries_total |
SRR060678 9x140_testes_total |
SRR060683 160_testes_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060657 140_testes_total |
SRR1106722 embryo_10-12h |
GSM1528803 follicle cells |
V053 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...................................................................................TGTTCGTAGACTAGGTAT...................................................................................................................................................... | 18 | 1 | 2 | 1.50 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................TTTCAGCTTTGTTGTAGTTCG.................................................................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................TCAACTATTCTTGCAGCCGTC................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................TCAGCTTTGTTGTAGTTCGTA................................................................................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................TTTAGTGAAAACCTAGTTCTG............................................................. | 21 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................CCCTGTTGTAGTTCGTAG............................................................................................................................................................... | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................TTGTGTAGGCATTGTGAATA................................................................................................................................................................................................................... | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................................................................TTTTTTTCGTAGACTAGGTAT...................................................................................................................................................... | 21 | 3 | 15 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................CTTTTTAGTTGAAACTTCGC................................................................. | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...............................................................................................................................................GCAGCCGTCTCTTATC............................................................................................ | 16 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:13607498-13607748 + | dvi_19838 | GCCCAA---AATGAATCCCGCCAAACACATCCCTAACATTGATGATTTAATGTGTAAGTAATCCG-CCATCCAAAGTCGAA--------------------------AC-------AACATCAAGC--------ATCTGATCCATATCAAAGCTCAATTTTATGTG--TTGTACCCAAAGTTGATAAGAACGTCGGCAGATAACAGAGCAGAAGAAAAATCAATTTTGAATCAAG-ACAGCAACAAATTGGAATTTTTTGTTTGTCTAACAAC------------TAATAAGTGTGCAAGGCACATATACT- |
| droMoj3 | scaffold_6680:13732805-13733026 - | GCC---------------CGCCAAACACATCCCTAATATTGATGATTTGATGTGTAAGTAACGCG-CCATCCAAAGTCGAATACATACATTATATGCATATTATGGAAT-------AACAGAAAGC--------ATCTGATCCATATCAAAGCTCAAT---ATGTGTTTCGTACCCTACTG--------------------------GCAGACGCAAAAACAGTTTGGAATCAAGTATACGAATTA---AGAAGGTCTTCTTGGTC-ACGA----------------AATGAAAATTGAAC----------A | |
| droGri2 | scaffold_15110:19029851-19030137 + | ATCCCATCTGATGGATCCCGCCAAACACATTCCGAATAGTGATGATTTGATGTGTAAGTAGGCCATCCATTCAAAGTGGAAAA---ATTTTATAT------------AGAACACGAACCATTAAGCTGAATCTGATCTGATCTATAACAAAGTACAAT----TGTGC-TTGTACCAAAAAGTGATAAGAACAACAGCAGATAACTAAATCAACGAA-AATAACTTTCAAATCAGG-AGTGAAATGC---AGAATGTTTTATTTGTCTACCAAAGATAACAAAATCTAAATCATAATTAAAGTCCATATTCAA | |
| droWil2 | scf2_1100000004762:4115251-4115285 - | GGCAAC---AAACATTTCCGGCAACAACATTCACATCA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000302193:5679223-5679275 - | CG---A---AATGTTTCCCGCCATTCAGATTCCCCAGGGTGATGAGCTCATGTGTAAGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dm3 | chr3R:25554240-25554244 + | ACCCA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
Generated: 05/17/2015 at 03:46 AM