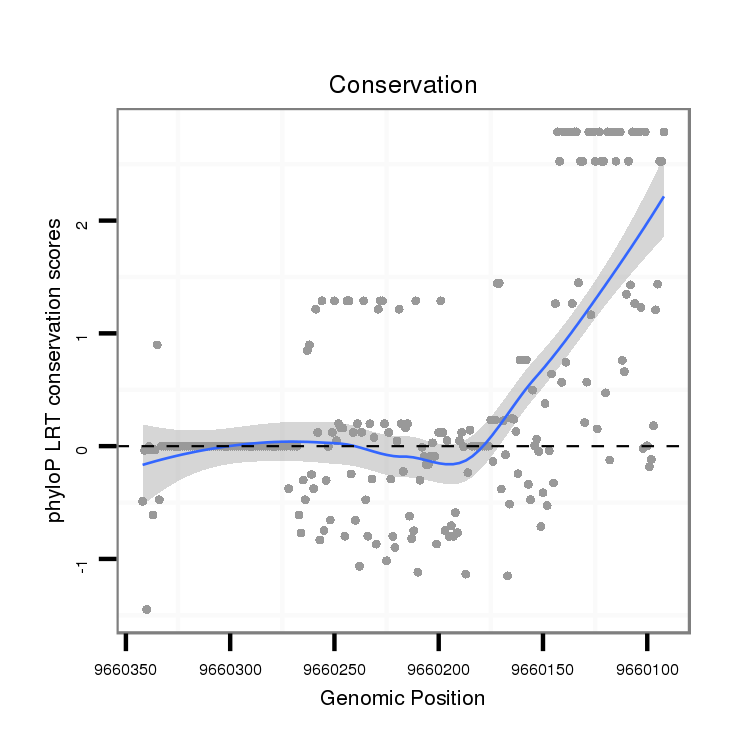
ID:dvi_19223 |
Coordinate:scaffold_13049:9660142-9660292 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ12070-cds]; exon [dvir_GLEANR_12066:2]; intron [Dvir\GJ12070-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## AACCAATTATAAATCCTCTTGCCGTAAATCGCATTTGAAAATAAGCCACAGAATTTCCAATCAAACGCTTAGCTCAATAGATTGTGAGTCGAAAAGGATTGTGAAAATAAAGCGAAAAATTGAGAAGAAAATCGCATAATAGAAATATGTTCTAAAACTAGAAGCTCTTAATCTTGATTAGATTTACTTTTAATATTGCAGCTCTACTTTGGCCTGTCCACGTTTATGTTTACGTTCTATCTAAAGGAGGA **************************************************.................((..((......((..(((((((....(((((((.((.......((.........((((.....((........))......))))..........)))).))))))).....)))))))..))......))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
GSM1528803 follicle cells |
SRR060684 140x9_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................................................................................................CCTGTCATCGTTTATGTTTCC.................. | 21 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 |
| ...................................................................................................................................................................................CGAGTTACTTTTAACATTGCA................................................... | 21 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 |
| ........................................................................................TCGAAAAGGATTCTGATAA................................................................................................................................................ | 19 | 2 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 |
| ................................................................................ATTCTGAGTGGAAAAGGA......................................................................................................................................................... | 18 | 2 | 8 | 0.13 | 1 | 0 | 0 | 1 | 0 |
| ........................................................................................TCGAAAAGGATTCTGATA................................................................................................................................................. | 18 | 2 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 |
| ....................................................................................................................................................................................................................CCTGTCACCGTTTATGTTTC................... | 20 | 3 | 15 | 0.07 | 1 | 1 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................CGGGTCCTCGTTTATGTTT.................... | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 |
| .....................................................................................................................AATTGAGAAGAAAATCTG.................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
|
TTGGTTAATATTTAGGAGAACGGCATTTAGCGTAAACTTTTATTCGGTGTCTTAAAGGTTAGTTTGCGAATCGAGTTATCTAACACTCAGCTTTTCCTAACACTTTTATTTCGCTTTTTAACTCTTCTTTTAGCGTATTATCTTTATACAAGATTTTGATCTTCGAGAATTAGAACTAATCTAAATGAAAATTATAACGTCGAGATGAAACCGGACAGGTGCAAATACAAATGCAAGATAGATTTCCTCCT
**************************************************.................((..((......((..(((((((....(((((((.((.......((.........((((.....((........))......))))..........)))).))))))).....)))))))..))......))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V116 male body |
M027 male body |
SRR060662 9x160_0-2h_embryos_total |
SRR060663 160_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
V053 head |
SRR060667 160_females_carcasses_total |
M047 female body |
SRR060666 160_males_carcasses_total |
SRR060689 160x9_testes_total |
SRR060665 9_females_carcasses_total |
SRR060669 160x9_females_carcasses_total |
V047 embryo |
SRR060679 140x9_testes_total |
SRR060671 9x160_males_carcasses_total |
M028 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................ATGGCTAGTTTGCGAATCGC................................................................................................................................................................................. | 20 | 3 | 1 | 7.00 | 7 | 2 | 1 | 1 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................TGGCTAGTTTGCGAATCGC................................................................................................................................................................................. | 19 | 3 | 7 | 2.57 | 18 | 5 | 2 | 4 | 2 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 1 | 1 | 1 | 0 | 0 | 0 |
| ......................................................ATGGTTAGTTTGCGAATCG.................................................................................................................................................................................. | 19 | 1 | 1 | 2.00 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................ATGGCTAGTTTGCGAATCG.................................................................................................................................................................................. | 19 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................TGGTTAGTTTGCGAATCG.................................................................................................................................................................................. | 18 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................TGGCTAGTTTGCGAATCG.................................................................................................................................................................................. | 18 | 2 | 5 | 1.00 | 5 | 1 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................CAGCATTTCCTAACACTTAAAT.............................................................................................................................................. | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................ACGGCTAGTTTGCGAATCGC................................................................................................................................................................................. | 20 | 3 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................TTAGTTTGCGACTCGCGATA............................................................................................................................................................................. | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........ATTAAGGTGAACGGCATT................................................................................................................................................................................................................................ | 18 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................GGGCTAGTTTGCGAATCGC................................................................................................................................................................................. | 19 | 3 | 17 | 0.12 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .....................................................GATGGCTAGTTTGCGAATC................................................................................................................................................................................... | 19 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................ACAAGAATTTCCTCTTCGAGA................................................................................... | 21 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ......................................................ATGGTTAGTTTGCGAATCGTA................................................................................................................................................................................ | 21 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:9660092-9660342 - | dvi_19223 | AACCAATTATAAATCCTCTTGCCGTAAATCGCATTTGAAAATAAGCCACAGAATTTCCAATCAAACGCTTAGCTCAATAGATTGTGAGTCGAAAAGGATTGTGAAAATA------AAGCGAAAAATTGAGAAGAAAATCGCATAA-TA----GAAATATGTTCTAA-----------------------------------------------------------------------------AACTAGAAGCTCTTAATCTTGATTAGATTT------------A-CTTT-TAATATTGCAGCTCTACTTTGGCCTGTCCACGTTTATGTTTACGTTCTATCTAAAGGAGGA |
| droMoj3 | scaffold_6680:11108275-11108502 + | AAAAACTTT-------------------------------------------------------------T----CTCTGACAGCAAACCAAAAAAATTTGCAACAAATTGCAGCAGCAGAAATGCATTGAAAATTCTGA--------ATATTGAGGAATAAATAAATTAC---CAATTTCTGAATTTAAATATGCACATTTCTTTATAATCTTTACCTTTCAACAATTACCTTTTTTCATCCAA------------------------------------CA-------------TTTTCAGATTTACTTTGGCCTGTCCACGTTAATGTTCACGTTCTATTTGAAAGAGGA | |
| droGri2 | scaffold_15110:17428194-17428283 - | TATA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATCTT---------TTAATTTGCATT---------------GCTG-ATTC-TCATCTTACAGATCTACTTTGGATTGTCCACGCTTATGTTTACGGTCTATCTGAAGGAGGA | |
| droWil2 | scf2_1100000004511:9249714-9249927 - | dwi_697 | CAAAGACTT-------------------------------------------------------------A----ACTTGACTGTCAACTTGAGAGGCTTATGATGAAC------AAGAGAAAGATGATGATGATGATGATGATGATGATGATAAAGGGAGGACGTGGTACCTTTTGTTTTTGGCCTAAAATT-----------------------------------------------CATGTC---------TTAATTT--CGTGA-------ATTTT-TCT-TTAC-CTTTCATTTTAGATTTATTTTGGCATCTCGACGCTGATGTTTTTGTTTTACTTAAAAGAGGA |
| dp5 | XR_group8:3466045-3466113 - | CTT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTA-TATACAATACAGATTTACTTTGGCTTCTCGACGTTAATGTTCCTGTTCTACCTGCGCGAGGA | |
| droPer2 | scaffold_65:270526-270603 - | TTA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCCT------------------------------------CCTT-TTTA-TATACAATACAGATTTACTTTGGCTTCTCGACGTTAATGTTCCTGTTCTACCTGCGCGAGGA | |
| droAna3 | scaffold_13337:12612838-12612892 - | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACAGGTTTACTTCGGGTTCTCGACGCTGATGTTTTTGTTCTACCTGCGCGAGGA | |
| droBip1 | scf7180000396589:1394819-1394873 + | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACAGGTTTACTTCGGATTCTCGACGCTGATGTTTTTGTTCTACCTGCGCGAGGA | |
| droKik1 | scf7180000302711:123956-124010 - | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCAGGTTTACTTTGGCTTCTCGACGCTTATGTTTTTGTTCTACCTGGTCGAGGA | |
| droFic1 | scf7180000453929:1706094-1706146 - | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGGTTTACTTTGGCTTCTCCACGCTGATGTTTTTGTTCTACGTGCGCCAGGA | |
| droEle1 | scf7180000491268:1420006-1420060 + | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCAGGTTTACTTTGGCTTCTCGACGCTGATGTTTTTGTTCTACCTGCTCGGGGA | |
| droRho1 | scf7180000779474:101840-101894 + | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACAGGTTTACTTTGGCTTCTCGACGCTGATGTTTTTGTTCTACCTGCGCGGGGA | |
| droBia1 | scf7180000302193:3412698-3412752 + | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACAGGTTTACTTTGGCTTCTCGACGCTGATGTTTTTGTTCTACTTGTTCGAGGA | |
| droTak1 | scf7180000415868:627998-628050 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGGTTTACTTTGGCTTCTCGACGCTGATGTTTTTGTTCTACCTGTTCGAGGA | |
| droEug1 | scf7180000409466:3671687-3671872 + | ATT--------------------------------------------------------------------------------GTCACTTGAAGGGGATTTTCGGAA-A------GGGGGAAGCACTTTGGGGGCTTTCGCAATA-CA-CAACAAATGTGTGCCATAA---TTTCCTTGTGTAGCCT--------------------------------------------------------------------TAAATGGTTTTTGTATTTTCT-----TCACA-ATTT-GTTCCACACAGGTTTACTTTGGCTTCTCAACGCTGATGTTTTTGTTCTACCTGTTCGAGGA | |
| dm3 | chr3L:5901679-5901733 + | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACAGGTTTACTTTGGCTTCTCGACGCTGATGTTTTTGTTCTACCTAATCGAGGA | |
| droSim2 | 3l:5814938-5815003 + | ACT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TT-TCCCCATACAGGTTTACTTTGGCTTCTCGACGCTGATGTTTTTGTTCTACCTAATCGAGGA | |
| droSec2 | scaffold_2:5837450-5837504 + | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACAGGTTTACTTTGGCTTCTCGACGCTGATGTTTTTGTTCTACCTAATCGAGGA | |
| droYak3 | 3L:6479131-6479185 + | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACAGGTTTACTTTGGCTTCTCGACGCTGATGTTTTTGTTCTACCTTATCGAGGA | |
| droEre2 | scaffold_4784:8598731-8598827 + | TTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TACGCC---------CGGATGTTCCTAGT-------ATTTT-TCA-CATTT-TTTCCGTACAGGTTTACTTTGGCTTCTCGACGCTGATGTTTTTGTTCTACCTAATCGAGGA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 09:52 PM