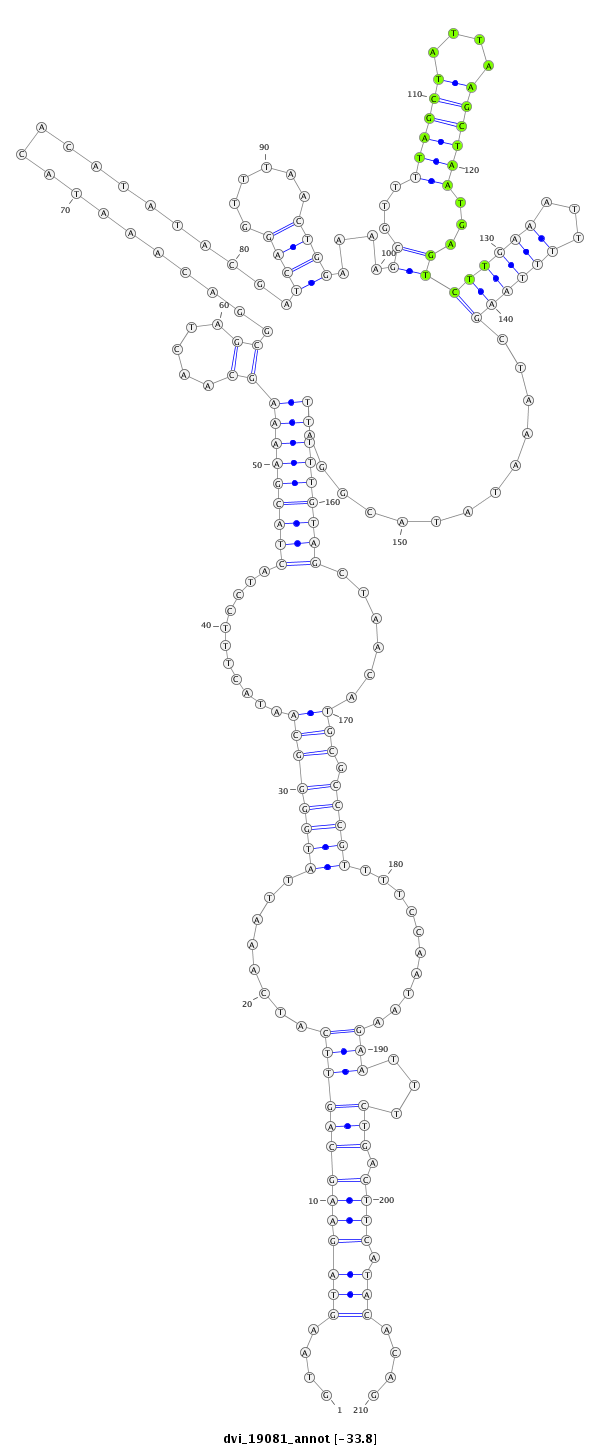
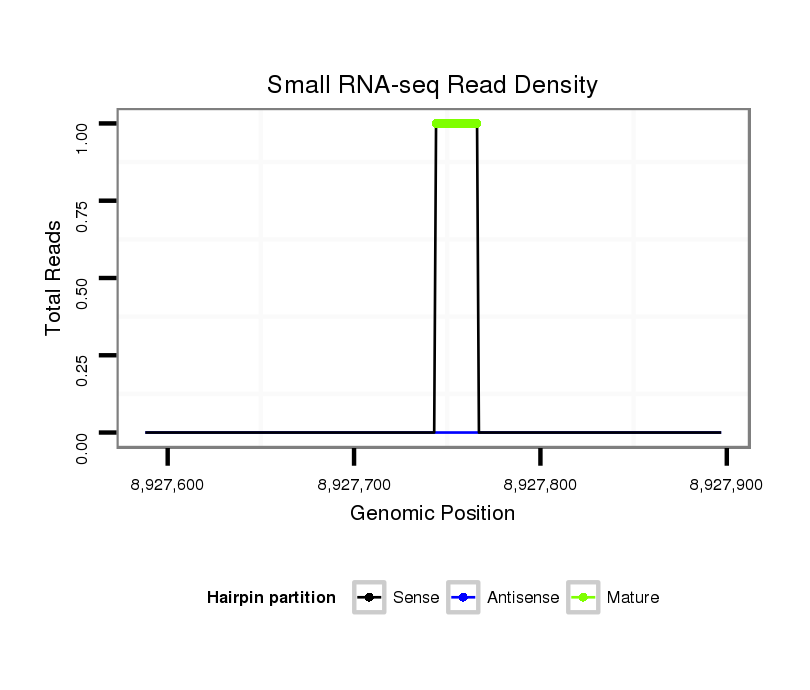

ID:dvi_19081 |
Coordinate:scaffold_13049:8927638-8927847 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -33.6 | -33.6 | -33.4 |
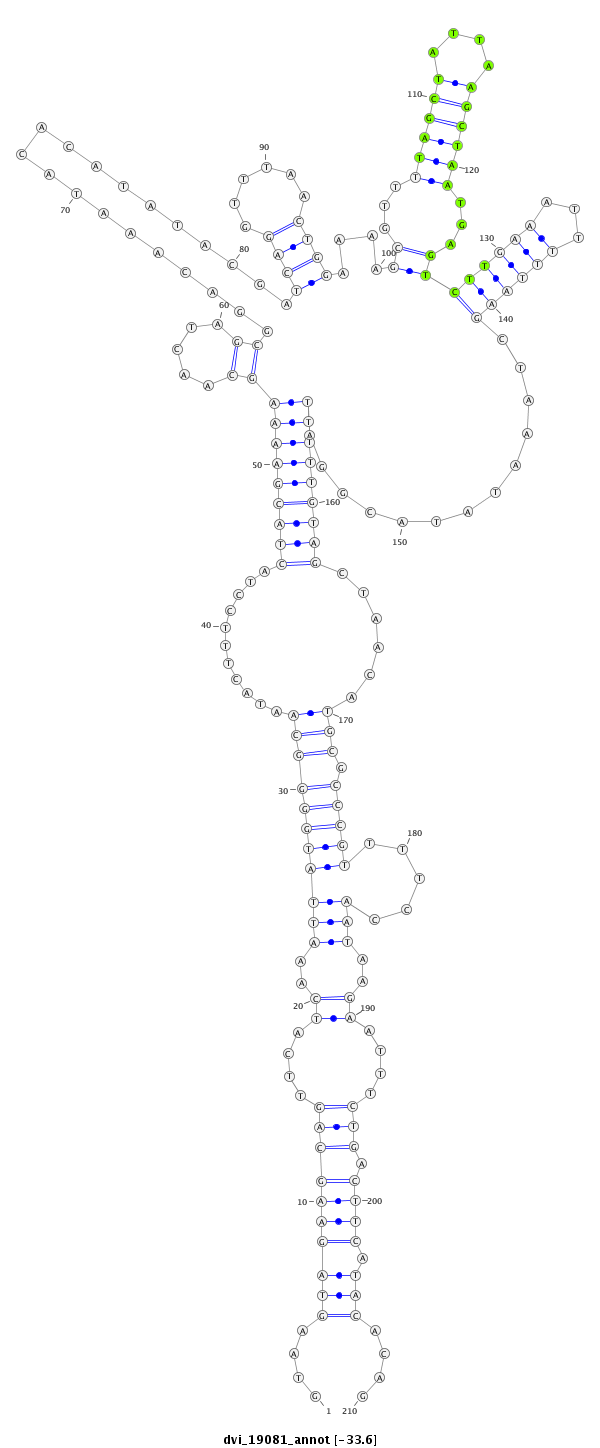 |
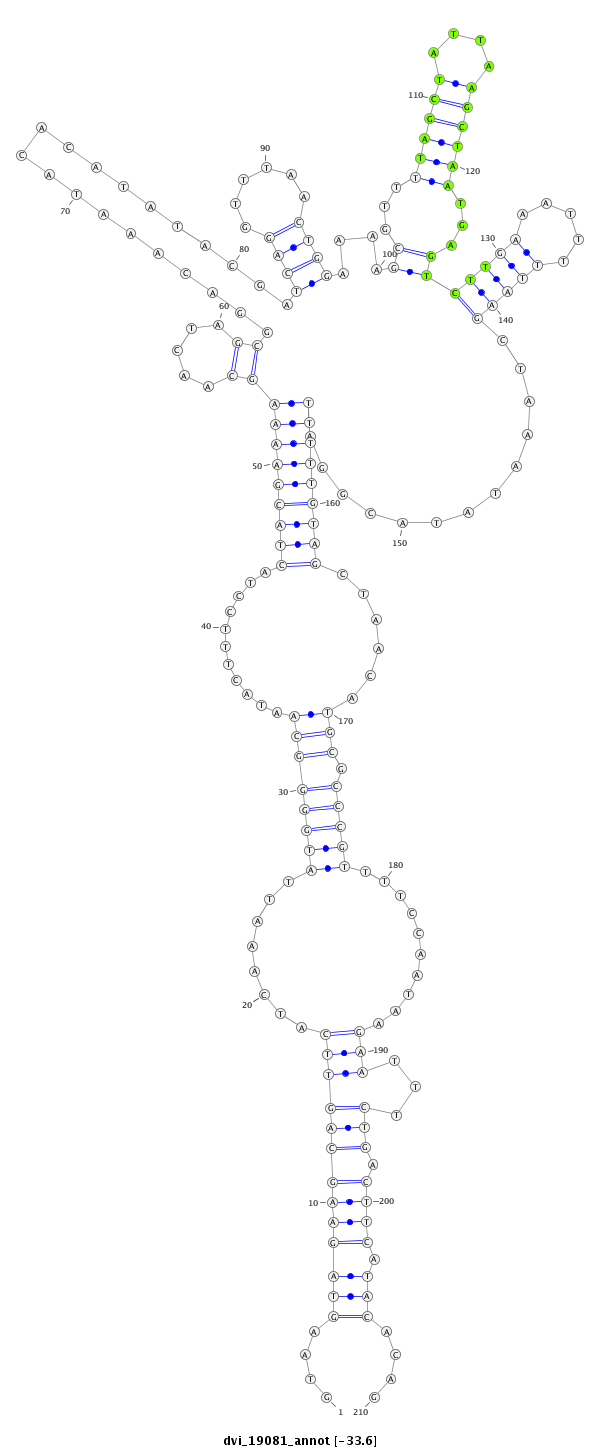 |
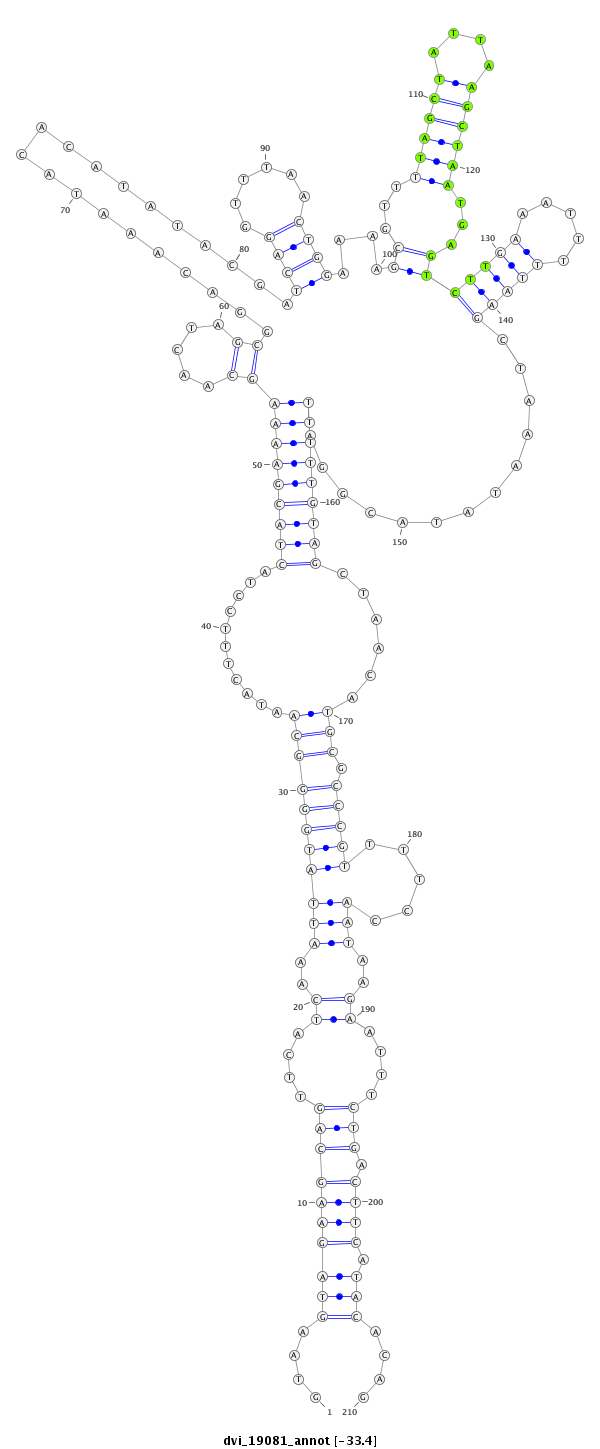 |
CDS [Dvir\GJ13236-cds]; exon [dvir_GLEANR_13141:1]; exon [dvir_GLEANR_13141:2]; CDS [Dvir\GJ13236-cds]; intron [Dvir\GJ13236-in]
No Repeatable elements found
| -----------------------------------------#########------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TGTCGCTGTGTGCGTGAGCGGGAGCGCCCGGAACTGAGCAAATGTCTGACGTAAGTAGAAGCAGTTCATCAAATTATGGGGCAATACTTTCCTACTACGAAAAGCAACTAGCGGACAAATACACATATACGATCAGGTTTAACTGGAAAAGCGTTTTAGCTATTAAGCTAATGAGTCTTGAAATTTTTAAGCTAAATATACGGATTTTTGTAGCTAACATGCGCCCGTTTTCCAATAAGAATTTCTGACTTCATACACAGGACGAGGCCAATCGGTTCGGTGGCAGCGCTGGCAGCTGCTCCCAACGTAT **************************************************....(((((((((((((........((((((((...........(((((((((((.....))....................((((......))))....((...((((((....))))))...))((((((...)))))).............)))))))))......))).)))))..........)))...))).)))).)))....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
SRR060679 140x9_testes_total |
SRR060687 9_0-2h_embryos_total |
M061 embryo |
SRR060666 160_males_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
SRR060681 Argx9_testes_total |
SRR060664 9_males_carcasses_total |
M047 female body |
SRR060665 9_females_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060671 9x160_males_carcasses_total |
M028 head |
SRR060668 160x9_males_carcasses_total |
SRR060689 160x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..................................................................................................GAAAACCAACTACCGGACGA................................................................................................................................................................................................ | 20 | 3 | 3 | 1.33 | 4 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ................................................ACGTAAGTGGAAGAAGATCAT................................................................................................................................................................................................................................................. | 21 | 3 | 2 | 1.00 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................TAGCTATTAAGCTAATGAGTCTT................................................................................................................................... | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................................................................................TGGCAGCACTGTCAGCTGCTCCG....... | 23 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................AATGTCTGACGCAGGTAGAA.......................................................................................................................................................................................................................................................... | 20 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................CGTAAGTGGAAGAAGATCAT................................................................................................................................................................................................................................................. | 20 | 3 | 4 | 1.00 | 4 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................ATGAAAACCAACTACCGGAC.................................................................................................................................................................................................. | 20 | 3 | 9 | 0.78 | 7 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 1 | 2 | 0 | 0 | 0 | 2 | 0 | 0 | 0 |
| ........................................AATGTCTGACGCAGGTAGAAA......................................................................................................................................................................................................................................................... | 21 | 3 | 4 | 0.75 | 3 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................TGTCTGACGCAGGTAGAA.......................................................................................................................................................................................................................................................... | 18 | 2 | 4 | 0.75 | 3 | 0 | 0 | 1 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................AATGTCTGACGCAGGTAGA........................................................................................................................................................................................................................................................... | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................................................................................TGGCAGCGCTGTCAGCTGCTCT........ | 22 | 2 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................................................................AGGACAATCGGTTATGTGGC.......................... | 20 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................CAATACTGTCCAACTACGA.................................................................................................................................................................................................................. | 19 | 2 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................ATGTCTGACGCAGGTAGA........................................................................................................................................................................................................................................................... | 18 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................GGTTTATCTGGAAAAGCCATT.......................................................................................................................................................... | 21 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................................................................................TGGCAGCGTGGGTAGCTGCTC......... | 21 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................AATGTCTGACGCAGGTAG............................................................................................................................................................................................................................................................ | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................TGTCTGACGCAGGTAGAAA......................................................................................................................................................................................................................................................... | 19 | 3 | 20 | 0.15 | 3 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .......................................CAATGTCTGACGCAGGTAGAA.......................................................................................................................................................................................................................................................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................AAGTAGTAGCAGTGCAGCAAA............................................................................................................................................................................................................................................. | 21 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................................................................CGGCACATCGGTTCGGTGG........................... | 19 | 3 | 16 | 0.06 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................AACTAAGGAAATGTCTGACT................................................................................................................................................................................................................................................................... | 20 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................................................................................CTACGAAAACCAACAAGGG..................................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................GTAAGTGGAAGAAGATCAT................................................................................................................................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...................GGGAGCGCCCTGAATTGAA................................................................................................................................................................................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................................................................................TGGCAGCGCTGTTAGCTACT.......... | 20 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
ACAGCGACACACGCACTCGCCCTCGCGGGCCTTGACTCGTTTACAGACTGCATTCATCTTCGTCAAGTAGTTTAATACCCCGTTATGAAAGGATGATGCTTTTCGTTGATCGCCTGTTTATGTGTATATGCTAGTCCAAATTGACCTTTTCGCAAAATCGATAATTCGATTACTCAGAACTTTAAAAATTCGATTTATATGCCTAAAAACATCGATTGTACGCGGGCAAAAGGTTATTCTTAAAGACTGAAGTATGTGTCCTGCTCCGGTTAGCCAAGCCACCGTCGCGACCGTCGACGAGGGTTGCATA
**************************************************....(((((((((((((........((((((((...........(((((((((((.....))....................((((......))))....((...((((((....))))))...))((((((...)))))).............)))))))))......))).)))))..........)))...))).)))).)))....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
SRR060687 9_0-2h_embryos_total |
M047 female body |
SRR060677 Argx9_ovaries_total |
SRR060666 160_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..............................................................................................................................................................................................................................................................................TAGCCAAACCACCATCGCGACCC................. | 23 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................TTCGCTTATTCAGGACTTTA.............................................................................................................................. | 20 | 3 | 7 | 0.14 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................................ACGCGGGCAACAGGTTCT......................................................................... | 18 | 2 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................CCTAATTGGCCTTATCGCA............................................................................................................................................................ | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................AAAAACATCGACTGTACGT....................................................................................... | 19 | 2 | 10 | 0.10 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................................................................................................................................TACTAAGAAATTTAAAAACTC....................................................................................................................... | 21 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..................................................................................................................................................................................................................................................................TGCTGTGCCGGTTAGCCAA................................. | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ............................................................................................................................................................................................................................CGGAGGCAAAAGGGTATTC....................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:8927588-8927897 + | dvi_19081 | TGTCGCTGTGTGCGTGAGCGGGAGCGCCCGGAACTGAGCAAATGTCTGACGTAAGTAGAAG--------------CAGTTCATCAAATTATGGGGCAATACTTTCCTACTACGAAAAGCAACTAGCGGACAAATACACATAT--ACGATCAGGTTTAACTGGAAAAGCGTTTTAGCTATTAAGCTAATGAGTCTTGAAATTT-TTAAGCTAAATATACGGATTTTTGTAGCTAACATGCGCC--------------------------------------------------------------CGTTTTCCAATAAGA------ATTTCTGACTTCATACACAGGACGAGGCCAATCGGTTCGGTGGCAGCGCT-GGCAGCTGCTCCCAACGTAT |
| droMoj3 | scaffold_6680:6417713-6418014 - | TGTTGCCGTGT----GTGCGTGAGCGTACGGAACAGAGCAAATGTCTGACGTAAGTACAAAAAAACAAACAAAAACAGTTCATCAAATTGTTAGGCGGTAATTTCCTACTA----GAGCAGCTAGTAGACAAATTT----AT--CTGATCAGGTTTAACTGGAATTGCATT---------------------------------------------------------GAAAGGTTCGCATTATATCTA----ATTTCAAACGAAAACAAAGGCAGTAATGTGAGAACATACTAAATGAATTTGAGTCTTTCAATAACATAGCGACGT------TATGCTTTCAGGACGACGCCAGTTCACTTGGAGC------TGGTT-------CTCAGCGCAT | |
| droGri2 | scaffold_15110:4690233-4690370 + | GTGAGCGT-------GATCGTGAGCGTACGGAACTGAGCAAATGTCTGACGTAAGCAGAGA--------------GAG------------CGGGGCGATGCTTTCGTATTACTCGAAACGACTTGTTGACGAATTTACATATCATTGATCAGGTCTAACTGGAATAGCGTT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004511:8899416-8899423 - | C----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCGCAT | |
| dp5 | XR_group8:4532786-4532843 + | GAT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCCATTTATAGGATGACGGCAAACAGTTTGAAGACGGCGGTGGGC-------AACTGCGCAT | |
| droPer2 | scaffold_29:768800-768857 + | GAT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCCATTTATAGGATGACGGCAAACAGTTTGAAGACGGCGGTGGGC-------AACTGCGCAT | |
| droAna3 | scaffold_13337:11653692-11653702 - | T-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGCAGCGTGT | |
| droBip1 | scf7180000396589:2319551-2319562 + | CT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCAATCGTGT | |
| droKik1 | scf7180000301816:15122-15262 + | TTTAATTTG-------AT--------------------------------------------------------------------------------------------------------------------------------------------------AAACATTC-------------AATCTGTTTTAAAATTTAGTAAGATATTAACAAAGATTTTCCTGTAAAAAATGTAATATATCTCTTCAAT-------------------------------------------------------------------------------TTCAG---AATGAAGACGACAATCAGAGCGATGGTGCTGGGGGAT-------CGGGACGGAT | |
| droFic1 | scf7180000453052:531480-531487 + | C----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCGCAT | |
| droEle1 | scf7180000491249:871876-871892 - | AG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGGAT-------CACAGCGCAT | |
| droRho1 | scf7180000780101:240093-240100 + | C----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCGCAT | |
| droBia1 | scf7180000300910:764786-764814 - | GGTGGCCTT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------C-GACAACCGCATCAAGCGCA- | |
| droTak1 | scf7180000414038:121172-121179 + | C----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCGCAT | |
| droEug1 | scf7180000409184:739189-739196 - | C----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AACGCAT | |
| dm3 | chr3L:707582-707592 - | T-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGCATCGCAT | |
| droSim2 | 3l:672278-672288 - | T-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGCATCGTAT | |
| droSec2 | scaffold_2:727505-727515 - | T-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGCATCGCAT | |
| droYak3 | 3L:699384-699394 - | T-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGCAGCGCAT | |
| droEre2 | scaffold_4784:717499-717516 - | CA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATGGAT-------CGCAGCGCAT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 08:06 PM