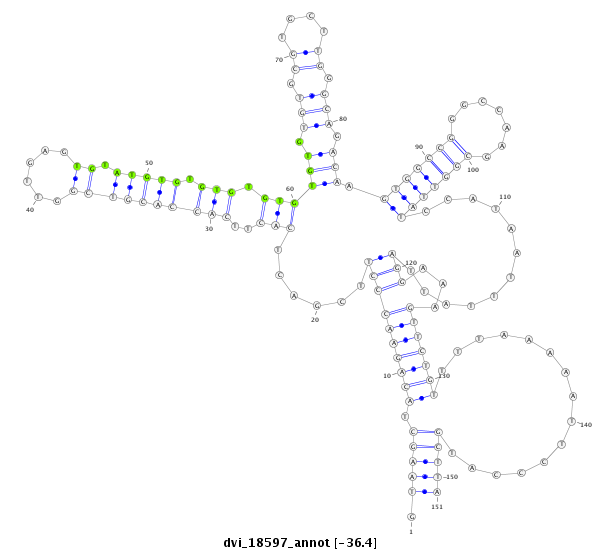
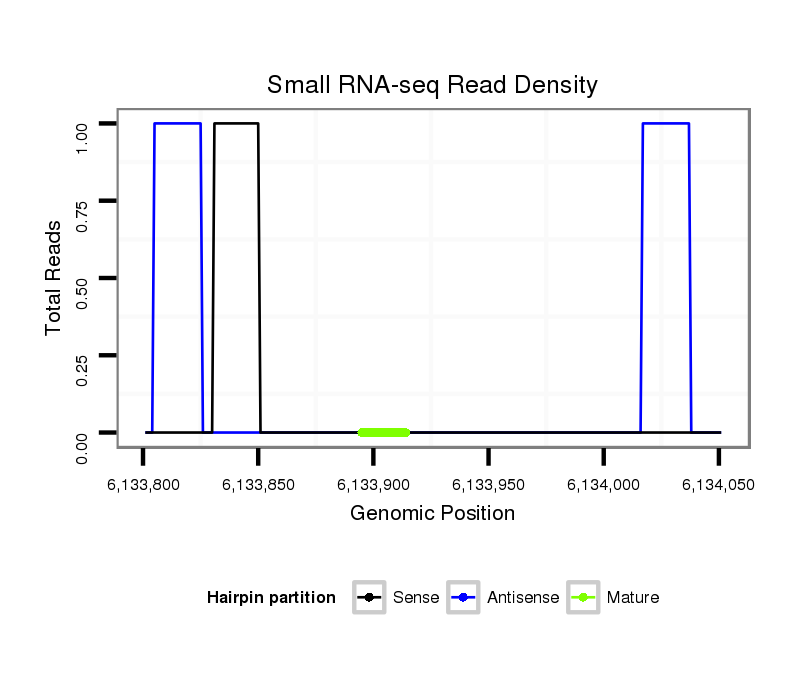
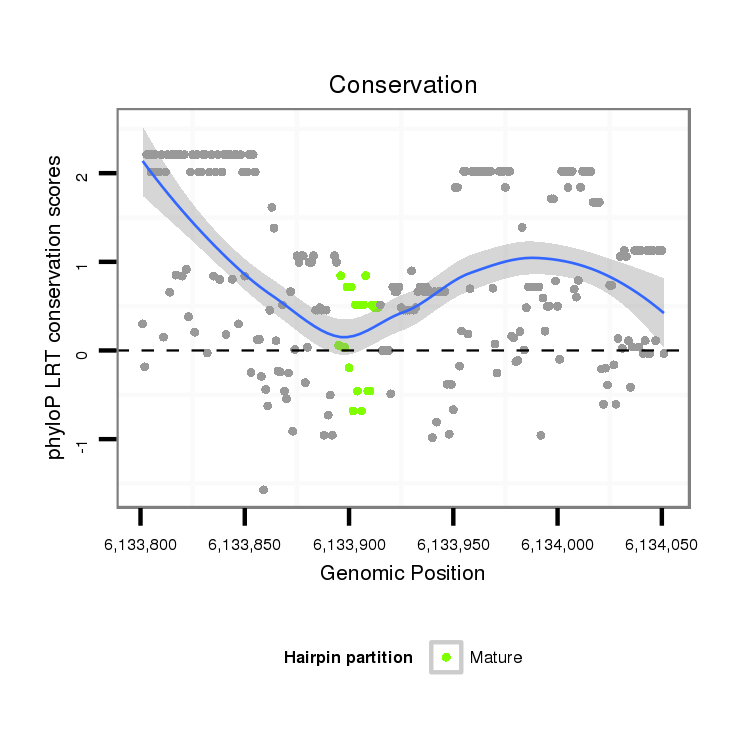
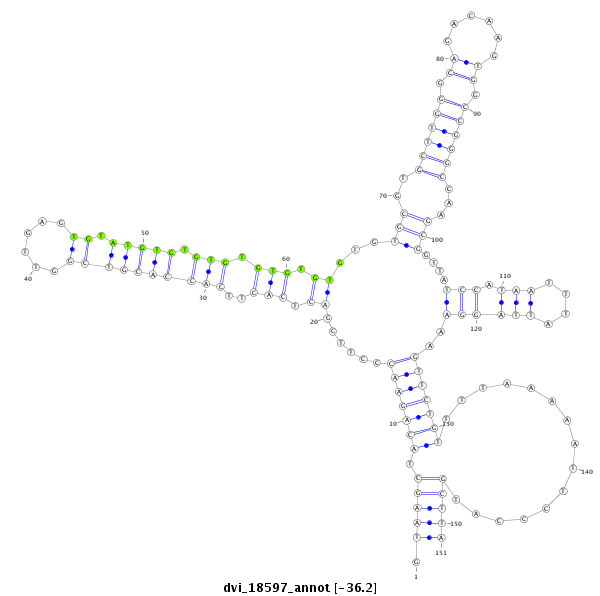
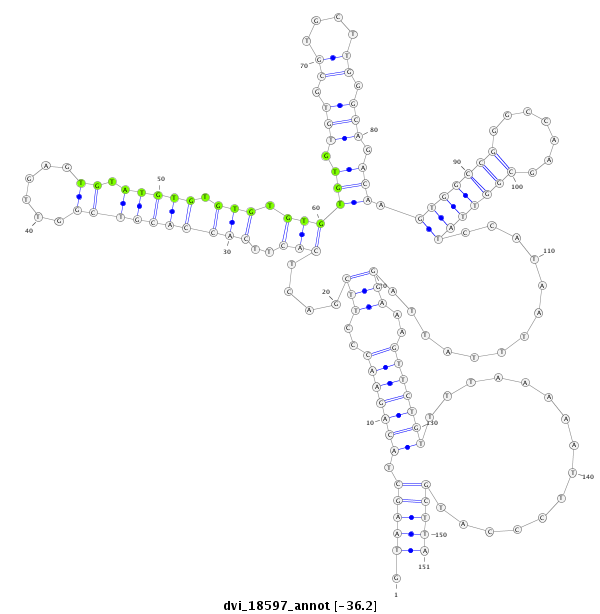
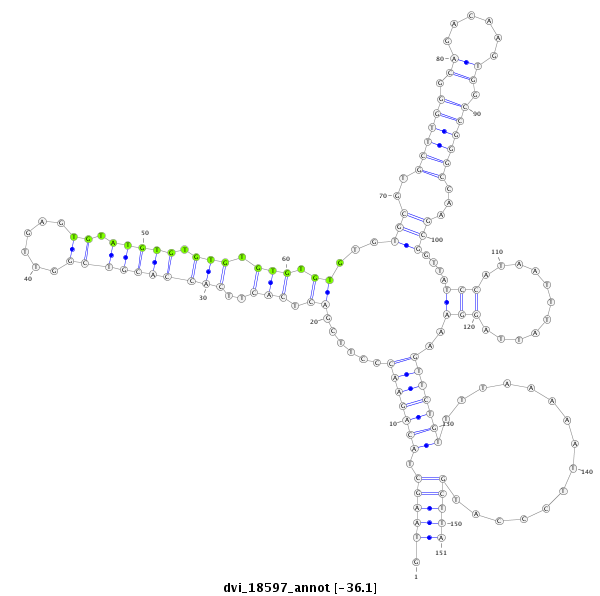
ID:dvi_18597 |
Coordinate:scaffold_13049:6133851-6134001 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -36.2 | -36.2 | -36.1 |
 |
 |
 |
exon [dvir_GLEANR_12965:1]; CDS [Dvir\GJ13042-cds]; intron [Dvir\GJ13042-in]
| Name | Class | Family | Strand |
| (TG)n | Simple_repeat | Simple_repeat | + |
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- ACTACAACGACGATTACTACAATCAATGGAATCACCAACTCTTCTTCACGGTAAGCTACAGAACCCTTCGACTCACTTCACCACGTCGGTTGAGTGTATGTGTGTGTGTGTGTGTGTGCGTGCTTGGGCAGACAAGTGGCCGGGCCAAGCGGTTATCCATAATTTATTAGGAAAGTTCTGTTTTAAAAATTCCCATGCTTAAAACTTACACAATATGCGCCGTGTTCGTGTTCGTGTTGAGCTCAACTTTC **************************************************.(((((.((((((((((......(((..((((((((((......)).))))).))).)))(((.(((.((....)).))).))).(((((((.......)))))))............)))...)))))))...............)))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060674 9x140_ovaries_total |
SRR060667 160_females_carcasses_total |
SRR060676 9xArg_ovaries_total |
M028 head |
V047 embryo |
SRR1106729 mixed whole adult body |
SRR060682 9x140_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
V116 male body |
SRR060671 9x160_males_carcasses_total |
SRR060666 160_males_carcasses_total |
SRR060679 140x9_testes_total |
SRR1106719 embryo_8-10h |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ............ATTAGTACAATCAATGGGAT........................................................................................................................................................................................................................... | 20 | 2 | 2 | 1.00 | 2 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................ATCACCAACTCTTCTTCACG......................................................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................ACAGAATATGCGCCGTGTTCG....................... | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............TTACTACAATCAATGGAATCACCAGC.................................................................................................................................................................................................................... | 26 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................TCAATGGAATCACCAGCTCTTCTTCACG......................................................................................................................................................................................................... | 28 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................GTTTCCAAAAATCCCATGCTT................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............TTAGTACAATCAATGGGAT........................................................................................................................................................................................................................... | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................TCCAATGATTAAGACTTACAC........................................ | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........AATTAGTACAATCAATGGGAT........................................................................................................................................................................................................................... | 21 | 3 | 10 | 0.20 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 |
| .......................................TATTCTTCACGGTAACGTA................................................................................................................................................................................................. | 19 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................................ATTCCACCACGTCGG.................................................................................................................................................................. | 15 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..........................................................................................................................................GACGGGCAAAGCGGTTGTC.............................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................ATTCTTCACGGTAACCTAA................................................................................................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................................................................................TGTATGTGTGTGTGTGTGTG......................................................................................................................................... | 20 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
TGATGTTGCTGCTAATGATGTTAGTTACCTTAGTGGTTGAGAAGAAGTGCCATTCGATGTCTTGGGAAGCTGAGTGAAGTGGTGCAGCCAACTCACATACACACACACACACACACACGCACGAACCCGTCTGTTCACCGGCCCGGTTCGCCAATAGGTATTAAATAATCCTTTCAAGACAAAATTTTTAAGGGTACGAATTTTGAATGTGTTATACGCGGCACAAGCACAAGCACAACTCGAGTTGAAAG
**************************************************.(((((.((((((((((......(((..((((((((((......)).))))).))).)))(((.(((.((....)).))).))).(((((((.......)))))))............)))...)))))))...............)))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060676 9xArg_ovaries_total |
SRR060678 9x140_testes_total |
SRR060670 9_testes_total |
SRR060679 140x9_testes_total |
SRR1106722 embryo_10-12h |
SRR060681 Argx9_testes_total |
SRR060664 9_males_carcasses_total |
V047 embryo |
M047 female body |
SRR1106715 embryo_4-6h |
SRR060658 140_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....GTTGCTGCTAATGATGTTAGT.................................................................................................................................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................CGCGGCACAAGCACAAGCACA.............. | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................TCCTTTCAAGACATAAGTTT............................................................... | 20 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................................ACAAGAGCAACTCGAGTAGAA.. | 21 | 3 | 11 | 0.36 | 4 | 0 | 0 | 0 | 0 | 0 | 2 | 1 | 0 | 1 | 0 | 0 |
| ..........................................................GTCTTGGGCAGCTGAGCGT.............................................................................................................................................................................. | 19 | 3 | 10 | 0.30 | 3 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................AAGTCTTGGGCAGCTGAGC................................................................................................................................................................................ | 19 | 3 | 7 | 0.29 | 2 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................................GACAAGCGCAACTCGAGTAGA... | 21 | 3 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................TCTTGGGCAGCTGAGCGT.............................................................................................................................................................................. | 18 | 3 | 20 | 0.25 | 5 | 0 | 0 | 5 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............AAAGATGCTAGTTACCT............................................................................................................................................................................................................................. | 17 | 2 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...................................................................................................................................TGCCCACCGGCCCTGTTCG..................................................................................................... | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................TCCTTAGTGGTCGAGAAAA.............................................................................................................................................................................................................. | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ........................................................AAGACTTGGGCAGCTGAG................................................................................................................................................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................TAGTGGTTTCAAGACAAAAT.................................................................. | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:6133801-6134051 + | dvi_18597 | ACTACAACGACGATTACTACAATCAATGGAATCACCAACTCTTCTTCACGGTAAGCTACAGAA-CCCTTCGA--------C----TCA---------------------------------------------CTTC--ACCAC---GTCGGTTGAGTGTATGTGTGTGTGTG-TGTGTGTGCGTGCTTGGGCA-----GACAAGTGG--CCGG-------------GCCAAGCGGTTATCCATAATTTATTAGGAAAGTTCTG------------------------TTTTAAAAATTC---CC--AT--GCTTAAAACTTACA--CAATATGCGCCG-----------TGTTCGTGTTCGTGTTGAGCTCAACTTTC |
| droMoj3 | scaffold_6680:4454984-4455242 + | ACTACAACGACGATTACTACAATCAATGGAATCACCAACTGTTCTTCACGGTAAGCCTTC----------GGCATATTAGC----GCA---------------------------------------------CTTC--ACCACACAGTCCGGCCAGC-CATGT-------------------GTGCTTGGGCAGGGCGGACAAGTGG-GTCCG-------------GCCACTGGGCTATTCATAATTTATTACCAAAGTTCAGAAACACAAAACAAAAACAAAAGCAACTGAAAAATTG---CG--AT--GCTTAAAACTTGCA--CAATATGCGCCG----------------GTGTTCGTGTTGAGCTCAACTTTC | |
| droGri2 | scaffold_7250:1908-2151 - | ATTACAACGACGACTACTACAATCAATGGAATCACCAACTGTTCTTCACGGTGAGCTCAA-AAGCCTCTTCA--------C----AAA---------------------------------------------CTTATGACCA------------AGTGCATATTTATTTGCACTGTGT----CTGCT-------------CAAGTGGTGACCGGCCACTGCTCCGTGCCCCGTGGTTATCTATAATTTATTAGTAAAGTTAAG------------------------CT-A-AAAATT---CCAATGC----GCAAAACTTGCACACAATATGCGCTC---------------TCTGCTCGCGTTGACCTCAACTTTG | |
| droWil2 | scf2_1100000004762:5150787-5150851 + | ATTACAACGATGACTACTATACTCAATGGAATCATCAGCTCTTCTTTACAGTAAGTTAAACAA-CC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | XR_group6:2484108-2484310 - | ACTACAACGACGACTACTACAACCAGTGGAACCACCAGCTCTTTTTCACGGTAAGCTCAC-AA-CTTT--C----------AAAGTCAAATATCCCGGAACATGCTGCTGTCATATGTGCCCCTAAGACTTGTCTTAGCCCCA------------AGTGCC---------G--C-------------------AGT-----------------------------------GCCGGTCATCCATAATTTATTGCCAAAGTTCGT------------------------TC-------------GATTGCGTGCTCTAAACTTGGA--CAATAT---------------------------------------------- | |
| droPer2 | scaffold_9:776353-776555 - | ACTACAACGACGACTACTACAACCAGTGGAACCACCAGCTCTTCTTCACGGTAAGCTCAC-AA-CTTT--C----------AAAGTCAAATATCCCGGAACATGCTGCTGTCATATGTGCCCCTAAGACTTGTCTTAGCCCCA------------AGTGCC---------G--C-------------------AGC-----------------------------------GCCGGTCATCCATAATTTATTGCCAAAGTTCGT------------------------TC-------------GATTGCGTGCTCTAAACTTGGA--CAATAT---------------------------------------------- | |
| droAna3 | scaffold_13337:1491310-1491376 + | C-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGCTATTCATAATTTATTGGCAAAGTTTGC------------------------CC-G--------AGCGAGTGC--GCTCCAAACTTGCT--CAATATGCGC------------------------------------------ | |
| droBip1 | scf7180000396544:596468-596534 - | C-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGCTATCCATAATTTATTGGCAAAGTTCGT------------------------CC-G--------GGCGAGTGC--GCTCCAAACTTGCA--CAATATGCGC------------------------------------------ | |
| droKik1 | scf7180000302391:297987-298041 - | ACTACAACGACGACTACTACAATCAGTGGAATCACCAGCTCTTCTTTACGGTGAG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droFic1 | scf7180000453839:922657-922766 + | C-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGTCATCCATAATTTATTGGCAAAGTTCGT------------------------CCTG--------CGCGAGTGC--GCTCCAAACTTGCA--CAATATGCGCTCCAATTCGAACACTTTCCCGTTCAATTTTAGTTGAATTTTC | |
| droEle1 | scf7180000491255:496782-496841 - | ACTACAACGACGACTACTACAATCAGTGGAACCACCAGCTCTTCTTCACGGTGAGTTCCA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000765516:32265-32326 - | ACTACAACGACGACTACTACAACCAGTGGAACCACCAGCTCTTCTTCACGGTGAGTTCTA-AA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000302428:4404298-4404365 - | C-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGCCATCCATAATTTATTGGCAAAGTTCGT------------------------CCTG--------CGCGAGTGC--GCTCCAAACTTGCA--CAATATGCGC------------------------------------------ | |
| droTak1 | scf7180000415254:448312-448366 + | ACTACAACGACGACTACTACAATCAGTGGAATCACCAGCTCTTCTTCACGGTGAG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droEug1 | scf7180000409466:4241771-4241825 - | ACTACAACGATGACTATTACAATCAATGGAACCACCAGCTCTTCTTCACGGTAAG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| dm3 | chr3L:12151865-12151935 - | AA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTGGCCATCCATAATTTATTGGGAAAGTTCGA------------------------CCTG--------CGCGAGTGC--GCTCAAAACTTGCA--CAATATGCGC------------------------------------------ | |
| droSim2 | 3l:11850391-11850461 - | AA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTGGCCATCCATAATTTATTGGGAAAGTTCGT------------------------CCTG--------CGCGAGTGC--GCTCAAAACTTGCA--CAATATGCGC------------------------------------------ | |
| droSec2 | scaffold_0:4336922-4336992 - | AA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTGGCCATCCATAATTTATTGGGAAAGTTCGT------------------------CCTG--------CGCGAGTGC--GCTCAAAACTTGCA--CAATATGCGC------------------------------------------ | |
| droYak3 | 3L:12199723-12199793 - | AA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTGGCCATCCATAATTTATTGGGAAAGTTCGT------------------------CCTG--------CGCGAGTGC--GCTCAAAACTTGCA--CAATATGCGC------------------------------------------ | |
| droEre2 | scaffold_4784:12141295-12141357 - | ACTACAACGATGACTACTACAATCAGTGGAACCACCAGCTCTTCTTCACGGTAAGTGCAG-GA-C-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 08:15 PM