
ID:dvi_18491 |
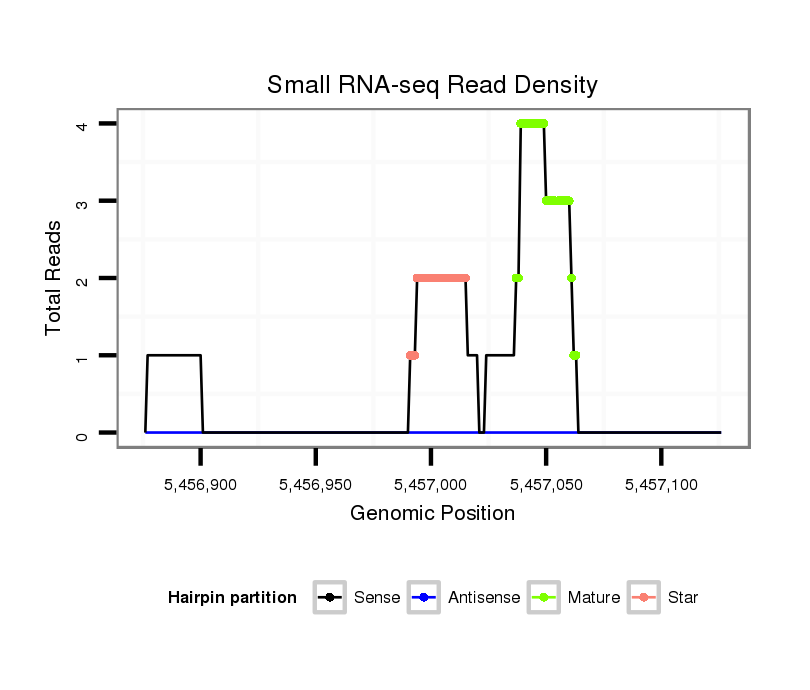
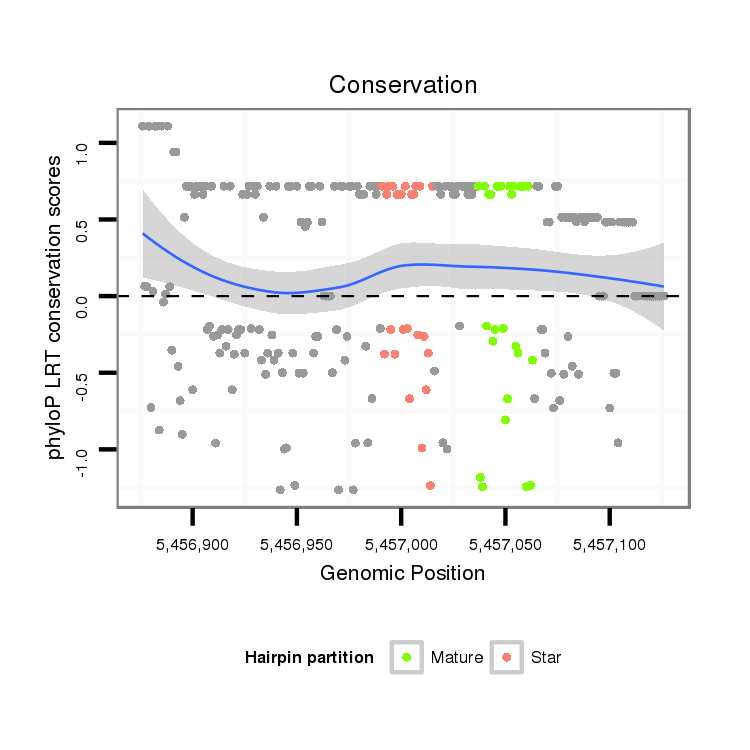
Coordinate:scaffold_13049:5456926-5457076 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -14.0 | -13.9 | -13.5 | -13.4 |
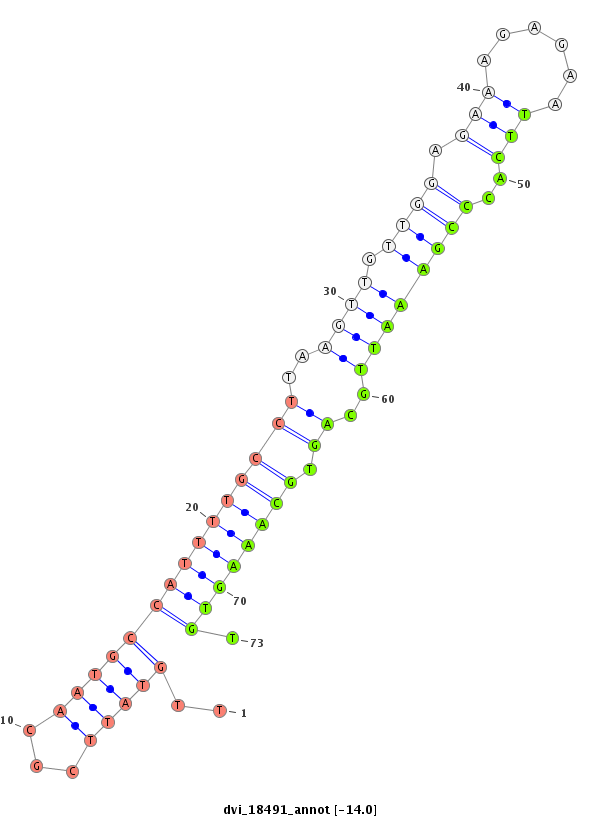 |
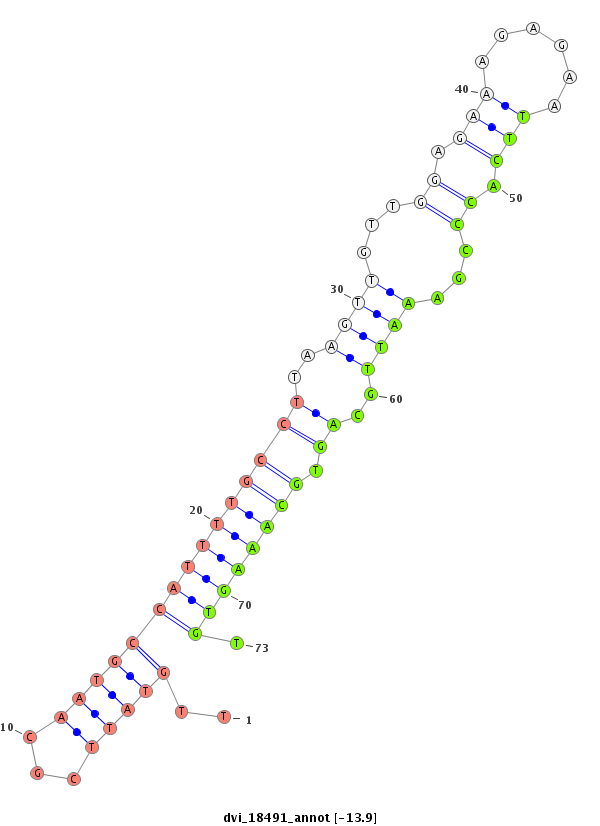 |
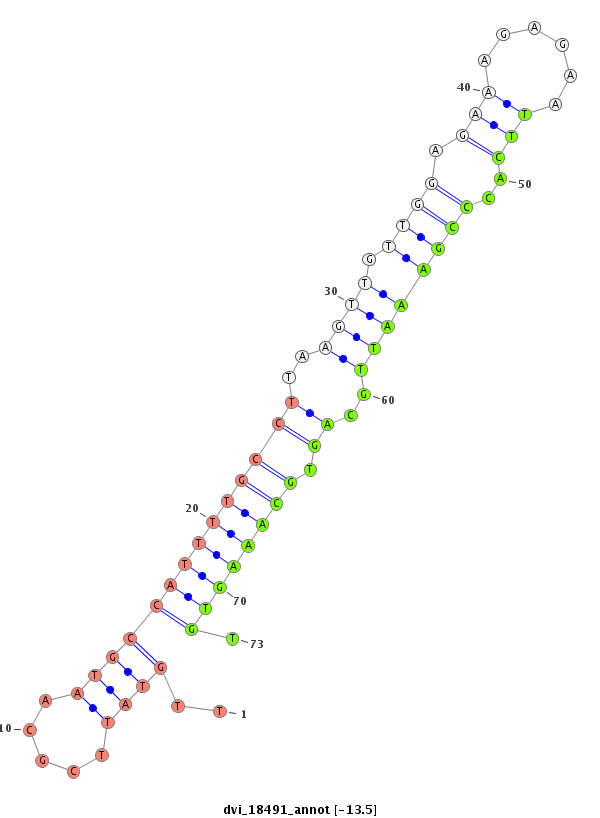 |
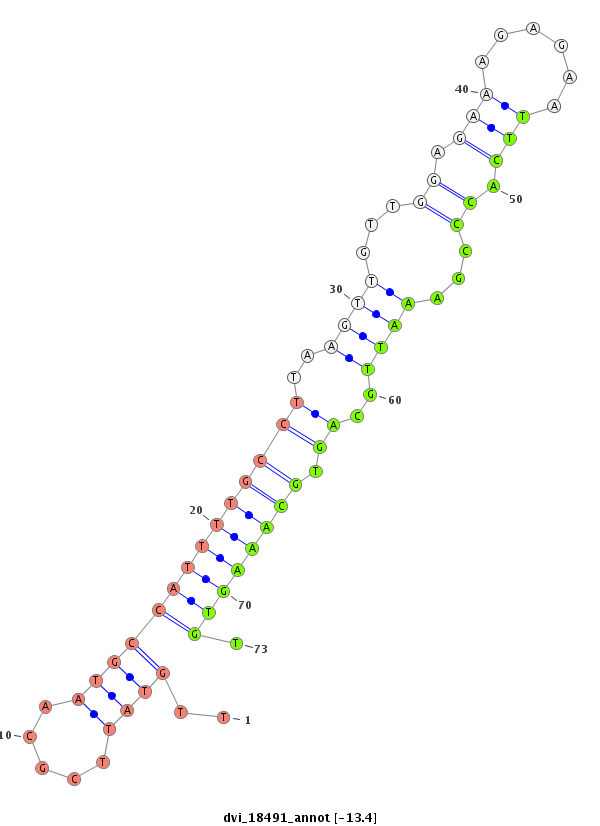 |
exon [dvir_GLEANR_12934:4]; CDS [Dvir\GJ13008-cds]; intron [Dvir\GJ13008-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- ATATCGAACTGGTTATTCAGATATGGTATCAGCTTAACGTGGTGATCTCCGTAAGTAATTGTATTCCACATACGATTTCTATCTATTGATATTCGTTAATTGTAGGGCTTGTGTTTTGTATTCGCAATGCCATTTTGCCTTAAGTTGTTGGAGAAAGAGAATTCACCCGAAATTGCAGTGCAAAGTGTGATAAGTACGTTTTAAAATTGTTTCTAAATTTAAATGTGTGTATTTTATTGTTATAAAACTTT *******************************************************************************************************************..(((((.(((.(((.((((((.....((((.((.......)).))))....)))))).))).))).)).)))*************************************************************** |
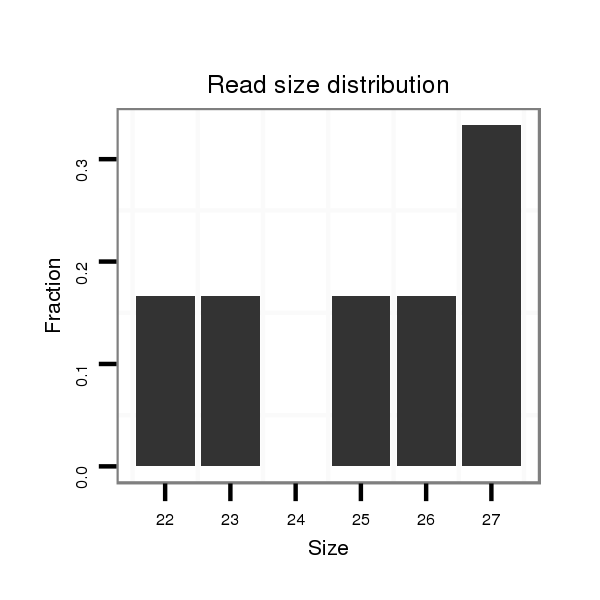
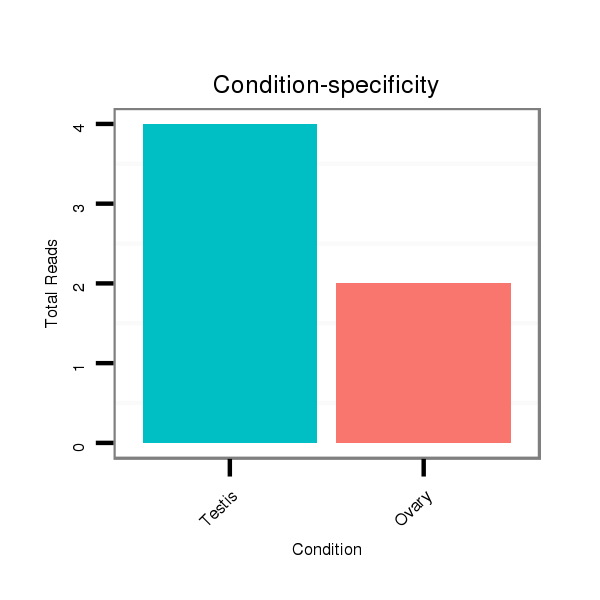
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060679 140x9_testes_total |
SRR060660 Argentina_ovaries_total |
SRR060676 9xArg_ovaries_total |
SRR060678 9x140_testes_total |
SRR060681 Argx9_testes_total |
SRR060683 160_testes_total |
SRR060666 160_males_carcasses_total |
SRR060685 9xArg_0-2h_embryos_total |
V053 head |
V116 male body |
SRR060657 140_testes_total |
SRR060677 Argx9_ovaries_total |
SRR060665 9_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................................................................................................................................TTCACCCGAAATTGCAGTGCAAAGTGT............................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................TGGAGAAAGAGAATTCACCCGAAATT............................................................................. | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................CACCCGAAATTGCAGTGCAAAG.................................................................. | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................TTGTATTCGCAATGCCATTTTGCCT............................................................................................................... | 25 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................TATTCGCAATGCCATTTTGCCTTAAGT.......................................................................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................CACCCGAAATTGCAGTGCAAAGT................................................................. | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .TATCGAACTGGTTATTCAGATATG.................................................................................................................................................................................................................................. | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................TGGTATCAGCTTACCGTGG................................................................................................................................................................................................................. | 19 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................TTTGTTGGAGAGAGAGACTTCA...................................................................................... | 22 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................................TTTCTTTTATTGTTATAAAAC... | 21 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................GTTGCAGAAAGAGAATTTAAC.................................................................................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................................................................................GTGATTTGTATTCGCCTTGCC........................................................................................................................ | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................CGTACGGTTTAAAATTGT......................................... | 18 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................................................................................................................................................AGTTGCAGAAAGAGAATT........................................................................................ | 18 | 2 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ........................................................................................................................ATCGCAATGTCATTTTGCGT............................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TATAGCTTGACCAATAAGTCTATACCATAGTCGAATTGCACCACTAGAGGCATTCATTAACATAAGGTGTATGCTAAAGATAGATAACTATAAGCAATTAACATCCCGAACACAAAACATAAGCGTTACGGTAAAACGGAATTCAACAACCTCTTTCTCTTAAGTGGGCTTTAACGTCACGTTTCACACTATTCATGCAAAATTTTAACAAAGATTTAAATTTACACACATAAAATAACAATATTTTGAAA
***************************************************************..(((((.(((.(((.((((((.....((((.((.......)).))))....)))))).))).))).)).)))******************************************************************************************************************* |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
V053 head |
SRR060667 160_females_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|
| ..............................................GAGGCATGCAGTAATATAAGGT....................................................................................................................................................................................... | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ..............................................GAGGTATTCAGTAATATAAGGT....................................................................................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 |
| ..............................................GAGGTATCCATGAACATAAGGT....................................................................................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 |
| ................................................................................................................CCAAACATAAGTGTGACGGT....................................................................................................................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 1 | 0 |
| ...........................CAGTCGACTTTCACCACTA............................................................................................................................................................................................................. | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 2 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:5456876-5457126 + | dvi_18491 | ATATCGAACTGGTTATTCAGATATGGTATCAGCTTAACGTGGTGATCTCCGTAAGTAATTGTATTCCACATACGA----------------TTTCTATCTAT-----------TGATATTCGTTAATTGTAGGGCTTGTGTTTTGTATTCGCAATGCCATTTTGCCTTAAGTTGTTGGAGAAAGAGAATTCACC------CGAAATTGCAGTGCAAAGTGTGATAAGTACGTTTTAAAATTGTTTCTAAATTTAAATGTGTGTATTTTATTGTTATAAAACTTT |
| droMoj3 | scaffold_6680:3810389-3810624 + | AAGTTGAATTGATCG------TATCGTATCAACTATGTATTATCACTTCTGTAAGTAT--ATGATTGCATTATCACCAAGGAAGCTGTTGACTCCTATTATTTTCTTTTTTTTT----GTTCTTTTTTCAAGGGACTTTGTTTTGTATTCGCAATTCCACTAACTGTGAAGCTTTTGGAGAAAGAGAATGAACCCGAACCCGAAATTCAAGTTTAAATTCATATAAATATTTT----T-----------------ATTTACCTATTTTA--------------- | |
| droGri2 | scaffold_15110:13558483-13558699 - | ATATTGAAATCATTATTTCCATATGGTATCAGTTTGATGTGGTGCTCACTGTGAGTTTTAATAATTAATCTATAACGAAAATTCGTA---------ATTTAT----------------TTCTTTTATTTCAGGGCATTTGTCTGGTTTGCGCTACTCCATTGTGTTTGAAGATATTGGAAAAAGAGAATCGATC------CTTAATCCAAGTGTAAACTAATATTTA----TTGTTAAACTGATTCTAAATT-------------------------------- | |
| droKik1 | scf7180000302344:1088784-1088800 - | ATATGTAACTGATTGTT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
Generated: 05/16/2015 at 07:46 PM