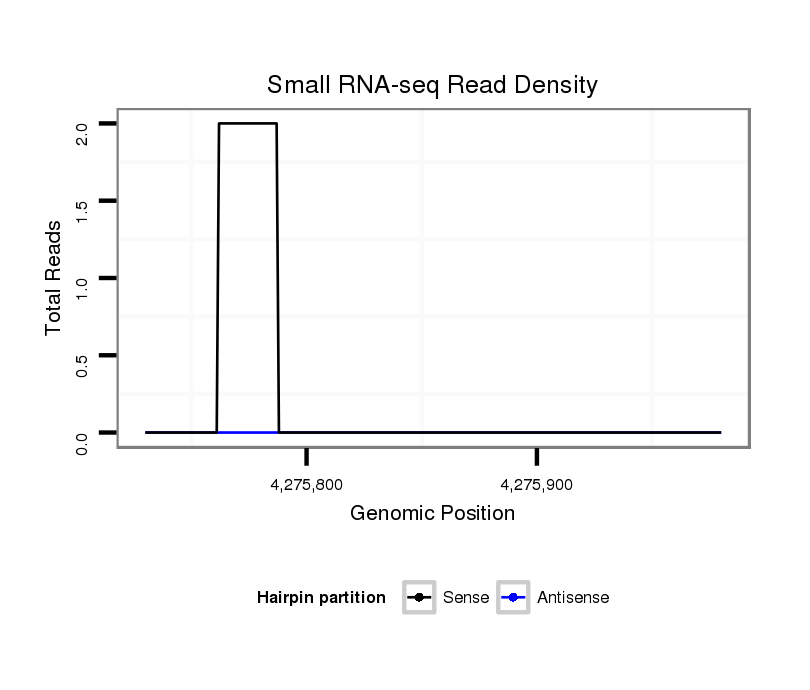
ID:dvi_18289 |
Coordinate:scaffold_13049:4275780-4275930 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ12954-cds]; exon [dvir_GLEANR_12886:5]; intron [Dvir\GJ12954-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------#####--------------------------------------------- TGGAGAATTTATTGAGAGATTTTAGAAAGGATTTCTGCAGGGATTTGCAGTAAAGATGTGAACATTAGGAAATATTATTATTACTTCAATAAGATAATCAGAGCTTTTAGAAATATCAAGAGAAGAAAAATATAATTAAATACTGATAAGATATGTACTTCTTTCTCCATATTCAATTTCATTAATTAGCCTCTCGTATAGAATAATCCCTCTCAGCAAACATTGCATACTTTAGGGTTGCGGCGAATAAC **************************************************....(((......((((((.((((....)))).))).)))......)))((((((.((((((((.....((((((((..(((((.(((........)))..)))))..))).)))))........)))))..)))..)).)))).......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060658 140_ovaries_total |
SRR1106716 embryo_4-6h |
SRR060662 9x160_0-2h_embryos_total |
SRR1106729 mixed whole adult body |
V047 embryo |
SRR060655 9x160_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ................................TTCTGCAGGGATTTGCAGTAAAGATG................................................................................................................................................................................................. | 26 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 |
| ............TGAGAGAAATTAGAAAGGATT.......................................................................................................................................................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...............................TTTCTGGAGGGATTTGCAGA........................................................................................................................................................................................................ | 20 | 2 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........ATTGAGAGAACTTCGAAAGGA............................................................................................................................................................................................................................ | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................................................................................................................AATATAATAAACTACTGAT........................................................................................................ | 19 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..................................................TTAATATGAGAACATTAGGA..................................................................................................................................................................................... | 20 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
|
ACCTCTTAAATAACTCTCTAAAATCTTTCCTAAAGACGTCCCTAAACGTCATTTCTACACTTGTAATCCTTTATAATAATAATGAAGTTATTCTATTAGTCTCGAAAATCTTTATAGTTCTCTTCTTTTTATATTAATTTATGACTATTCTATACATGAAGAAAGAGGTATAAGTTAAAGTAATTAATCGGAGAGCATATCTTATTAGGGAGAGTCGTTTGTAACGTATGAAATCCCAACGCCGCTTATTG
**************************************************....(((......((((((.((((....)))).))).)))......)))((((((.((((((((.....((((((((..(((((.(((........)))..)))))..))).)))))........)))))..)))..)).)))).......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060663 160_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
SRR060677 Argx9_ovaries_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060664 9_males_carcasses_total |
SRR060658 140_ovaries_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060673 9_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ........................................................................................................................................................TACATGGAGAAAGATGTAGAAG............................................................................. | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................AGTCTATACATGAAGAAATTG.................................................................................... | 21 | 3 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................GAAGTTATTCTATGAGACCC.................................................................................................................................................... | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................AGAGGTGTAAGTCAATGTAAT................................................................... | 21 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................AAACTAATTAATTGGGGAGCA...................................................... | 21 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................CTTTATCATAATCATGAGGT................................................................................................................................................................... | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .........................................................................................................................................................TCAGGAAGAGAGAGGTATAA.............................................................................. | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...................................................................................................................................................................AGAGGTGTAAGTTAATGCAA.................................................................... | 20 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................................................................................................................................................AGCTTAGTAGGGAGATTC................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:4275730-4275980 + | dvi_18289 | TGGA------GAATTTATTGAGAGATTTTAGAAAGG------------------------------------------ATTTCTGCAGGGATTTGCAGT--AAAG--ATGTGAAC------------ATTAGGAAAT-ATTATTATTACTTCAA-TAAGATAATCAGAGCTT-----TTAGAAATATCAAGAGAAGAAAAATATAATTAAATACTGATA----AGATATGTACTTCTTTCTCCATATTCAATTTCATT------A---------------------------ATTAGCCTCTCGTATAGAATAATCCCTCTCAGCAAACATTGCATACTTTAGGGTTGCGG-CGAATAAC |
| droMoj3 | scaffold_6680:2687950-2688025 + | CAAT------TACA---------------------------------------------------------------------------------------------------------TTC-----------------------------------------------------------------------------------------------------------------CGT-----------------------------------------------------------CTCCTCAGACAGATTTGTTGCCACCCGCAAGCATTGCATACTTTGGGGCTGCCA-CGATTAGC | |
| droGri2 | scaffold_8066:817-1106 - | CTGG------AACATTATTCCAAGACTTTGTCAGGA------------------------------------------ATTTCGATGGT--TGTAAAAT--TCTG--CTCAGAACTTTGCTCATTCCTTTAACCAAT-ATTATTTCTATTTGAT-TGATATACATAG--TTTATGCTGTCAATCAATCAACCGATTTATATTATAATTATATTTTCAATCAACTGATTTGTTTTTA--TAAAACTAATCACTTTCATTTTCGTTTTAACAAGTGTTCTATAATATCAGCATAATTTGTCA-----AACGCATT----GTGGCAGAAAGCATTGCATACTTTGGGGTTGTGC-CACAAAGC | |
| droWil2 | scf2_1100000004768:1661264-1661295 - | GCAT------------------------------------------------------------------------------------------------------------------ACTT---------------TTCTGCTGCTTTGT---T---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T-TAAATGCC | |
| dp5 | XR_group6:6822865-6822868 - | TAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------C | |
| droPer2 | scaffold_41:182490-182493 - | TAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------C | |
| droKik1 | scf7180000302577:434500-434694 + | TGCACTAGAAAAAAGAAAATTAAATATTAAAAATTATTAGAAAAATAAGAAAAAAAGGTACTATATAACTTATAGAAAAACATATTAAT--TAAAAAATAAACAACACTGAAATTTATACTC-----------------------------------------------------------------------------------------------------------------TTTTCTCTCTTAGTAATTTT--------------------------------------CCAACTCCTCCTATGAAAATATT------------AACCATTAACTTTTAAGTGACCATCATCTGGC | |
| droBia1 | scf7180000302075:1143116-1143204 + | AAAT--------------------------------------------------------------------------------------------------------------------------------------ACTGCAACTACAT---TTAAAAGCATGATTTATT-----TTATTATCATAAAGAAGATACATGTATAATTTAATTATCATA----AAATATATACT-----------------------------------------------------------------------------------------------------------------------------T | |
| droEug1 | scf7180000409466:1512613-1512626 - | TTAA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGGTCGCAG--------T | |
| dm3 | chrX:18324596-18324596 - | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | 3L:16503522-16503579 + | TGTA------GGAATGATATTAAGCGTTGAAAATGA------------------------------------------ATTTCTGTGGGATTTTACA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------G-TGGATATG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/16/2015 at 11:16 PM