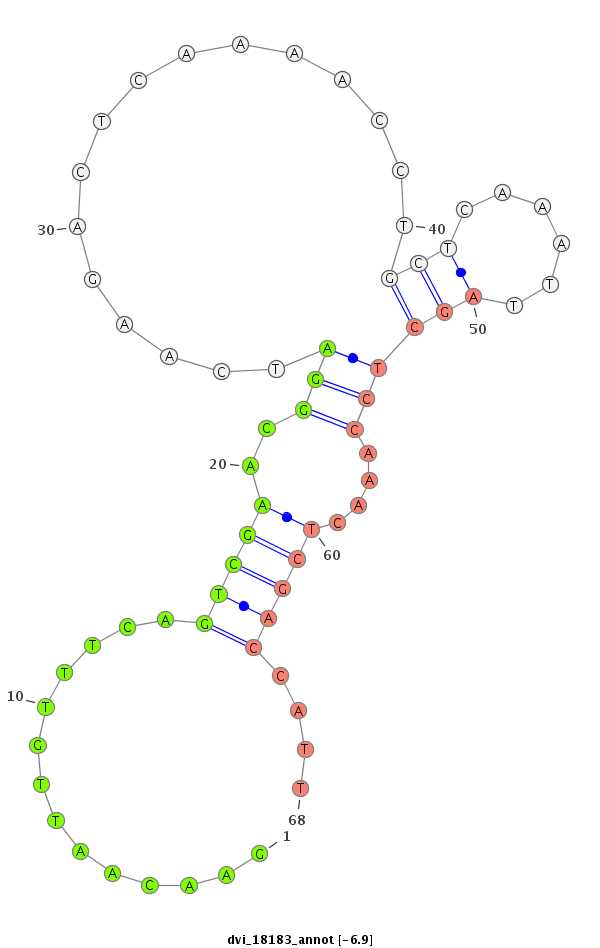
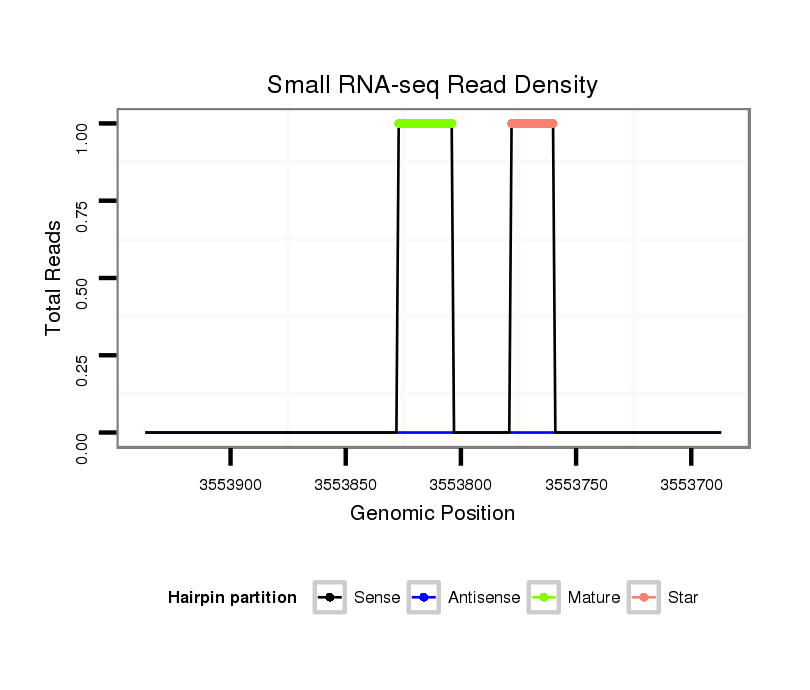
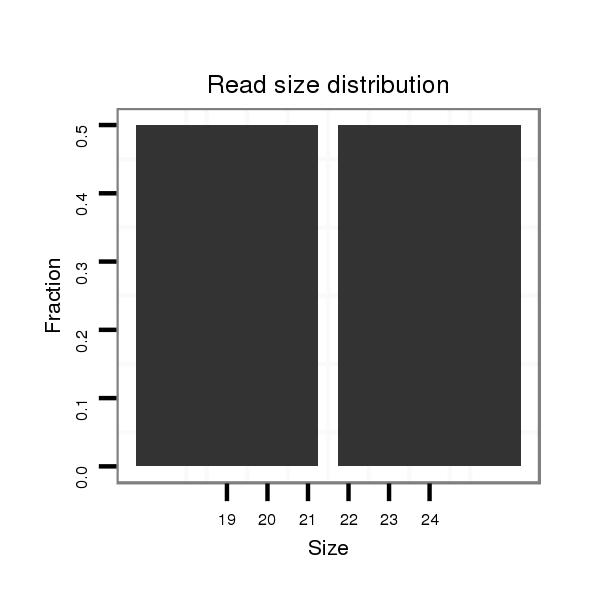
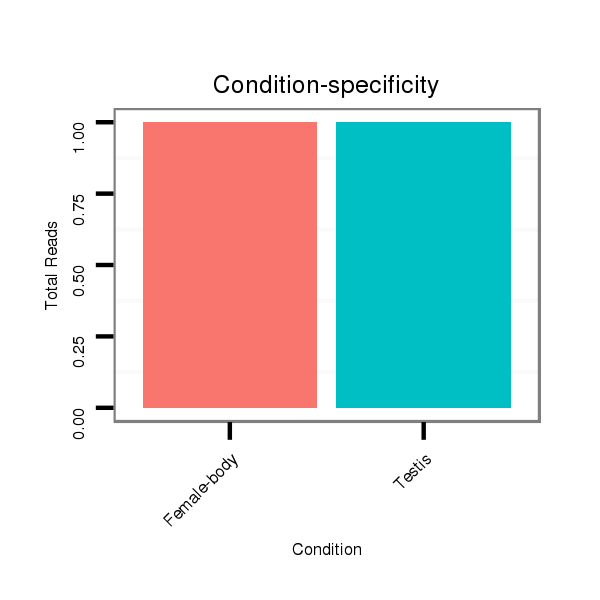
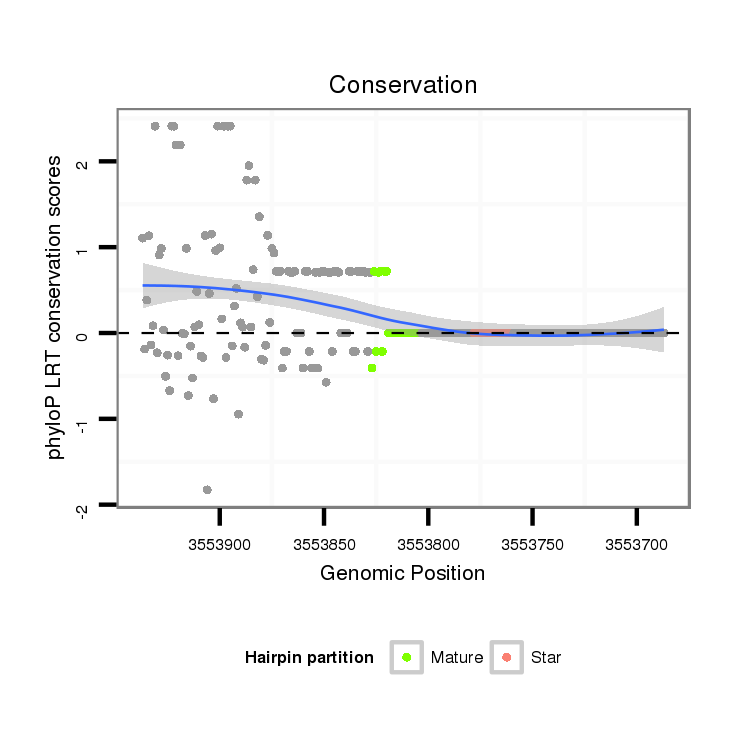
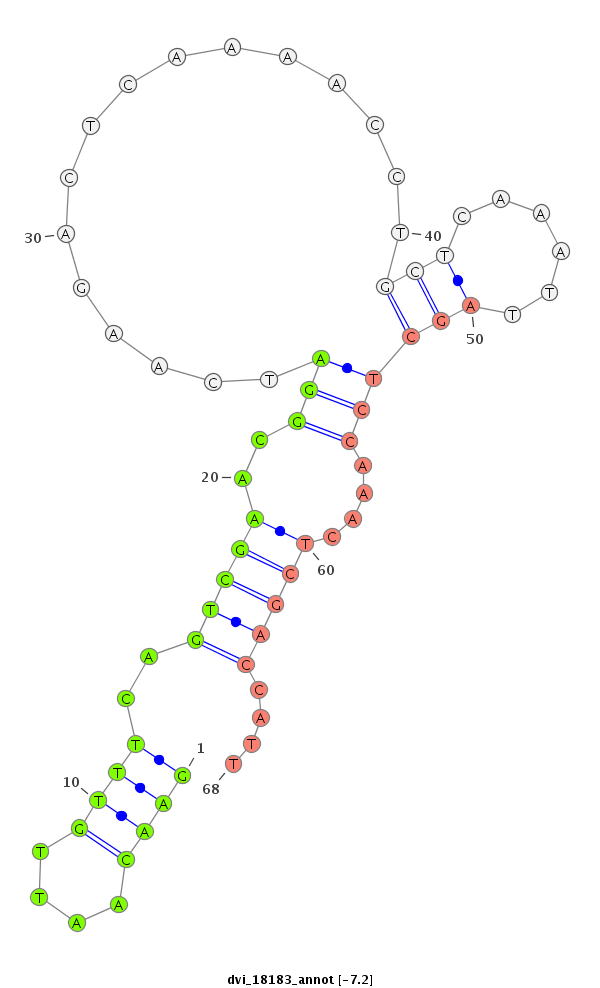
ID:dvi_18183 |
Coordinate:scaffold_13049:3553737-3553887 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -7.2 | -6.4 |
 |
 |
exon [dvir_GLEANR_12437:4]; CDS [Dvir\GJ12471-cds]; intron [Dvir\GJ12471-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- ACGTGATGCCCGTGATGCGCTCTTGCATTGTGCTGCAGTATTACGAGCGTGTAAGTGGAGCAAGAAATAGAGCTCTGCATCGAGTCAACCCAAAGGCTTTTCGAATACTCGAACAATTGTTTCAGTCGAACGGATCAAGACTCAAAACCTGCTCAAATTAGCTCCAAACTCGACCATTTCAATTGTGTTCGAGCTCGAACATGTTGCATGTTCGAGCTGATCAAACCTCGTTTGAATAAATATCTGCGGGG **************************************************************************************************************..............(((((..(((................(((......))))))....)))))....************************************************************************* |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060670 9_testes_total |
SRR060679 140x9_testes_total |
M028 head |
V053 head |
SRR060663 160_0-2h_embryos_total |
V116 male body |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..............................................................................................................GAACAATTGTTTCAGTCGAACGGA..................................................................................................................... | 24 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................AGCTCCAAACTCGACCATT......................................................................... | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................GAACGGATCAAGACTCAAAACCA..................................................................................................... | 23 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................CAGTAGAAAGGATCAAGAC.............................................................................................................. | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................CTCCAAACTCGGTCATTT........................................................................ | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................TCACAACCTGCTTAAGTTAGC......................................................................................... | 21 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................................................................................CAGTAGAAAGGATCAAGA............................................................................................................... | 18 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................................TGAAGTATTACGAACGGGT....................................................................................................................................................................................................... | 19 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .........................................................................................................................................................................................................................TGAACAAACCTCGTGTGAT............... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................GGACAAGTTTCATGTTCGA.................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................................CTGCATCGAGCGAACCCG............................................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................GTCTAAGAAGAGCAAGAAAT....................................................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TGCACTACGGGCACTACGCGAGAACGTAACACGACGTCATAATGCTCGCACATTCACCTCGTTCTTTATCTCGAGACGTAGCTCAGTTGGGTTTCCGAAAAGCTTATGAGCTTGTTAACAAAGTCAGCTTGCCTAGTTCTGAGTTTTGGACGAGTTTAATCGAGGTTTGAGCTGGTAAAGTTAACACAAGCTCGAGCTTGTACAACGTACAAGCTCGACTAGTTTGGAGCAAACTTATTTATAGACGCCCC
*************************************************************************..............(((((..(((................(((......))))))....)))))....************************************************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060689 160x9_testes_total |
V047 embryo |
SRR1106729 mixed whole adult body |
SRR060665 9_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|
| .............................................................................ATAGATCAGTTGGGTTTCAGA......................................................................................................................................................... | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................TCGAATAGTGCGGAGCAAACT................ | 21 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 |
| ..........TCACTAGGCGAGAACGTATC............................................................................................................................................................................................................................. | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 1 | 0 |
| ..........................................................................CACGTAGTTCAATTGGGTTT............................................................................................................................................................. | 20 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:3553687-3553937 - | dvi_18183 | ACGTGATGCCCGTGATGCGCTCTTGCATTGTGCTGCAGTATTACGAGCGTGTAAGT----GGA----------GCAAGAAATAGAGCTCTGCATCGAGTCAACCCAAAGGCTTTTCGAATACTCGAACAATTGTTTCAGTCGAACGGATCAAGACTCAAAACCTGCTCAAATTAGCTCCAAACTCGACCATTTCAATTGTGTTCGAGCTCGAACATGTTGCATGTTCGAGCTGATCAAACCTCGTTTGAATAAATATCTGCGGGG |
| droMoj3 | scaffold_6654:477477-477535 + | ATGTGATGCCAGTAATGCGCGCCTGCATTGTGCTGCAGTACTACGACAGCGTAAGT----GGA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_15110:7719642-7719696 - | ATGTGATGGTCATGATGCGCACTTCCATTGCCCTCCAGCATTATAGGACAGTAAG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droWil2 | scf2_1100000004521:11846129-11846183 - | ATTTAATGCCCACAATGCGGGCTAGCATAATATTTCAGCACTATCATCGGGTAAG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| dp5 | XR_group3a:667343-667388 + | ATGCAGTGCCCATCATGAGGTTCCGCTTCATTCTGCAGCACTACGA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_23:698568-698613 + | ATGCAGTGCCCATCATGAGGTTCCGCTTCATTCTGCAGCACTACGA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_13337:2014590-2014651 + | GTGTGGTGCCCAGCATGAGGGCTTCTATAATATTTCAGCACTACGACAAGGTGAGT----GGG----------GCA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000396569:2054971-2055028 + | ACGTGGTGCCCAGCATGCGGGCATCCGTAGTGTTCCAGCACTACGACAAGGTGAGT----GG----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302468:191291-191411 - | ACATAATACCAACAATGCGGGCGGCCAGTATTTTTAAATATTATGTCAAGGTGAGT----GTTTTATTTAGAAACAAGAAAGGAAGCT---AATTTCGGCAAGCCGAAG----TTTAAATACCCTAGCAGTT------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000454113:642257-642302 + | ACGTGATCCCCGCCATGAGAGCCTGCAAGGTCTTTCAGTATTACGA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491249:4169337-4169382 + | ATGTATTTCCCGTCATGAGAGCCTCTAAGGTCTTTCAGTATTACGA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000776850:524167-524212 + | ATGTGTTCCCCATCATGAGAGCCTCCAAGATCTTTCAGTATTACGA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000300910:1546735-1546802 - | ATGTGATTCCACCAATGCGCGCCGCCAGGATCTATCAGTACTATGAAAAGGTTGGCAAGTGCA----------ACGAG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415095:391341-391396 + | ATGTGATCCCAGCCATGCGAGCCTGTAAGATTTTCCAGTATTACGAAAAGGTTAGT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
Generated: 05/16/2015 at 09:09 PM