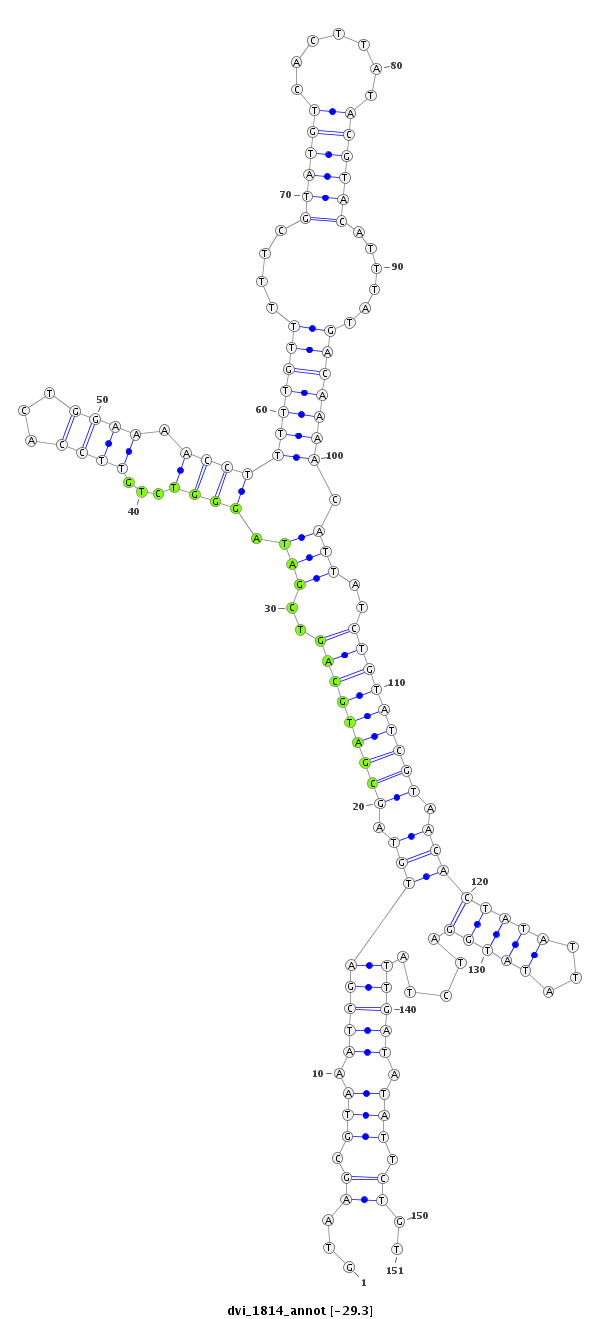
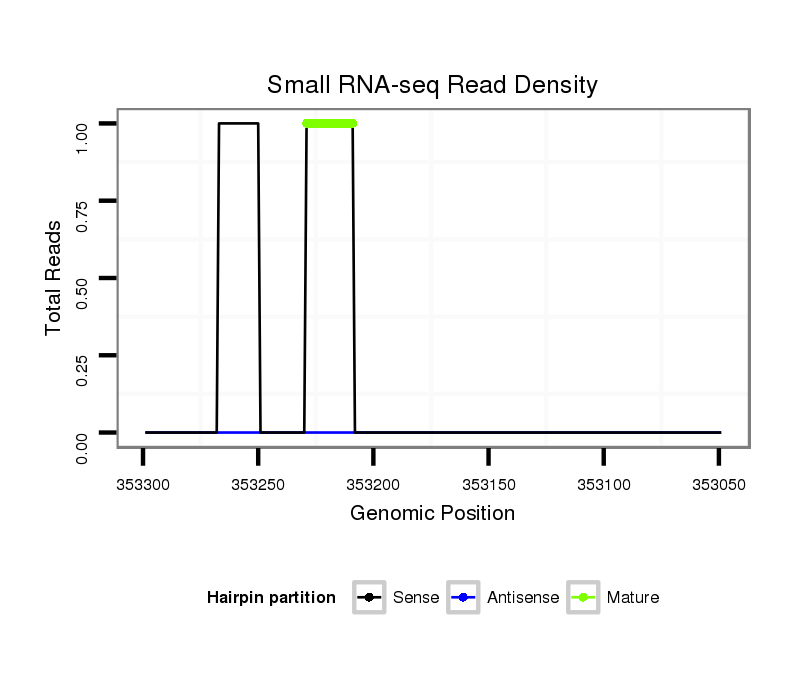
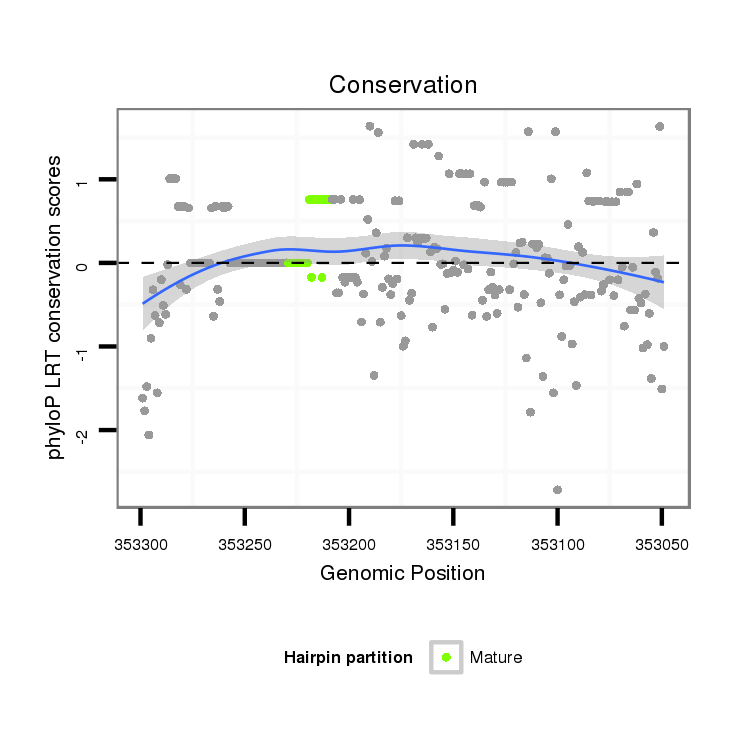
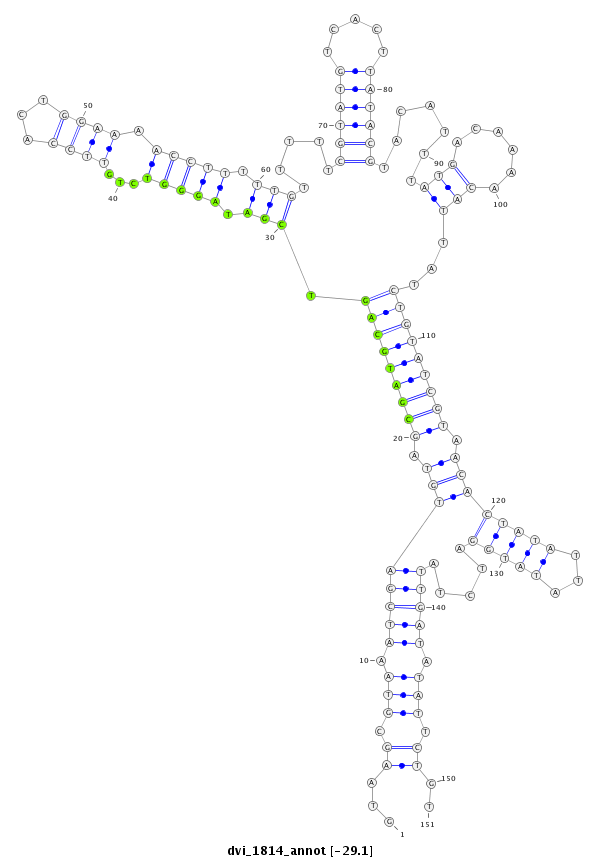
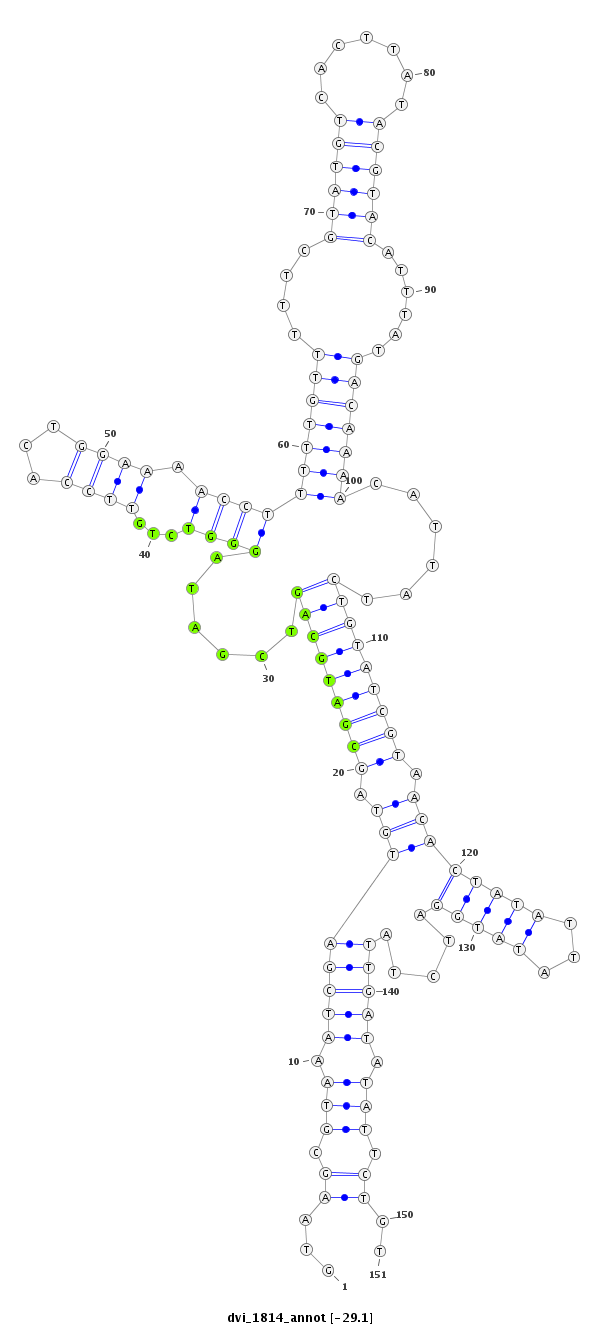
ID:dvi_1814 |
Coordinate:scaffold_12726:353099-353249 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -29.2 | -29.1 | -29.1 |
 |
 |
 |
CDS [Dvir\GJ15271-cds]; exon [dvir_GLEANR_15591:1]; intron [Dvir\GJ15271-in]
No Repeatable elements found
| ----------------------------------------##########--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- ATTTTATAAATTCATATTCATAGACTAAAGAAGCCAAGAGATGTACTCGGGTAAGCGTAAATCGATGTAGCGATGCAGTCGATAGGGTCTGTTCCACTGGAAAACCTTTTTGTTTTTCGTATGTCACTTATACGTACATTTATGACAAAACATTATCTGTATCGTAACACTATATTATATGGATCTATTGATATATTCTGTCGCAAAATGAAATTGGAAAGAAATTCTTTGTTAGCCGAACTGTCATATCA **************************************************...((.(((.((((((((.(((((((((..(((.((((...((((...)))).))))(((((((....((((((.......))))))......))))))).)))..))))))))).)))(((((...))))).....))))).))).))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
SRR060659 Argentina_testes_total |
SRR060678 9x140_testes_total |
SRR060676 9xArg_ovaries_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|
| ..................................................GTAAGCGTATAGCGAGGTAGC.................................................................................................................................................................................... | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ................................GCCAAGAGATGTACTCGG......................................................................................................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................CGATGCAGTCGATAGGGTCTG................................................................................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 |
| ...................................................................................................................................................................................TGGATCTATTGGTATGTTGTG................................................... | 21 | 3 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................GAACATTATCTGCATCGGA..................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
| .................................................................................................TGGTAAGCCTTTTTGTTTT....................................................................................................................................... | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 |
|
TAAAATATTTAAGTATAAGTATCTGATTTCTTCGGTTCTCTACATGAGCCCATTCGCATTTAGCTACATCGCTACGTCAGCTATCCCAGACAAGGTGACCTTTTGGAAAAACAAAAAGCATACAGTGAATATGCATGTAAATACTGTTTTGTAATAGACATAGCATTGTGATATAATATACCTAGATAACTATATAAGACAGCGTTTTACTTTAACCTTTCTTTAAGAAACAATCGGCTTGACAGTATAGT
**************************************************...((.(((.((((((((.(((((((((..(((.((((...((((...)))).))))(((((((....((((((.......))))))......))))))).)))..))))))))).)))(((((...))))).....))))).))).))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060678 9x140_testes_total |
SRR060679 140x9_testes_total |
V047 embryo |
M061 embryo |
SRR060672 9x160_females_carcasses_total |
SRR060658 140_ovaries_total |
GSM1528803 follicle cells |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................AGACAAGGTGACCTTTTGGTA............................................................................................................................................... | 21 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................TTCGCTACATCGCTACGTCAG........................................................................................................................................................................... | 21 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................CATAGAAGGTGACCTTTTGGAAGA............................................................................................................................................. | 24 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................................AGGTGACCTTTTGGAAGAG............................................................................................................................................ | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................TGCGTTTTACTGTAACCTTT............................... | 20 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................................................................................................................................TGTTTTGCAGTAGACATA......................................................................................... | 18 | 2 | 10 | 0.20 | 2 | 0 | 0 | 0 | 0 | 0 | 2 | 0 |
| ...........................................................................GTCAGCTATCCTAAGCAAGG............................................................................................................................................................ | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ....................................................TTTTCATTTTGCTACATCGC................................................................................................................................................................................... | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12726:353049-353299 - | dvi_1814 | ATTTTATAAATTCATATTCATAGACTAAAGAAGC---CAAGAGATGTACTCGGGTAAGCGTAAATCGATGTAGCGATGCAGTCGATAGGGTCTGTTCCACTGGAAAACCTTTTTG-TTTTTCGTATGTCACTTATACGTACATTTAT------GACAAAACATTATCTGTATCGTAACACTATATTATATGGAT-----------------------------------------------CT--ATTGAT-----ATATTCTGTCGCAAAA----------TGAAATTGGAAAGAAATTCTTTGTTAGCCGAACTGTCATATCA |
| droWil2 | scf2_1100000004762:2470826-2470948 + | GATAGC------------------------------------------------------------------------------------------------------TTTTTTG-TTTATT------TTAAAATGCATATATGTGTATTTAAAAATATGCTTTATCTCTATCT---------------TTGCCAAACTTAAGA-------------------------------------CTTT-----------------TTCTGCATTTGCCAGCAAATTGGACT------------------------TGAGGTAAAATTC | |
| dp5 | XL_group1a:3062360-3062399 + | ACGTTGTC----------------------------------------------------------------------------------------------------TCTTTTC-GTGTGT------TTCTTATACATATATCTAT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_2453:327-399 + | ATGCTATAAGGCCATATTTATTG----------CTATGATTAGAT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTAC-----------------TCGTACATTTGTATATGTTTAGAT-----------------------------TCCGATGTAG | |
| droKik1 | scf7180000302694:148697-148735 + | TATATATA----------------------------------------------------------------------------------------------------TAA---------------------------------------------------------------TTACGCGATATTATTTGGAT-----------------------------------------------CT--GTTGAT-------------------------------------------------------------------------- | |
| droEle1 | scf7180000486503:1-152 + | TTTTTAATT----------------------------------------------------------------------------------------------------TTTTTAGTT----------TCTTCGTACATATAGTAAT------GGTTAAATATTTTAT------------------------ACAACGGTTAA-ATTTCATCAATATCGGACGACTATATCATATAGCTCC----ATAGAA-----ATAATATAATATATAA----------------------ATAACTATCTAATAATTGAGCTG-CAAATCA | |
| droRho1 | scf7180000780249:40237-40327 + | ATCATAATAATATATAT-------------------------------------------------------------------------------------------------------------------------------------------------------------TCTTAATTTATTTTTATGAC-----------------------------------------------AT--TCTGACAAAAGCAA---TTAAAAATTATTAAAT-------------------------TATTAGTCGAAAAAAAATATAA | |
| droBia1 | scf7180000302250:12175-12299 - | TGGAAAGC----------------------------------------------------------------------------------------------------ACATTTT-TTCATTAGTTATGAATTTTAAATTTA--------------------------------------------------------------ATTTTAACAAAGTCGAAAGACTATATCATATAGCTAT----ATTGGT-----GTGTTGTGACATATTATAAAATAATTG----------------------------GGAATATACTTTTT | |
| droTak1 | scf7180000415388:211999-212054 - | AGCAGCATA----------------------------------------------------------------------------------------------------TTATGT---------------------------------------------------------------------------GTAT-----------------------------------------------TT--CTTTAT-----ATATTTTCACTTGAAG----------CATAATTTGAAAG------------------------------ | |
| droYak3 | v2_chrUn_280:5711-5756 + | GGCGCAGTC--------------------------------------------------------------------------GGTAGGATCTGTTAAATAAAGAGTCCTTTTAG-TT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/16/2015 at 10:37 PM