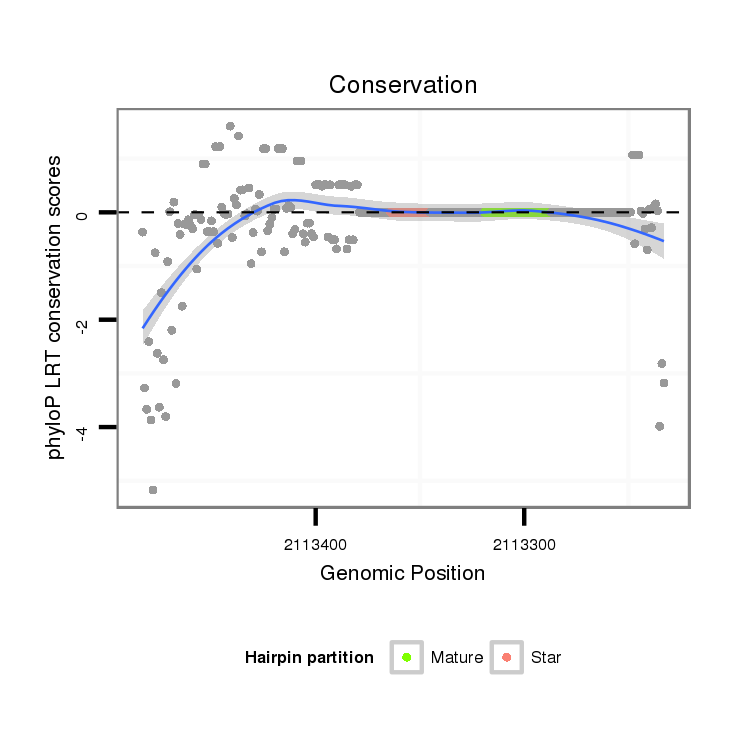
ID:dvi_17971 |
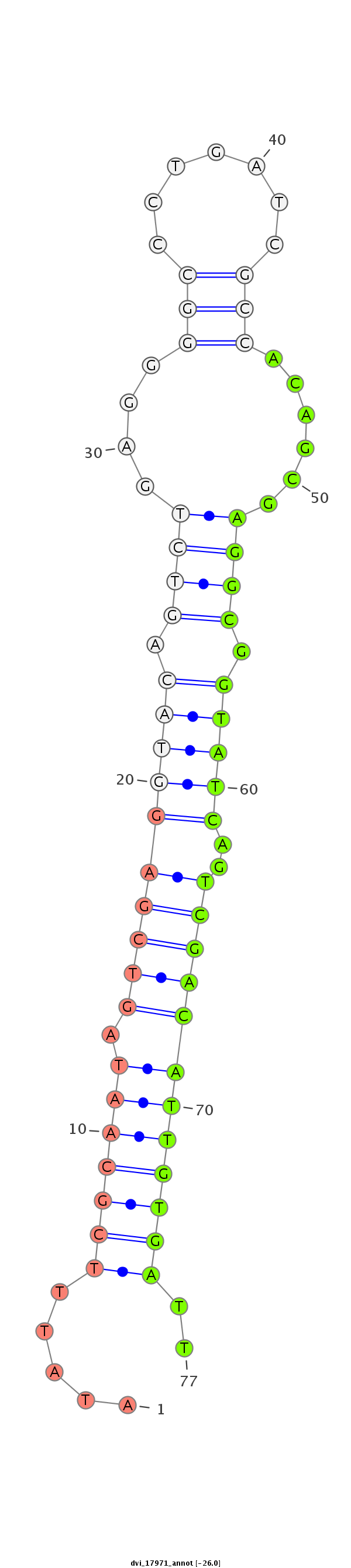
Coordinate:scaffold_13049:2113283-2113433 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
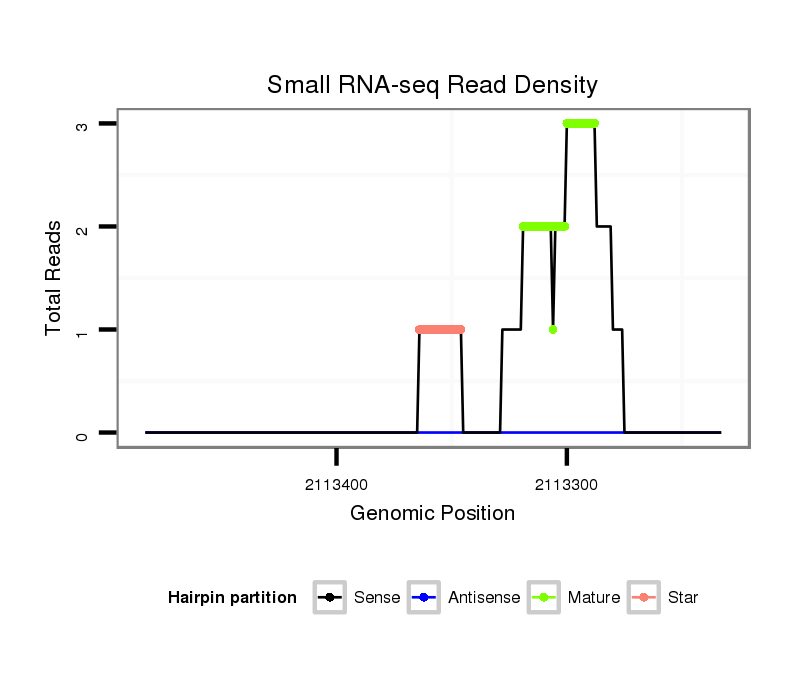
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -25.1 |
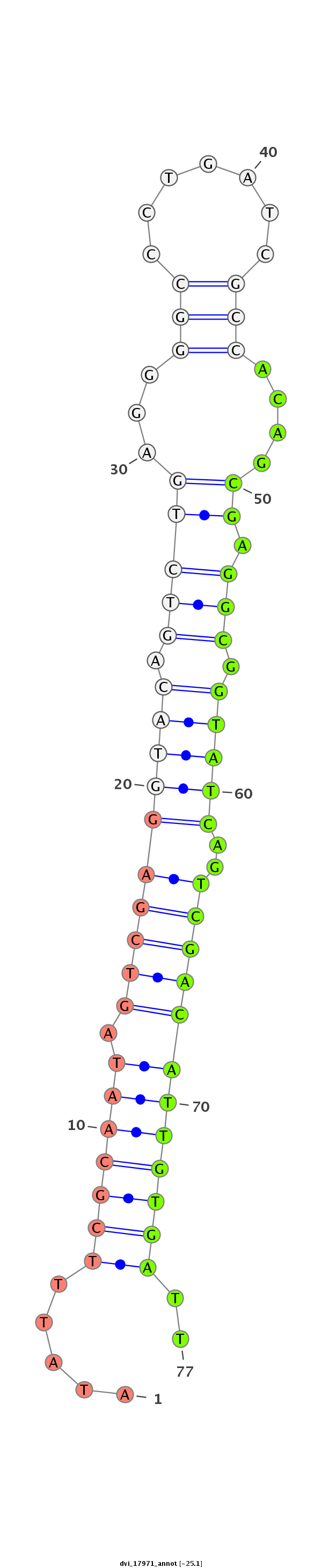 |
exon [dvir_GLEANR_12524:2]; CDS [Dvir\GJ12564-cds]; intron [Dvir\GJ12564-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## ATTAAGTAAAAGAAATATTTGATGAAGTGAATTTTAGATGCAATTCAAATATATACAAATGGCATTTATTTCTTAAATGCCTGAAAGAAAATTCTTATCAGAATTTCATATACTATGAAATATTTCGCAATAGTCGAGGTACAGTCTGAGGGGCCCTGATCGCCACAGCGAGGCGGTATCAGTCGACATTGTGATTTATAGCCTCGGCTAAAGCTGGCCCGGGACTTCGCCTGGCTGATAACTGATAAGGA ***********************************************************************************************************************.....(((((((.((((((((((.((((....(((.......)))......)))).)))))..))))))))))))..******************************************************* |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060676 9xArg_ovaries_total |
M047 female body |
V053 head |
SRR060671 9x160_males_carcasses_total |
SRR060675 140x9_ovaries_total |
SRR1106722 embryo_10-12h |
SRR1106729 mixed whole adult body |
V047 embryo |
V116 male body |
SRR060672 9x160_females_carcasses_total |
SRR060658 140_ovaries_total |
SRR060674 9x140_ovaries_total |
SRR060679 140x9_testes_total |
SRR060681 Argx9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................................................ACAGCGAGGCGGTATCAGTCGACATTGTGATT....................................................... | 32 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................TCAGTCGACATTGTGATTTATAGCC................................................ | 25 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................ATATTTCGCAATAGTCGAG................................................................................................................. | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................TTGTGACTTAGAGCCTCGGCTT......................................... | 22 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................CTGATCGCCACAGCGAGGCGGT.......................................................................... | 22 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................CGACATTGTGATTTATAGCCTCGGC........................................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................................TGGACGAGAACTGATAAGG. | 19 | 3 | 20 | 0.70 | 14 | 0 | 4 | 2 | 0 | 0 | 0 | 0 | 0 | 4 | 0 | 1 | 1 | 1 | 1 |
| ......................................................................................................................................................................................................................................CTGACTGAGAACTGATAAGG. | 20 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......TAAAAGATATATTTGATAAAGTGC............................................................................................................................................................................................................................. | 24 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................................TGACTGAGAACTGATAAGG. | 19 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................ATGACAGATAACTGATAAGGA | 21 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................................TGGACGATAACTGATAAGGG | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................TTGCCGGATAACTGATAAGG. | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................GTGACGGATAACTGATAAG.. | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................AGCGTCGACTAGAGCTGGC................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
|
TAATTCATTTTCTTTATAAACTACTTCACTTAAAATCTACGTTAAGTTTATATATGTTTACCGTAAATAAAGAATTTACGGACTTTCTTTTAAGAATAGTCTTAAAGTATATGATACTTTATAAAGCGTTATCAGCTCCATGTCAGACTCCCCGGGACTAGCGGTGTCGCTCCGCCATAGTCAGCTGTAACACTAAATATCGGAGCCGATTTCGACCGGGCCCTGAAGCGGACCGACTATTGACTATTCCT
*******************************************************.....(((((((.((((((((((.((((....(((.......)))......)))).)))))..))))))))))))..*********************************************************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V116 male body |
SRR060660 Argentina_ovaries_total |
SRR060666 160_males_carcasses_total |
SRR060685 9xArg_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|
| ..............................................................................................................CTGATACTTTATAAAGCGTTAT....................................................................................................................... | 22 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ................................................................AAAAAAAGAGTTTACGGACT....................................................................................................................................................................... | 20 | 2 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 |
| ................................................................................................................................................................................ATAGTCACCTGTAACAATCAA...................................................... | 21 | 3 | 11 | 0.09 | 1 | 0 | 0 | 1 | 0 |
| ...............................AAAATCTACGTTTAGTTTA......................................................................................................................................................................................................... | 19 | 1 | 16 | 0.06 | 1 | 0 | 0 | 0 | 1 |
| .....................................................................................................................................................................................................................................GAACCGACTATTGACGATG... | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:2113233-2113483 - | dvi_17971 | ATTAAGTAAAAGAAATA-----------------TTTGAT---GAAGTGAATTTT-----------------AGATGCAATTCAAATATATACAAATGGCATTTATT-TCTTAAATGCCTGAAAGAAAATTCTTATCAGAATTTCATATACTATGAAATATTTCGCAATAGTCGAGGTACAGTCTGAGGGGCCCTGATCGCCACAGCGAGGCGGTATCAGTCGACATTGTGATTTATAGCCTCGGCTAAAGCTGGCCCGGGACTTCGCCTGGCTGATAAC---TGATAA-------------------------------------------------------------------------------------------GGA |
| droMoj3 | scaffold_6473:5780628-5780741 - | ATTATATACAAAAAATA-----------------TTT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAAATTACATTGAGATAAAAAAAAAAAAAATGTAAATTACATTAAAATAAAAATGTTTTCAATTTTCATATATATCATTAAT------CTTTTCATA----ATA | |
| droGri2 | scaffold_15203:8981526-8981651 + | ATAGC--AAAACAAATC-----------------CAGAAT-----ACTGAAAACA-----------------AAAAGCAAAGCAAACATTCACAGAT---ATTTAAAGTTACAAAA---AAAAAGAAGAAAATTATATGTAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTCGTTGATTGACAACTATTCCAAATAGTTG | |
| droWil2 | scf2_1100000004511:346079-346138 + | ACTTACTAAAACTGAAT-----------------TTTATT---ATGAATAAAACA-----------------AAATGTGATTTAAATTTGAGCA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAT | |
| dp5 | 4_group5:1769704-1769730 + | AGAGAGAGAGAGATGTA-----------------TTCAGA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAAG | |
| droPer2 | scaffold_6:2710737-2710805 + | GTTCGTTATACCAATTG-----------------ATT------------------GGTGGTGTAGGTTTTTT----ACAA----AAAGTATATATATTTTGTTTACT-TC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTA | |
| droKik1 | scf7180000302413:70195-70268 - | ATTAAGAAAAATCATTCTTAACTTATTTTATTTTTCT------------------TTTGAA-----------ATATGTTATTTACATATATAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAATTACTTT | |
| droEle1 | scf7180000491006:1487750-1487837 + | AATCAACAAATGTACTG-----------------TGGGACATG------------GGAAACACTAGTGTTTT----ATAACTTAACTACCTGCT-ATGTCAGTTATT-ACTTAAATCTTT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAA | |
| droRho1 | scf7180000779786:19037-19067 - | GAGACCTCCCCTGA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTATAGG---TGGTGA-------------------------------------------------------------------------------------------TAA | |
| droBia1 | scf7180000302428:936249-936273 - | GGTCTCCCCCTGGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTATAGG---A------------------------------------------------------------------------------------------------GGT | |
| droSim2 | 3l:4185934-4185967 - | GAGACCTCCCCTGA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTAATG-GAGGCGGTGA------------------------------------------------------------------------------------------TAAG | |
| droYak3 | X:3423525-3423548 + | ACCACGTTTATGGA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGATG------------------------------------------------------------------------------------------AAGTG | |
| droEre2 | scaffold_4784:12896773-12896805 + | AGAATATAAAAGCAGCT-----------------GTT-----------------------------------------------------CAC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAAGCAAATT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 05:11 AM