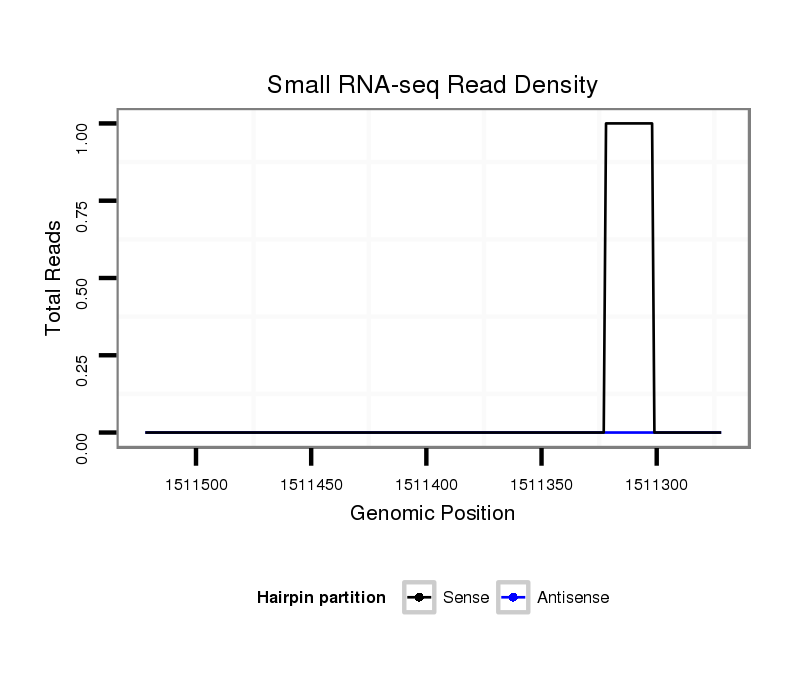
ID:dvi_17839 |
Coordinate:scaffold_13049:1511322-1511472 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ12611-cds]; exon [dvir_GLEANR_12568:6]; intron [Dvir\GJ12611-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TATATATCCACTTAAGCTAGCATGAAACAACTCGAAAATCGAATATATCTTAAACAATTTCGCGTGTAGCTCGTTTTTAGCTGCTATTTGAGTCTAAAGGAGTACCATGCAGCCGGTTGCACCCGTCCGCATCATTCATATGTGTTTTTGTATTGACTTCGATTTACCATTACCCTACGTCTGCAACTGTCCCCATTTCAGATACAAACGTGCCGCGCAGCAACTGTTCGCTGACATTGACCGACTGAATG **************************************************...........(((((..((((..((((((((........)).))))))))))..)))))...((((((((..(((.(((((.......))))).....(((.((............)).)))...)))..))))))))............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M061 embryo |
M047 female body |
SRR060666 160_males_carcasses_total |
SRR060679 140x9_testes_total |
SRR060678 9x140_testes_total |
SRR060667 160_females_carcasses_total |
GSM1528803 follicle cells |
SRR060657 140_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ........................................................................................................................................................................................................GATACAAACGTGCCGCGCAGC.............................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................TTGACTTCGATTGACCATC................................................................................ | 19 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................TGTAGCTCGTGTATTGCTG........................................................................................................................................................................ | 19 | 3 | 20 | 0.50 | 10 | 0 | 0 | 0 | 5 | 4 | 0 | 0 | 1 |
| .......................................................................................................................................................ATTGACTTCGCTGTACCACTAC.............................................................................. | 22 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................TTTACCACTACACTACGTG...................................................................... | 19 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................................................................................................................................................................................................................................ACTTTCACCGACTGAAT. | 17 | 2 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
ATATATAGGTGAATTCGATCGTACTTTGTTGAGCTTTTAGCTTATATAGAATTTGTTAAAGCGCACATCGAGCAAAAATCGACGATAAACTCAGATTTCCTCATGGTACGTCGGCCAACGTGGGCAGGCGTAGTAAGTATACACAAAAACATAACTGAAGCTAAATGGTAATGGGATGCAGACGTTGACAGGGGTAAAGTCTATGTTTGCACGGCGCGTCGTTGACAAGCGACTGTAACTGGCTGACTTAC
**************************************************...........(((((..((((..((((((((........)).))))))))))..)))))...((((((((..(((.(((((.......))))).....(((.((............)).)))...)))..))))))))............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
GSM1528803 follicle cells |
SRR060660 Argentina_ovaries_total |
SRR060665 9_females_carcasses_total |
M047 female body |
SRR060680 9xArg_testes_total |
SRR060684 140x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................................................................................................GGCGCGTAGTTGATAAGGGAC.................. | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................CGAGTCGTGGACAAGCGAC.................. | 19 | 2 | 4 | 0.50 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................GCCCATCGAGCAAAATTTGA......................................................................................................................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................................TCATTTACAAGCGACTGGAA............. | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................................................................................................................................................................................ACCTTGAAAGGGGTGAAGT................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................................................................AAAAATCGACGAGAACCT................................................................................................................................................................ | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..............................................................GCCCTTCGAGCAAAAATAG.......................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:1511272-1511522 - | dvi_17839 | TATATATCCACTTAAGCTAGCATGAAACAACTCGAAAATCGAATATATCTTAAACAATTTCGCGTGTAGCTCGTTTTTAGCTGCTATTTGAGTCTAAAGGAGTACCATGCAGCCGGTTGCACCCGTCCGCATCATTCATATGTGTTTTTGTATTGACTTCGA-TTTACCATTA-CCCTACGTC---TGCAA--CTGT--CCCCATTTCAGATACAAACGTGCCGCGCAGCAA---------CTGTTCGCTGACATTGACCGA--------CTG---------AATG |
| droMoj3 | scaffold_6654:2310670-2310773 + | TTTACATATCAA-------------------------------------------------------------------------------------------------------------------------------------------------------CTTATA--CACCTGTACCACGCCCA---CACCACGCCTATAATTCAGATACAAACGTGGCATACAGGAGCAGCATAAACGG---------ATTGACCAC--------GTG---------ATTG | |
| droGri2 | scaffold_15110:2611208-2611329 - | TGTTTTAGTATT----------------------------------------------------------------------------------------------------------------------------------------------CACTTCGAACTTACAACCGTCTCTACCCCTTCC--AACCCTGCACCCACCTTTCAGATACAAACGTGCCAC---TGAGCCGCATAAACTGATTGTTGACATTGGCCAG--------CCG---------GATG | |
| droWil2 | scf2_1100000004909:10329835-10329914 + | TGTATATACATACATAT-------AAGTAGATAGACAATCATGTATATATTAAGTG-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGTTTTGATATTTGAATTTG-CGAA--------AAT---------ACCA | |
| dp5 | XL_group1a:4444213-4444213 + | G--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_18:354907-354947 - | GCGC-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGGA-------------GCTGCTGCTGTTGCACGATGTACTGCTGG---------AGGG | |
| droKik1 | scf7180000302469:122553-122589 - | TATACATCCATTAATAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAACACTAATTAG--------CTC---------AAAA | |
| droFic1 | scf7180000453839:1912176-1912206 - | CAGC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGATTGTTGACACTGCCCCC--------GAT---------GATG | |
| droRho1 | scf7180000779409:148819-148840 + | CAAC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGATTGTTGACACTGCCC-------------------------- | |
| droEug1 | scf7180000409711:819952-819988 + | CAAC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGATTGTTGACTCTGCCCCC--------AACGAG---AATGATG | |
| dm3 | chr3L:16511599-16511629 - | CAGC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGATTGTTGACACTGCCACC--------GAT---------GATG | |
| droSim2 | 3l:16122812-16122851 - | CGGT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGATTGTTGACACTGCCCCC--------CATGATGCTGATGATG | |
| droSec2 | scaffold_0:8601990-8602026 - | CAGT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGATTGTTGACACTGCCCCC--------CATGAT---GATGATG | |
| droEre2 | scaffold_4784:17301296-17301323 + | CAGC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGATTGTTGACACTGCCCCC--------A------------ATG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 03:48 AM