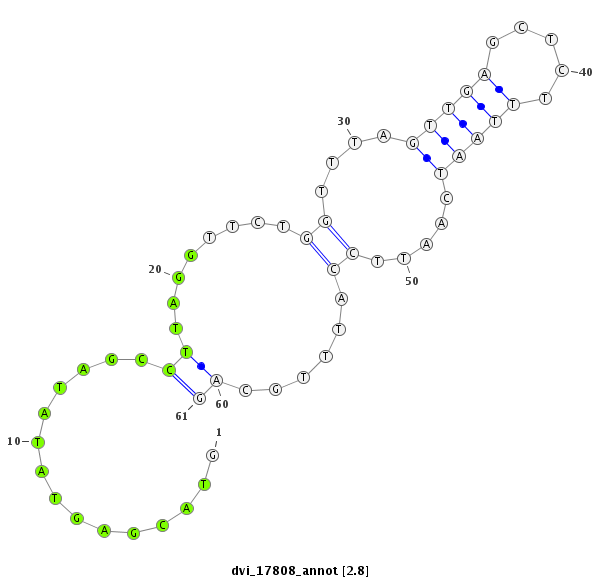
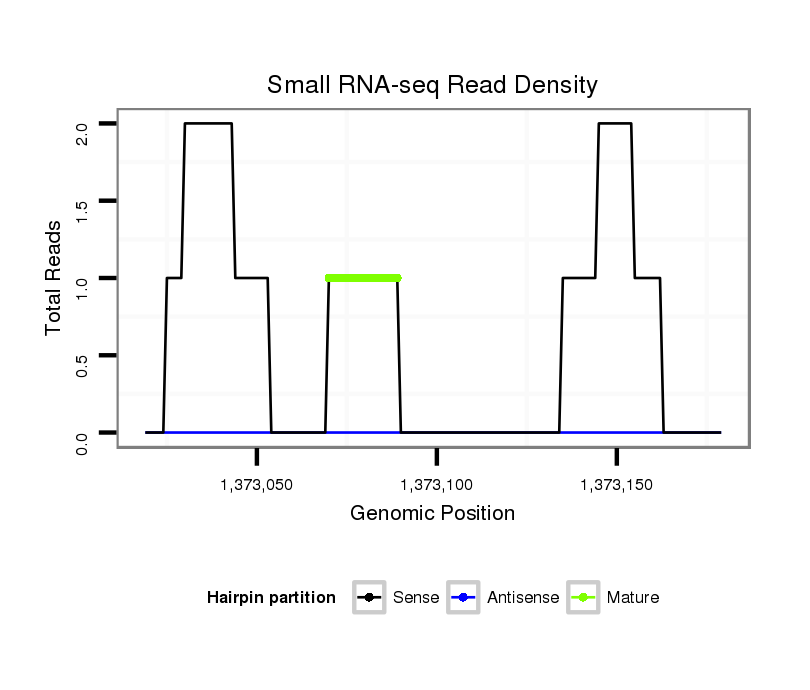
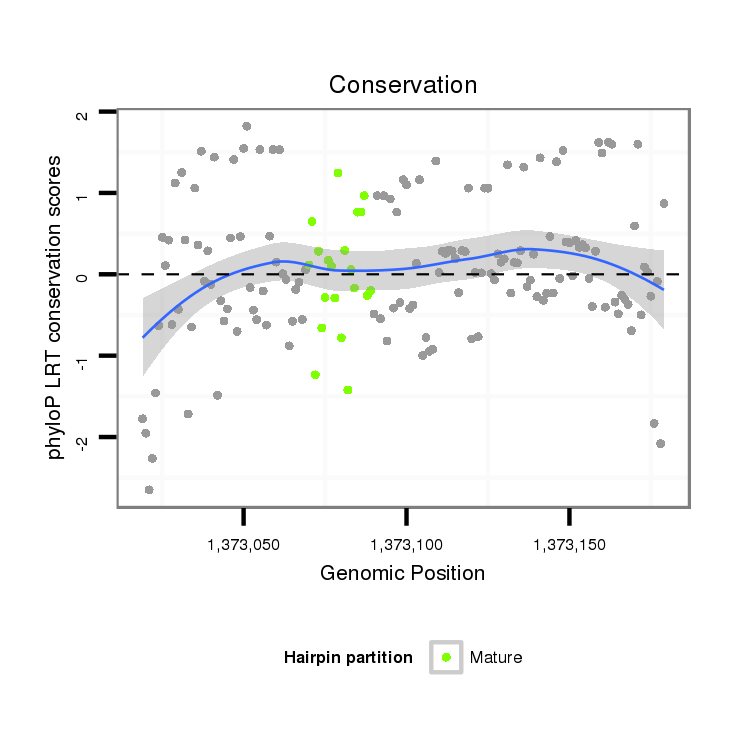
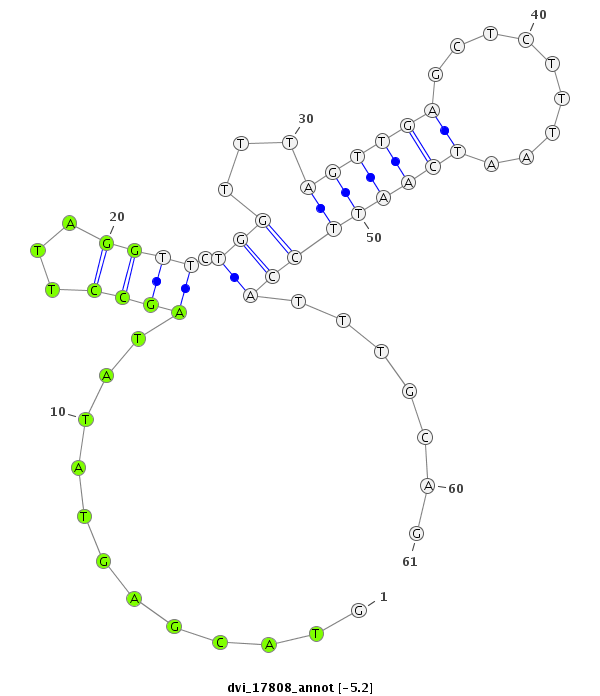
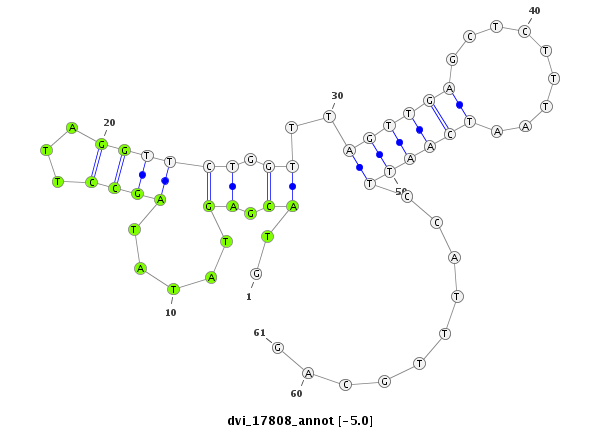
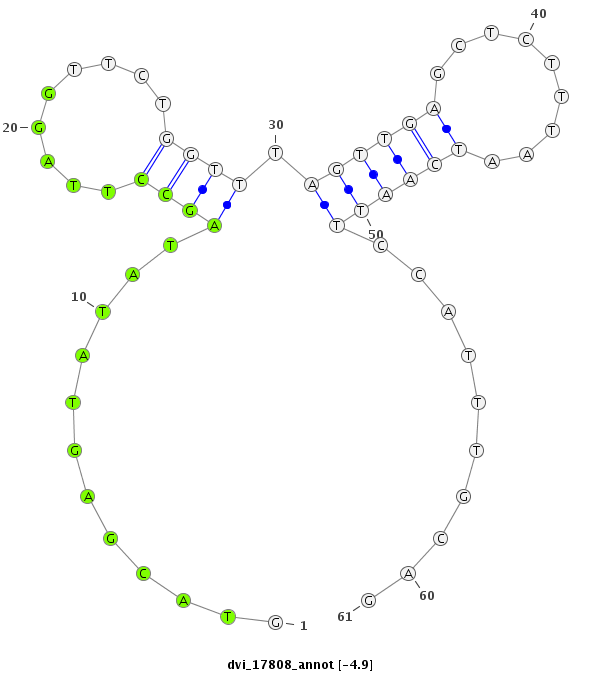
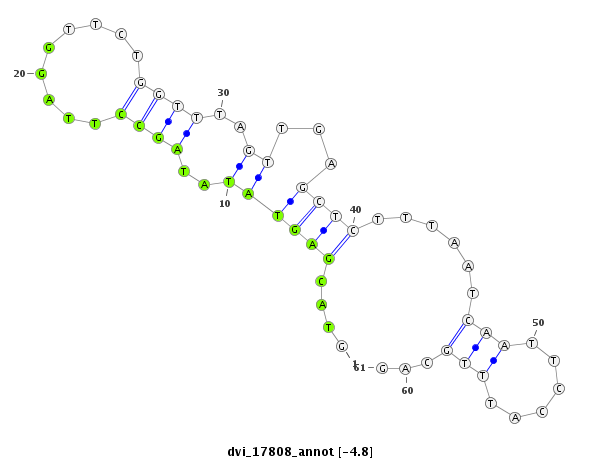
ID:dvi_17808 |
Coordinate:scaffold_13049:1373069-1373129 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -5.2 | -5.0 | -4.9 | -4.8 |
 |
 |
 |
 |
exon [dvir_GLEANR_12738:3]; CDS [Dvir\GJ12793-cds]; exon [dvir_GLEANR_12738:4]; CDS [Dvir\GJ12793-cds]; intron [Dvir\GJ12793-in]
No Repeatable elements found
| ##################################################-------------------------------------------------------------################################################## CCTGAGGAGTTGCTCGGCAAGAGTAATAAACTACAGCCGCCACAGTCACGGTACGAGTATATAGCCTTAGGTTCTGGTTTAGTTGAGCTCTTTAATCAATTCCATTTGCAGTTATCAGCAGCTGATCAATGAGTCCAAGCAGCTGACAGGCTCTACAACGG **************************************************...............((........((....(((((.....))))).....))......))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060672 9x160_females_carcasses_total |
GSM1528803 follicle cells |
SRR060662 9x160_0-2h_embryos_total |
SRR060681 Argx9_testes_total |
M061 embryo |
SRR060679 140x9_testes_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060689 160x9_testes_total |
V116 male body |
M047 female body |
SRR060665 9_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........GCTCGGCAAGAGTAATAAACTACA.............................................................................................................................. | 24 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................CCTTAGGTTGTGGTTTCGTCGA........................................................................... | 22 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................AGCAGCTGATCAATGAGTCC......................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................CAATGAGTCCAAGCAGCT................. | 18 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................ATCTATGAGTCCAAGCAGCT................. | 20 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................TACGAGTATATAGCCTTAGG.......................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................................................................................CCAGGCAGCTGACGGGCTCTA...... | 21 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......GAGTTGCTCGGCAAGAGTA........................................................................................................................................ | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................TACAGCCGCCACAGTCACGTTAT........................................................................................................... | 23 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................................................CTTAGGTTCTGGTCAAGGTGA........................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..................................................CAACGAGTATATAGCCTT............................................................................................. | 18 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ................................................................CCTTAGGTTCTGATT.................................................................................. | 15 | 1 | 9 | 0.11 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................TGCCTTAGGTTGTGGTTT................................................................................. | 18 | 2 | 12 | 0.08 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................ACGAGTCTATAGCCTTGGC.......................................................................................... | 19 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........TTGCTCGGGGATAGTAATA..................................................................................................................................... | 19 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GGACTCCTCAACGAGCCGTTCTCATTATTTGATGTCGGCGGTGTCAGTGCCATGCTCATATATCGGAATCCAAGACCAAATCAACTCGAGAAATTAGTTAAGGTAAACGTCAATAGTCGTCGACTAGTTACTCAGGTTCGTCGACTGTCCGAGATGTTGCC
**************************************************...............((........((....(((((.....))))).....))......))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060684 140x9_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
V116 male body |
SRR060682 9x140_0-2h_embryos_total |
SRR1106729 mixed whole adult body |
V047 embryo |
SRR060678 9x140_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .............................................................................................................................AGTTGCTCAGGTTCGTCGACA............... | 21 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................TGCTCATAAATCGGAACC........................................................................................... | 18 | 2 | 5 | 0.60 | 3 | 0 | 0 | 2 | 1 | 0 | 0 | 0 |
| ...................................................ATGCTCATAAATCGGAACC........................................................................................... | 19 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................TTTGCTGTCGTCGGTCTCAGT................................................................................................................. | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...................................................ATGCTCATAAATCGGAACCT.......................................................................................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...................................CGCCGGTGTCAGAGCCAT............................................................................................................ | 18 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........................TGATTTGATGTGGGCG......................................................................................................................... | 16 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:1373019-1373179 + | dvi_17808 | CCTGAGGAG-T-----T-----GCTCGG-CAAGAGTAATAAACTA-CAGCCGCCACAGTCAC-----------------------------------------GGTACGAGTATATAGCCTTAGGTT---------CTGGTTTAGTTGAGC--------------------T----CTTTAATCAATTCCATTTGCAGTTATCAGCAGCTGA---------------------------TCAATGAGT---CCAAGCAGCTGACAGGCTCTACAACGG |
| droMoj3 | scaffold_6654:2459001-2459147 - | CTTCAGGAG-C-------------TGAG-CAAGCGCAATA----AG---------------------------CCCCCT--------------------TCTCGGTA--------------------------GAGT-----CAGCAGATATTGTTGTATATGAAGAAGTTG----ATTTAGTCCATTTACTTTGCAGTTATCAGCAGCTGA---------------------------TCAATGAGT---CCAGACAGCTGACTTGTTCTACAGCCG | |
| droGri2 | scaffold_15110:2485425-2485590 + | GCTGAGGAG-TCAATGT-----ACTCGGCCAGGCGCCATAAACTA-CAGCCGCCACAGTCAC-----------------------------------------GGTTAGGATTCAGTCT---------GAATCAAAT-----CAGTTGAAA--------------------TTTTATTAACAACAACTTCCCTTGCAGTTTTCAGCAGCTAA---------------------------TCAATGAGT---CCAAGCAGCTGATAAATTCTACACCGG | |
| droWil2 | scf2_1100000004516:1770617-1770660 - | TAG--------------------------------------GCTA-TAGCTGCCACAGCTAC-----------------------------------------TGTATATGTATATAT-----------------------------------------------------------------------------------------------------------------------AC-----------------------------------ATAG | |
| droPer2 | scaffold_2:7541072-7541126 + | TTGGCCGAG-T----------------------------------------------------------------------------------------------------------------------------------------------------------------C----CTTTAATCAATTGCATTTTGAACTAGAAGCCGATGG---------------------------CGAA-------------------------------AGGG | |
| droAna3 | scaffold_13335:2203429-2203433 + | AACG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------G | |
| droFic1 | scf7180000453929:854614-854696 + | CCTCAGTTGGT-----T-----GCTCAG-CAGCAGTCGCAACT-AGTGACCACCACTGCCAC-----------------------------------------GGGAGGAACACAAATC----------------------------------------------------------------------------------------------------------------------------------------------GTGAGGACCTCGTTCAG | |
| droEle1 | scf7180000491249:7049783-7049937 - | AACGAAGATAA-----TGCTCCACCAT-CCAAGCGTCCAAAGCTAT-TCCACCCGCTAGACAGGTGGGAAGAGTTACCA--------------------ACATTTTA-----------------------------------------------------------------------------------------GACTTGCAGTTGCTCAT-TAACAAATGGCGTATATATATATTT---GTAGAT---ATCAAGAGATGGTAAATTCCACGCCCA | |
| droRho1 | scf7180000778273:20916-21059 + | TAAAG--C-----------TCCACTTG-CTAAGCGGCCAAAACTAT-TTCACTCGCAAGACAGGTGGGAAGACTAT-----------------------------------------------------------------------------GCTATAGATCCT-------------T------------------------TGGTTTTCTTCTTACGAATAT--TCAATTATTTCAAT---TAGATACCACCAAGAGATGGTCAACTCCACCCCAA | |
| droTak1 | scf7180000415857:767042-767065 + | AG--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAGATGACTAGCTCGACGCCAA | |
| droEug1 | scf7180000409711:948876-949026 - | CCACTA----------------------------------------------------GACAGGTAAGAAAACTTGCCTTCAACAATGATGGTTGCTTAACATTTTA---------AGCTTTAGCTTTGTATAGTGCTTGTTTAGAAAA-C--------------------A----TTTTAATAGGT------------------------------------T--TCTATTTATTTGTT---TAGATACCACCAAGAGCTGTCAAACTCTACGCCCA | |
| droSim2 | 2r:10780550-10780594 + | CGGCTGGA--------------------------GTGAGGAGCTA-CAGCAGCCGCAGCCAG-----------------------------------------AGCAAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCA | |
| droEre2 | scaffold_4784:18628688-18628823 + | CGTACAGAG-C--------TCCGCTAG-CCAAGCGACCAAAGCTAA-TGCACCCGCAAGACAGGTGAGAAAATAAAAATAC-----------------------------------------AACAT---------TTCGC-------------------------------------A---------------------------------TAATTTGAATGTCATATA-----A---TTATTAGGA---ACCTGGAGGTGTCAAGCTCCTTGGCAA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 11:32 PM